Volume 11, No.5 Pages 319 - 328
2. 最近の研究から/FROM LATEST RESEARCH
関節リウマチ原因タンパク質PAD4の構造生物学
Structural Biology of Peptidylarginine Deiminase 4 (PAD4) Associated with Rheumatoid Arthritis
横浜市立大学大学院 国際総合科学研究科 International Graduate School of Arts and Sciences,Yokohama City University
Download PDF (409.78 KB)
1.はじめに
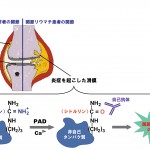
関節リウマチは、様々な遺伝的・環境的な要因によって骨と骨をつなぐ関節にある滑膜で炎症が生じ、寛解・増悪を繰り返しながら骨や軟骨の組織を破壊する難治性の自己免疫疾患で(図1)、罹患率は世界人口の約1%ときわめて高い。自己免疫疾患とは、さまざまな細菌やウイルスなどの異物(非自己=抗原)が体内に侵入したときに「抗体」という武器を作って異物を攻撃する仕組み(免疫)に狂いが生じ、自己の細胞を非自己とみなして抗体(自己抗体)を作って攻撃してしまう免疫異常のことである。関節リウマチにおいては、1998年にオランダの研究グループによって多くの関節リウマチ患者の血清中に存在するケラチンやフィラグリンに対する自己抗体がシトルリン化されたペプチドと反応することが明らかにされ[1]、関節リウマチとタンパク質をシトルリン化する酵素Peptidylarginine deiminase(PAD)の関連が注目されるようになった。さらに、2003年には理化学研究所の遺伝子多型研究センターのグループが大規模ケースコントロール関連解析を行い、関節リウマチの発症とPAD4遺伝子の一塩基多型の間に強い相関があることが示され[2]、PAD4が関節リウマチの発症に深く関わることが明らかになった。また、関節リウマチの患者は関節リウマチに特異的な自己抗体を産生しているが、いずれの自己抗体もPADによってシトルリン化されたタンパク質を非自己(自己抗原)として認識して産生されている[3]ことや、関節リウマチの発症に関与する主要組織適合系複合体[MHC II (HLA-DRB1*0401)]がシトルリン化されていないペプチドよりもシトルリン化されたペプチドと高い親和性を示すこと[4]など、いずれも骨と骨をつなぐ関節にある滑膜中のタンパク質がPADによってシトルリン化されて免疫異常を引き起こすものと考えられている(図1)。したがって、タンパク質のシトルリン化が抑制できればこのような免疫異常を防ぐことができるので、PAD4の活性阻害剤は関節リウマチの根本的な治療薬になるものと期待される。
関節リウマチは、様々な遺伝的・環境的な要因によって骨と骨をつなぐ関節にある滑膜で炎症が生じ、寛解・増悪を繰り返しながら骨や軟骨の組織を破壊する難治性の自己免疫疾患で(図1)、罹患率は世界人口の約1%ときわめて高い。自己免疫疾患とは、さまざまな細菌やウイルスなどの異物(非自己=抗原)が体内に侵入したときに「抗体」という武器を作って異物を攻撃する仕組み(免疫)に狂いが生じ、自己の細胞を非自己とみなして抗体(自己抗体)を作って攻撃してしまう免疫異常のことである。関節リウマチにおいては、1998年にオランダの研究グループによって多くの関節リウマチ患者の血清中に存在するケラチンやフィラグリンに対する自己抗体がシトルリン化されたペプチドと反応することが明らかにされ[1]、関節リウマチとタンパク質をシトルリン化する酵素Peptidylarginine deiminase(PAD)の関連が注目されるようになった。さらに、2003年には理化学研究所の遺伝子多型研究センターのグループが大規模ケースコントロール関連解析を行い、関節リウマチの発症とPAD4遺伝子の一塩基多型の間に強い相関があることが示され[2]、PAD4が関節リウマチの発症に深く関わることが明らかになった。また、関節リウマチの患者は関節リウマチに特異的な自己抗体を産生しているが、いずれの自己抗体もPADによってシトルリン化されたタンパク質を非自己(自己抗原)として認識して産生されている[3]ことや、関節リウマチの発症に関与する主要組織適合系複合体[MHC II (HLA-DRB1*0401)]がシトルリン化されていないペプチドよりもシトルリン化されたペプチドと高い親和性を示すこと[4]など、いずれも骨と骨をつなぐ関節にある滑膜中のタンパク質がPADによってシトルリン化されて免疫異常を引き起こすものと考えられている(図1)。したがって、タンパク質のシトルリン化が抑制できればこのような免疫異常を防ぐことができるので、PAD4の活性阻害剤は関節リウマチの根本的な治療薬になるものと期待される。
図1 タンパク質のシトルリン化と関節リウマチの発症
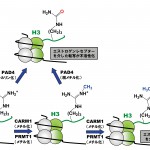
PADはCa2+イオン存在下でタンパク質中のアルギニン残基をシトルリン残基に変換する酵素で(図1)、5つのタイプ(PAD1−PAD4とPAD6)の酵素が広く体内の組織中に分布している。この5つのタイプPADs中でPAD4だけが核移行シグナル(NLS:nuclear localization signal)をもち、細胞の核内においてヒストンのシトルリン化に関与している。ヒストンのシトルリン化はメチル化と拮抗して真核生物の遺伝子の発現を調節している。事実、ヒストンH3のArg17とH4のArg3はそれぞれCARM1(cofactor associated arginine methyltransferase 1)とPRMT1(protein arginine methyltransferase 1)によってジメチル化されるが、これらの部位がメチル化されるとエストロゲンレセプターを介した転写が活性化される(図2)[5,6]。一方で、PAD4はヒストンH3のArg2、Arg8、Arg17、Arg26およびH4のArg3をシトルリン化するが、ヒストンH3のArg17とヒストンH4のArg3がシトルリン化されるとCARM1とPRMT1によるメチル化が阻害され、エストロゲンレセプターを介した転写が不活性化される(図2)[5,6]。また、興味あることに、CARM1とPRMT1によるアルギニン残基のジメチル化はモノメチル化を経て起こるが、PAD4はこのモノメチル化されたアルギニン残基もシトルリン化し、結果的にモノメチル化されたアルギニン残基を脱メチル化する(図2)[7,8]。ヒストンのメチル化は他の翻訳後修飾(アセチル化やリン酸化など)に比べて安定で、これまでヒストンの脱メチル化反応を触媒する酵素は見つかっていなかったが、モノメチル化されたアルギニン残基をシトルリン化することが示されたことによって、PAD4はヒストンの脱メチル化に関与する初めての酵素としても注目されるようになった。
図2 PAD4によるヒストンアルギニンのシトルリン化と転写調節
われわれは、これまでにSPring-8及びPFの強力な放射光X線を用いてヒトPAD4のX線結晶構造解析を行い、Ca2+イオンの結合部位やCa2+イオンによる新規の酵素の活性化機構や基質結合部位を含む酵素の全容を明らかにしてきたが、今回新たに、ヒストン(H3とH4)N末端ペプチドとPAD4との複合体のX線結晶構造解析にも成功した。その結果、PAD4はヒストンN末端ペプチドと相互作用することによって柔軟性の高いヒストンN末端ペプチドにβターン様の構造が誘導されることがわかり、これまでにないまったく新しいヒストン認識の仕組みを明らかにすることができた。さらに、PAD4によるヒストン認識がヒストンペプチドの主鎖部分を介して行われていることが示され、なぜPAD4がヒストンのN末端尾部の複数のアルギニン残基をシトルリン化できるかなども構造科学的に明らかにすることができた。そこで、本稿ではPAD4のX線結晶構造解析から明らかにされた新しい酵素の活性化機構[9]およびヒストン認識について紹介する[10]。
2.結晶化とX線結晶構造解析
ヒトPAD4は大腸菌の発現系で大量に発現され、アフィニティーカラムおよびイオン交換カラムで高度に精製された。Ca2+非結合型PAD4の結晶化はハンギングドロップ蒸気拡散法で行い、0.1M Imidazole pH 8.0、0.2M Li2SO4、8% PEGMME2000の条件で高分解能のX線結晶構造解析に適した結晶が得られた[11]。構造解析はEMTS(Ethy1 mercurithiosalicylate)、TMLA(Trimethy1 lead acetate)、KAu(CN)2、K2PtCl4、LuCl3で調製された重原子誘導体結晶を用いたMIRAS法(異常分散を考慮した多重同型置換法)で行った。また、Ca2+結合型PAD4とCa2+結合型PAD4−基質(benzoy1 L-arginine amide: BA)複合体の結晶は、PAD4のC645A不活性変異体の結晶をそれぞれCa2+およびCa2+と基質BAを含む溶液に浸漬して調製した。なお、PAD4のC645A不活性変異体[Ca2+非結合型PAD4(C645A)]の精製および結晶化はCa2+非結合型PAD4と同じ条件で行った。
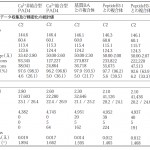
一方、PAD4−ヒストンペプチド複合体の結晶は、Ca2+非結合型PAD4(C645A)の結晶をCa2+存在下それぞれヒストンH3のArg8を含むペプチド、ヒストンH3のArg17を含むペプチド及びヒストンH4のArg3を含むペプチドを含む溶液に浸漬して調製した。表1に結晶学的データおよび回折強度データと精密化の統計値を示す。
表1 結晶学的データ、データ収集及び精密化の統計値
3.全体構造
表1 結晶学的データ、データ収集及び精密化の統計値

3.全体構造
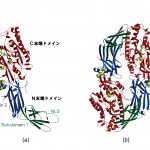
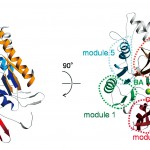
図3(a)にPAD4の全体構造を示す。PAD4は2つのドメイン[N末端ドメイン(N-terminal domain)とC末端ドメイン(C-terminal domain)]から構成され、その形は細長いブーツ状である。Ca2+はCa2+結合型PAD4およびCa2+結合型PAD4−基質BA複合体の異常分散差フーリエ図から5つ確認され、そのうち3つはN末端ドメインに、残り2つはC末端ドメインに見出された。これら5つのCa2+はいずれもEFハンドとは異なるモチーフで結合しており(図4)、既知のCa2+結合タンパク質との比較において興味がもたれる。また、基質BAの電子密度はC末端ドメインの2つのCa2+の近傍に確認された。
図3 PAD4(Ca2+結合型PAD4と基質BAとの複合体)の構造。(a)単量体、(b)2量体
図4 PAD4のCa2+および基質結合部位の構造。
(a)C末端ドメインに結合している2つのCa2+(Ca1, Ca2)と基質BA、
(b)N末端ドメインに結合している3つのCa2+(Ca3, Ca4, Ca5)
(a)C末端ドメインに結合している2つのCa2+(Ca1, Ca2)と基質BA、
(b)N末端ドメインに結合している3つのCa2+(Ca3, Ca4, Ca5)
またPAD4はいずれの結晶においても結晶学的な2回軸で関連付けられるもう1つの分子に近接していた(図5(a))。この2分子の分子量はゲルろ過クロマトグラフィーや動的光散乱の結果とも一致し、その形状(図5(b))はX線小角散乱法によって決定された低分解能溶液構造(図5(c))と一致しているので、PAD4は結晶中で近接している2分子(2量体)(図3(b))で機能することが示唆される。
図5 PAD4(Ca2+非結合型)の構造。
(a)結晶構造の電子密度図(楕円で囲まれた部分が2分子のPAD4(2量体)に相当)、(b)2量体の空間充填モデル、(c)X線溶液散乱法で決定されたPAD4(Ca2+非結合型)の低分解能溶液構造(DAMモデル)。
(a)結晶構造の電子密度図(楕円で囲まれた部分が2分子のPAD4(2量体)に相当)、(b)2量体の空間充填モデル、(c)X線溶液散乱法で決定されたPAD4(Ca2+非結合型)の低分解能溶液構造(DAMモデル)。
4.N末端ドメイン
N末端ドメインは1〜300番目のアミノ酸から構成されているが、このドメインはさらに2つのサブドメイン(sub-domain 1とsub-domain 2)に分けることができる(図3(a))。Sub-domain 1とsub-domain 2はともに免疫グロブリン様の構造をとり、sub-domain 1には56PPAKKKST63という核移行シグナル(NLS)が存在している。この領域は2つのβ-strandsを結ぶ分子表面のループ領域に存在しているが、いずれの結晶もこの領域はdisorderしてその電子密度は確認できなかった。また、冒頭で述べた関節リウマチ感受性の一塩基多型はG55S、V82A、G112Aというアミノ酸置換を引き起こすが、これらの置換部位はすべてsub-domain 1に局在し、C末端ドメインに存在する活性部位(後述)からは遠く離れている。さらに、G55S、V82A、G112Aの置換はCa2+の結合や2量体形成にも関与していないので、これらの関節リウマチ感受性の一塩基多型はPAD4の活性には影響しないものと思われる。
一方、sub-domain 2の分子表面には3つのCa2+がクラスターを形成して結合している(図3(a))。この領域はCa2+非結合型PAD4ではdisorderしているが、Ca2+結合型PAD4とCa2+結合型PAD4−基質複合体ではCa2+が結合することにより安定化され、α-helix構造が誘起されている(図4(b))。Ca2+の結合(配位)にはAsp388と153番目から179番目の酸性のアミノ酸が関与しているが、Asp155やAsp157やAsp176のように一つのアミノ酸の主鎖と側鎖が複数のCa2+に配位する様式も認められる。このようなCa2+がクラスターを形成してタンパク質表面に結合する様式はProtein kinase C(PKC)のC2ドメインにも認められる。PKCのC2ドメインはCa2+結合による表面電荷の変化によってリン脂質との結合が制御されているが、sub-domain 2のCa2+クラスターの生理学的な機能はまだよくわかっていない。
5.C末端ドメイン
301〜663番目のアミノ酸残基からなるC末端ドメイン(図3(a))は、α/βプロペラ構造と呼ばれる擬似の5回軸で関係付けられる5つのββαβmoduleをとっている(図6)。α/βプロペラ構造は、L−アルギニンをL−シトルリンに変換するarginine deiminase(ADI)[12]やL−アルギニンとグリシンからguanidinoacetic acidを合成するarginine:glycine amidinotransferase(AT)[13]などのアルギニン修飾酵素において共通に認められる構造である。このα/βプロペラ構造の中心部にはクレフトが形成されていて、基質BAと1つのCa2+(Ca1)が結合している。もう1つのCa2+(Ca2)は2番目のββαβモジュール(modules 2、図6)のα-helixとβ-strandの間に存在している。
N末端ドメインは1〜300番目のアミノ酸から構成されているが、このドメインはさらに2つのサブドメイン(sub-domain 1とsub-domain 2)に分けることができる(図3(a))。Sub-domain 1とsub-domain 2はともに免疫グロブリン様の構造をとり、sub-domain 1には56PPAKKKST63という核移行シグナル(NLS)が存在している。この領域は2つのβ-strandsを結ぶ分子表面のループ領域に存在しているが、いずれの結晶もこの領域はdisorderしてその電子密度は確認できなかった。また、冒頭で述べた関節リウマチ感受性の一塩基多型はG55S、V82A、G112Aというアミノ酸置換を引き起こすが、これらの置換部位はすべてsub-domain 1に局在し、C末端ドメインに存在する活性部位(後述)からは遠く離れている。さらに、G55S、V82A、G112Aの置換はCa2+の結合や2量体形成にも関与していないので、これらの関節リウマチ感受性の一塩基多型はPAD4の活性には影響しないものと思われる。
一方、sub-domain 2の分子表面には3つのCa2+がクラスターを形成して結合している(図3(a))。この領域はCa2+非結合型PAD4ではdisorderしているが、Ca2+結合型PAD4とCa2+結合型PAD4−基質複合体ではCa2+が結合することにより安定化され、α-helix構造が誘起されている(図4(b))。Ca2+の結合(配位)にはAsp388と153番目から179番目の酸性のアミノ酸が関与しているが、Asp155やAsp157やAsp176のように一つのアミノ酸の主鎖と側鎖が複数のCa2+に配位する様式も認められる。このようなCa2+がクラスターを形成してタンパク質表面に結合する様式はProtein kinase C(PKC)のC2ドメインにも認められる。PKCのC2ドメインはCa2+結合による表面電荷の変化によってリン脂質との結合が制御されているが、sub-domain 2のCa2+クラスターの生理学的な機能はまだよくわかっていない。
5.C末端ドメイン
301〜663番目のアミノ酸残基からなるC末端ドメイン(図3(a))は、α/βプロペラ構造と呼ばれる擬似の5回軸で関係付けられる5つのββαβmoduleをとっている(図6)。α/βプロペラ構造は、L−アルギニンをL−シトルリンに変換するarginine deiminase(ADI)[12]やL−アルギニンとグリシンからguanidinoacetic acidを合成するarginine:glycine amidinotransferase(AT)[13]などのアルギニン修飾酵素において共通に認められる構造である。このα/βプロペラ構造の中心部にはクレフトが形成されていて、基質BAと1つのCa2+(Ca1)が結合している。もう1つのCa2+(Ca2)は2番目のββαβモジュール(modules 2、図6)のα-helixとβ-strandの間に存在している。
図6 C末端ドメインのα/βプロペラ構造。
擬似の5回軸で関係づけられる5つのββαβモジュールは、
modules 1、2、3、4、5として色分けして示されている。
擬似の5回軸で関係づけられる5つのββαβモジュールは、
modules 1、2、3、4、5として色分けして示されている。
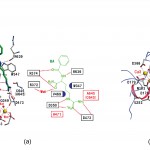
図4(a)に基質BA認識の様子を示す。基質BAの アルギニン側鎖のグアニジノ基の窒素原子は2つの酸性残基Asp350とAsp473によって認識され、その近傍にはCys645(Ala645)とHis471が位置している。また、アルギニン側鎖のアルキル基(-CH2-CH2-CH2-)の部分にはTrp347とVal469との疎水的な相互作用も認められる。一方、基質BAの主鎖部分ではBAのアルギニンのカルボニル酸素と窒素がそれぞれArg374とArg639の主鎖カルボニルと水素結合を形成している。また、BAのベンゾイル基のカルボニル酸素とArg374の間にも水素結合が認められる。PAD4はタンパク質中のアルギニン残基(ペプチジルアルギニン)を基質として認識するが、遊離のL−アルギニンは認識しない。これは基質BAのベンゾイル基のカルボニル酸素に相当する部分が遊離のL−アルギニンには存在しないためで、この部分の分子認識がPAD4の基質認識の特異性に重要であることが示唆される。また、基質BAのアルギニン側鎖のグアニジノ基部分を認識する残基(Asp350, His471, Asp473, Cys645)がPAD4の活性残基である。
6.酵素の活性化
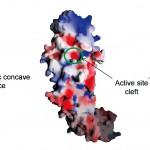
Ca2+非結合型PAD4、Ca2+結合型PAD4およびCa2+結合型PAD4−基質BA複合体の三者の構造を比較すると、PAD4の活性部位周辺の高次構造がCa2+が結合することによってダイナミックに変化することがわかる。Ca2+非結合型PAD4の活性部位周辺はdisorderしていて非常に不安定ある。そのために活性部位付近に大きな窪み(concave surface)が現れ、その表面に酸性のアミノ酸残基が露出している(図7(a))。この酸性の窪みの表面に2つのCa2+(Ca1とCa2)が結合すると、disorderしていた領域が安定化して活性部位(active cleft)が誘起される(図7(b))。また、Ca2+結合型PAD4とCa2+結合型PAD4-BA複合体の活性部位の構造は一致しているので、2つのCa2+(Ca1とCa2)が結合することによって酵素の活性部位が誘起され、そこに基質BAが結合することがわかる(図7(c))。
Ca2+非結合型PAD4、Ca2+結合型PAD4およびCa2+結合型PAD4−基質BA複合体の三者の構造を比較すると、PAD4の活性部位周辺の高次構造がCa2+が結合することによってダイナミックに変化することがわかる。Ca2+非結合型PAD4の活性部位周辺はdisorderしていて非常に不安定ある。そのために活性部位付近に大きな窪み(concave surface)が現れ、その表面に酸性のアミノ酸残基が露出している(図7(a))。この酸性の窪みの表面に2つのCa2+(Ca1とCa2)が結合すると、disorderしていた領域が安定化して活性部位(active cleft)が誘起される(図7(b))。また、Ca2+結合型PAD4とCa2+結合型PAD4-BA複合体の活性部位の構造は一致しているので、2つのCa2+(Ca1とCa2)が結合することによって酵素の活性部位が誘起され、そこに基質BAが結合することがわかる(図7(c))。
図7 分子表面の静電ポテンシャル。
(a)Ca2+非結合型PAD4、(b)Ca2+結合型PAD4、(c)Ca2+結合型PAD4と基質BAとの複合体。
基質BAは緑色の空間充填モデルで表示。図はすべて図3(a)と同じ方向から描かれている。
2つのCa2+(Ca1とCa2)の結合が活性部位の形成(誘起)に必須であることを実験的に証明するために、Ca1とCa2に配位するアミノ酸残基をそれぞれアラニンに置換した変異体を作成してPAD活性を測定した。その結果、E351Aを除くすべての変異体でPAD活性の消失が確認された。さらに、N末端ドメインに結合しているCa2+の配位に関与する残基の中で唯一C末端ドメインに存在するAsp388(Ca3に配位)をアラニンに置換した変異体では活性の失活が認められないので、PAD4の活性発現にはCa1とCa2の結合が必須であることがわかる。
これまでPAD4によるアルギニン残基のシトルリン化になぜCa2+が必要なのかわからなかったが、今回のX線結晶構造解析によって、Ca2+の結合が活性部位を誘起して酵素反応の場(active cleft)の形成に関与していることが明らかとなった。Ca2+依存性の酵素としてはこれまでにシステインプロテアーゼのCalpain[14]やタンパク質中のLysとGlnのイソペプチド結合を形成するTransglutaminase[15]の立体構造が報告されている。しかし、これらの酵素ではCa2+の結合により活性部位の構造変化は認められるものの(Calpainでは予測されるものの)、PAD4のようにdisorderしていた領域が安定化されて活性部位が誘起されるといったダイナミックな構造変化は認められていない。したがって、今回のX線結晶構造解析で示されたCa2+によるPAD4の構造変化はこれまでにないまったく新しい酵素の活性機構であるといえる。
(a)Ca2+非結合型PAD4、(b)Ca2+結合型PAD4、(c)Ca2+結合型PAD4と基質BAとの複合体。
基質BAは緑色の空間充填モデルで表示。図はすべて図3(a)と同じ方向から描かれている。
2つのCa2+(Ca1とCa2)の結合が活性部位の形成(誘起)に必須であることを実験的に証明するために、Ca1とCa2に配位するアミノ酸残基をそれぞれアラニンに置換した変異体を作成してPAD活性を測定した。その結果、E351Aを除くすべての変異体でPAD活性の消失が確認された。さらに、N末端ドメインに結合しているCa2+の配位に関与する残基の中で唯一C末端ドメインに存在するAsp388(Ca3に配位)をアラニンに置換した変異体では活性の失活が認められないので、PAD4の活性発現にはCa1とCa2の結合が必須であることがわかる。
これまでPAD4によるアルギニン残基のシトルリン化になぜCa2+が必要なのかわからなかったが、今回のX線結晶構造解析によって、Ca2+の結合が活性部位を誘起して酵素反応の場(active cleft)の形成に関与していることが明らかとなった。Ca2+依存性の酵素としてはこれまでにシステインプロテアーゼのCalpain[14]やタンパク質中のLysとGlnのイソペプチド結合を形成するTransglutaminase[15]の立体構造が報告されている。しかし、これらの酵素ではCa2+の結合により活性部位の構造変化は認められるものの(Calpainでは予測されるものの)、PAD4のようにdisorderしていた領域が安定化されて活性部位が誘起されるといったダイナミックな構造変化は認められていない。したがって、今回のX線結晶構造解析で示されたCa2+によるPAD4の構造変化はこれまでにないまったく新しい酵素の活性機構であるといえる。
7.PAD4によるヒストン認識と配列特異性
これまで述べてきた基質BAはPAD4の人工の基質で、生体内の本来の基質はタンパク質(ヒストン)のアルギニン残基(ペプチジルアルギニン)である。そこで、PAD4によるヒストン認識機構を明らかにすることも踏まえ、Ca2+結合型PAD4とヒストンのN末端領域にあるアルギニン残基を含む10残基のペプチドとの複合体のX線結晶構造解析を行った。なお、用いたヒストンのN末端ペプチドは次の3つである。
ヒストンH3のN末端ペプチド1(Peptide H3-1)
Ac-4KQTARKSTGG13
ヒストンH3のN末端ペプチド2(Peptide H3-2)
Ac-14KAPRKQLATK23
ヒストンH4のN末端ペプチド(Peptide H4)
Ac1-SGRGKGGKGL10
これまで述べてきた基質BAはPAD4の人工の基質で、生体内の本来の基質はタンパク質(ヒストン)のアルギニン残基(ペプチジルアルギニン)である。そこで、PAD4によるヒストン認識機構を明らかにすることも踏まえ、Ca2+結合型PAD4とヒストンのN末端領域にあるアルギニン残基を含む10残基のペプチドとの複合体のX線結晶構造解析を行った。なお、用いたヒストンのN末端ペプチドは次の3つである。
ヒストンH3のN末端ペプチド1(Peptide H3-1)
Ac-4KQTARKSTGG13
ヒストンH3のN末端ペプチド2(Peptide H3-2)
Ac-14KAPRKQLATK23
ヒストンH4のN末端ペプチド(Peptide H4)
Ac1-SGRGKGGKGL10
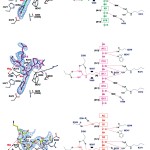
この3つのペプチドにおいて、ターゲットのアルギニン残基の位置をNとし、そのN末端側の残基を順次(N-1)、(N-2)、(N-3)、C末端側を順次(N+1)、(N+2)、(N+3)とすると(図8、9(b))、いずれの10残基のヒストンペプチドも(N-2)から(N+2)の5残基のアミノ酸残基の電子密度が確認され、そのうち(N-2)から(N+1)の4残基がPAD4によって認識されている(図8)。さらに、その認識様式を比較してみると、いずれのヒストンペプチドも(N-2)から(N+1)の4残基の主鎖原子が認識されていることがわかる。一方、ヒストンペプチドの側鎖の認識はターゲットとなるアルギニン残基の側鎖と(N-2)の残基の側鎖だけで[ペプチドH3-2の(N-2)はAlaで、γ位に酸素原子がないために側鎖の相互作用は観測されない]、基質認識の配列特異性はきわめて低い。ただ、(N-2)の残基の側鎖が大きいとPAD4と立体障害を起こすことが考えられるので、(N-2)の残基は小さな側鎖を持つことが必須であると考えられる。事実、これまでに報告されている5つのPAD4によるシトルリン化されるヒストンペプチドの(N-2)の位置はGly、Ala、Serなどのアミノ酸残基が占めている。
図8 PAD4による基質(ヒストンペプチド)認識。
(a)ヒストンペプチドH3-1、(b)ヒストンペプチドH3-2、(c)ヒストンペプチドH4。
(a)ヒストンペプチドH3-1、(b)ヒストンペプチドH3-2、(c)ヒストンペプチドH4。
8.ヒストン認識の構造(コンフォメーション)特異性
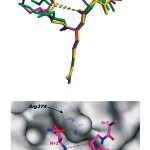
次に、ヒストンペプチドの構造(コンフォメーション)を比較してみると、その構造は共通してペプチド鎖は折れ曲がったβターン様の構造をとっていることがわかる(図9(b))。いずれのヒストンペプチドも(N-2)から(N+2)の5残基のアミノ酸残基の電子密度が確認され、そのうち(N-2)から(N+1)の4残基がPAD4によって認識されている(図8)。したがって、(N+2)の残基は、電子密度は確認されるもののPAD4には認識されていないことになる。これはヒストンペプチドがPAD4の活性部位周辺の形状とArg374との相互作用によって折れ曲がったβターン様の構造が誘起されるためである(図9(b))。そのためにヒストンペプチド分子内の(N-2)のカルボニル酸素と(N+2)の窒素の間に水素結合様の弱い相互作用が形成され、(N+2)の残基が安定化される。なお、典型的なβターン構造では(N-1)のカルボニル酸素と(N+2)の窒素の間に水素結合が形成されているが、ヒストンペプチドのβターン様の構造では両原子間の距離は水素結合の距離よりも長く、水素結合の形成までには至っていない。
次に、ヒストンペプチドの構造(コンフォメーション)を比較してみると、その構造は共通してペプチド鎖は折れ曲がったβターン様の構造をとっていることがわかる(図9(b))。いずれのヒストンペプチドも(N-2)から(N+2)の5残基のアミノ酸残基の電子密度が確認され、そのうち(N-2)から(N+1)の4残基がPAD4によって認識されている(図8)。したがって、(N+2)の残基は、電子密度は確認されるもののPAD4には認識されていないことになる。これはヒストンペプチドがPAD4の活性部位周辺の形状とArg374との相互作用によって折れ曲がったβターン様の構造が誘起されるためである(図9(b))。そのためにヒストンペプチド分子内の(N-2)のカルボニル酸素と(N+2)の窒素の間に水素結合様の弱い相互作用が形成され、(N+2)の残基が安定化される。なお、典型的なβターン構造では(N-1)のカルボニル酸素と(N+2)の窒素の間に水素結合が形成されているが、ヒストンペプチドのβターン様の構造では両原子間の距離は水素結合の距離よりも長く、水素結合の形成までには至っていない。
図9 ヒストンペプチドの構造。破線はヒストンペプチド内に形成されているβターン様の相互作用。
(a)3つのヒストンペプチド(H3-1,H3-1,H4)の比較、
(b)PAD4の分子表面で認識されているヒストンペプチドH3-2の構造。
(a)3つのヒストンペプチド(H3-1,H3-1,H4)の比較、
(b)PAD4の分子表面で認識されているヒストンペプチドH3-2の構造。
このように、通常は一定の決まった構造をとっていないヒストンのN末端領域もPAD4に認識されると折れ曲がったβターン様の構造をとるようになる。このことはPAD4の基質特異性を考える上で非常に重要である。PAD4は二次構造を形成している領域のアルギニン残基は認識しないことが生化学的な実験から明らかにされているが[16]、これは一定の決まった構造をとっていない柔軟性に富む領域にあるアルギニン残基のみがPAD4によって認識されるためである。PAD4のターゲットとなるアルギニン残基が一定の二次構造を形成している領域にあると、折れ曲がったβターン様の構造が誘起できないのでPAD4には認識されない。すなわち、PAD4によるヒストン認識では、アミノ酸残基の配列特異性はきわめて低いが、構造(コンフォメーション)の特異性は非常に高いといえる。ヒストンのN末端領域にあるアルギニン残基が前後のアミノ酸配列にあまり関係なく優先的に認識されてPAD4の格好のターゲットとなるのは、まさにPAD4のこのようなヒストン(基質)認識の特異性に起因しているといえる。
9.まとめ
今回、私たちはCa2+非結合型PAD4、Ca2+結合型PAD4、Ca2+結合型PAD4−基質複合体の構造を決定し、3者の構造比較から「C末端ドメインに結合するCa2+によって酵素の活性部位が誘起され、そこに基質分子が結合する」という新規の酵素活性化機能を明らかにしてきた。このようなCa2+による酵素活性化機構はこれまでに構造が決定されているCa2+依存性の酵素ではまったく認められていないので、まったく新しい酵素活性化機構としてたいへん興味がもたれる。また、ヒストンペプチドとの複合体の構造解析からは、「通常は一定の決まった構造をとっていないヒストンのN末端領域もPAD4に認識されると折れ曲がったβターン様の構造をとるようになる」ことや、「PAD4によるヒストン認識では、配列特異性は低いが、構造の特異性は高い」ことなど、新規のヒストン認識に関する構造科学的な知見が数多く得られた。これらの研究成果は今後の真核生物の転写研究に大いに役立つものと思われ、今後の展開が楽しみである。さらに、今回得られた基質認識に関する構造学的知見は、関節リウマチの新規薬剤の開発に必要なPAD4の阻害剤候補物質の設計にも有効で、PAD4−阻害剤複合体のX線結晶構造解析を通じて、より特異性に優れ阻害活性の高い化合物の創製が期待される。
今回、私たちはCa2+非結合型PAD4、Ca2+結合型PAD4、Ca2+結合型PAD4−基質複合体の構造を決定し、3者の構造比較から「C末端ドメインに結合するCa2+によって酵素の活性部位が誘起され、そこに基質分子が結合する」という新規の酵素活性化機能を明らかにしてきた。このようなCa2+による酵素活性化機構はこれまでに構造が決定されているCa2+依存性の酵素ではまったく認められていないので、まったく新しい酵素活性化機構としてたいへん興味がもたれる。また、ヒストンペプチドとの複合体の構造解析からは、「通常は一定の決まった構造をとっていないヒストンのN末端領域もPAD4に認識されると折れ曲がったβターン様の構造をとるようになる」ことや、「PAD4によるヒストン認識では、配列特異性は低いが、構造の特異性は高い」ことなど、新規のヒストン認識に関する構造科学的な知見が数多く得られた。これらの研究成果は今後の真核生物の転写研究に大いに役立つものと思われ、今後の展開が楽しみである。さらに、今回得られた基質認識に関する構造学的知見は、関節リウマチの新規薬剤の開発に必要なPAD4の阻害剤候補物質の設計にも有効で、PAD4−阻害剤複合体のX線結晶構造解析を通じて、より特異性に優れ阻害活性の高い化合物の創製が期待される。
ここで紹介した研究は、文部科学省「タンパク3000プロジェクト」(中核機関:横浜市立大学)の助成のもと、横浜市立大学の山田道之教授(現、名誉教授)及び日高雄二博士(近畿大学理工学部)と共同で行ったものである。
なお、本研究は放射光X線の利用なしでは決して成功しませんでした。SPring-8でのデータ収集にご協力いただいた山本雅貴博士、熊坂 崇博士(現、東工大)、三浦圭子博士、また、PF-ARでのデータ収集にご協力いただいた若槻壮市教授、加藤龍一助教授、松垣直宏博士、五十嵐教之博士、鈴木 守博士(現、大阪大学蛋白質研究所)に、この場をお借りして深く感謝いたします。
参考文献
[1]G.A.Schellekens,B.A.W.de Jong,F.H.J. van den Hoogen,L.B.A.van de Putte and W.J.van Venrooij : J. Clin.Invest.101(1998)273-281.
[2]A.Suzuki,et al.: Nat.Genet.34(2003)395-402.
[3]M.A.van Boekel,E.R.Vossenaar,F.H.van den Hoogen and W.J.van Venrooij : Arthritis Res.4(2002)87-93.
[4]J.A.Hill, S.Southwood,A.Sette,A.M.Jevnikar,D.A.Bell and E.Cairns : J. Immunol.171(2003)538-541.
[5]U.M.Bauer,S.Daujat,S.J. Nielsen,K.Nightingale and T.Kouzarides : EMBO Rep.3(2002)39-44.
[6]H.Wang,Z.Q.Huang,L.Xia,Q.Feng, H.Erdjument-Bromage,B.D.Strahl,S.D.Briggs,C.D.Allis,J.Wong, P.Tempst and Y.Zhang : Science 293(2001)853-857.
[7]G.L.Cuthbert,et al.: Cell 118(2004)545-553.
[8]Y.Wang,et al.:Science 306(2004)279-283.
[9]K.Arita,H.Hashimoto,T.Shimizu,K.Nakashima,M.Yamada and M.Sato : Nat.Struct.Mol.Biol.11(2004)777-783.
[10]K.Arita,T.Shimizu,H.Hashimoto,Y.Hidaka,M.Yamada and M.Sato : Proc.Natl.Acad.Sci.USA 103(2006)5291-5296.
[11]K.Arita,H.Hashimoto,T.Shimizu,M.Yamada and M.Sato : Acta Crystallogr.D59(2003)2332-2333.
[12]K.Das,et al.: Structure 12(2004)657-667.
[13]A.Humm,E.Fritsche,S.Steinbacher and R.Huber : EMBO J. 16(1997)3373-3385.
[14]A.Khorchid and M.Ikura : Nat. Struct.Biol.9(2002)239-241.
[15]B.Ahvazi,H.C.Kim,S.H.Kee,Z.Nemes and P.M.Steinert : EMBO J.21(2002)2055-2067.
[16]E.Tarcsa,L.N.Marekov,G.Mei,G.Melino,S.E.Lee and P.M.Steinert : J.Biol.Chem.271(1996)30709-30716.
[1]G.A.Schellekens,B.A.W.de Jong,F.H.J. van den Hoogen,L.B.A.van de Putte and W.J.van Venrooij : J. Clin.Invest.101(1998)273-281.
[2]A.Suzuki,et al.: Nat.Genet.34(2003)395-402.
[3]M.A.van Boekel,E.R.Vossenaar,F.H.van den Hoogen and W.J.van Venrooij : Arthritis Res.4(2002)87-93.
[4]J.A.Hill, S.Southwood,A.Sette,A.M.Jevnikar,D.A.Bell and E.Cairns : J. Immunol.171(2003)538-541.
[5]U.M.Bauer,S.Daujat,S.J. Nielsen,K.Nightingale and T.Kouzarides : EMBO Rep.3(2002)39-44.
[6]H.Wang,Z.Q.Huang,L.Xia,Q.Feng, H.Erdjument-Bromage,B.D.Strahl,S.D.Briggs,C.D.Allis,J.Wong, P.Tempst and Y.Zhang : Science 293(2001)853-857.
[7]G.L.Cuthbert,et al.: Cell 118(2004)545-553.
[8]Y.Wang,et al.:Science 306(2004)279-283.
[9]K.Arita,H.Hashimoto,T.Shimizu,K.Nakashima,M.Yamada and M.Sato : Nat.Struct.Mol.Biol.11(2004)777-783.
[10]K.Arita,T.Shimizu,H.Hashimoto,Y.Hidaka,M.Yamada and M.Sato : Proc.Natl.Acad.Sci.USA 103(2006)5291-5296.
[11]K.Arita,H.Hashimoto,T.Shimizu,M.Yamada and M.Sato : Acta Crystallogr.D59(2003)2332-2333.
[12]K.Das,et al.: Structure 12(2004)657-667.
[13]A.Humm,E.Fritsche,S.Steinbacher and R.Huber : EMBO J. 16(1997)3373-3385.
[14]A.Khorchid and M.Ikura : Nat. Struct.Biol.9(2002)239-241.
[15]B.Ahvazi,H.C.Kim,S.H.Kee,Z.Nemes and P.M.Steinert : EMBO J.21(2002)2055-2067.
[16]E.Tarcsa,L.N.Marekov,G.Mei,G.Melino,S.E.Lee and P.M.Steinert : J.Biol.Chem.271(1996)30709-30716.
有田 恭平 ARITA Kyouhei
横浜市立大学大学院 国際総合科学研究科 生体超分子科学専攻
現在、学術振興会特別研究員(京都大学大学院工学研究科分子工学専攻)
〒230-0045 横浜市鶴見区末広町1-7-29
TEL:045-508-7227 FAX:045-508-7365
e-mail : kyouhei@tsurumi.yokohama-cu.ac.jp
清水 敏之 SHIMIZU Toshiyuki
横浜市立大学大学院 国際総合科学研究科 生体超分子科学専攻
〒230-0045 横浜市鶴見区末広町1-7-29
TEL:045-508-7226 FAX:045-508-7365
e-mail : shimizu@tsurumi.yokohama-cu.ac.jp
橋本 博 HASHIMOTO Hiroshi
横浜市立大学大学院 国際総合科学研究科 生体超分子科学専攻
〒230-0045 横浜市鶴見区末広町1-7-29
TEL:045-508-7227 FAX:045-508-7365
e-mail : hash@tsurumi.yokohama-cu.ac.jp
佐藤 衛 SATO Mamoru
横浜市立大学大学院 国際総合科学研究科 生体超分子科学専攻
〒230-0045 横浜市鶴見区末広町1-7-29
TEL:045-508-7225 FAX:045-508-7365
e-mail : msato@tsurumi.yokohama-cu.ac.jp
横浜市立大学大学院 国際総合科学研究科 生体超分子科学専攻
〒230-0045 横浜市鶴見区末広町1-7-29
TEL:045-508-7227 FAX:045-508-7365
e-mail : hash@tsurumi.yokohama-cu.ac.jp
佐藤 衛 SATO Mamoru
横浜市立大学大学院 国際総合科学研究科 生体超分子科学専攻
〒230-0045 横浜市鶴見区末広町1-7-29
TEL:045-508-7225 FAX:045-508-7365
e-mail : msato@tsurumi.yokohama-cu.ac.jp