Volume 29, No.3 Pages 195 - 199
1. 最近の研究から/FROM LATEST RESEARCH
クライオ電顕で捕捉したシャペロニン複合体形成の構造基盤
Structural basis of chaperonin complex formation captured by cryo-electron microscopy
東京大学 大学院農学生命科学研究科 Department of Biotechnology, The University of Tokyo
- Abstract
- シャペロニンは細胞内のタンパク質の恒常性に重要な役割を果たすタンパク質複合体である。大腸菌のシャペロニンGroELの構造と機能はよく研究されているが、蓋状分子GroESの結合とATP加水分解による大きな構造変化などの理由で、シャペロニン複合体の構造解析には制約があり、複合体形成段階およびその動きに関する理解は限られている。本研究では、クライオ電子顕微鏡により、ATPおよびAMP-PNP存在下で、2種類の好熱性水素酸化細菌由来のシャペロニン複合体の立体構造を解析した。高分解能のフットボール型複合体の構造に加えて、今まで報告されていなかった非対称フットボール型複合体も捕捉することに成功した。それらの構造をもとに、弾丸型複合体からフットボール型複合体への移行における重要なステップであるtrans-GroESの結合のメカニズムを解釈した。
1. はじめに
タンパク質の立体構造に関する基本的な情報はアミノ酸配列に記されているが、細胞内の環境は複雑であり、新生ポリペプチドは機能を持つ正しい構造にまでフォールドできず、ミスフォールドや凝集に陥ることがあり得る。グループIに属するシャペロニンGroEL(真核生物の場合、HSP60とも呼ばれる)はホモ7量体のリング2つで構成され、蓋状タンパク質GroESと結合することによって、リング内側に形成されるチャンバー(空洞)の中に入り込んだ基質タンパク質のフォールディングを補助する(図1)。GroELのプロトマー(ひとつのユニット)は赤道、中間、頂点の3つのドメインで構成される[1][1] K. Braig et al.: Nature 371 (1994) 578-586.。赤道ドメインのヌクレオチド結合部位におけるATPの加水分解により、頂点ドメインが外向きに大きく構造変化し、保存された2本のαヘリックス(ヘリックスH/I)がGroESの可動ループ(mobile loop)と呼ばれる部位と相互作用することが知られている[2, 3][2] N. A. Ranson et al.: Cell 107 (2001) 869-879.
[3] D. K. Clare et al.: Cell 149 (2012) 113-123.。生理活性を持つGroEL-GroES複合体は、両者が1:1で結合する弾丸型(GroEL-GroES)と、1:2で結合するフットボール型(GroEL-GroES2)の主に2種類が存在する。GroELはその分子量や対称性などの性質から、昔から電子顕微鏡や結晶構造解析のモデル試料として研究に用いられてきた。しかし、GroELのATPase活性は、同じ側のリングにおける正の協同性(positive cooperativity)と異なるリングにおける負の協同性(negative cooperativity)が存在するため、2分子目のGroESが結合した状態を捕捉するには変異体作成などの工夫が必要だった。そこで、我々は2種類の至適温度の異なる好熱性水素酸化細菌(Hydrogenophilus thermoluteolus TH-1 (Hp1): 52°C[4][4] H. Arai, Y. Shomura, Y. Higuchi and M. Ishii: Microbiol. Res. Announc. 7 (2018) e00857-18.、Hydrogenobacter thermophilus TK-6 (Hb6): 70°C[5][5] H. Arai, H. Kanbe, M. Ishii and Y. Igarashi: J. Bacteriol. 192 (2010) 2651-2652.)由来のシャペロニンに着目し、クライオ電子顕微鏡を用いた単粒子解析によりそれらの立体構造を解析した[6][6] Z. Liao et al.: Structure 32 (2024) 679-689.。
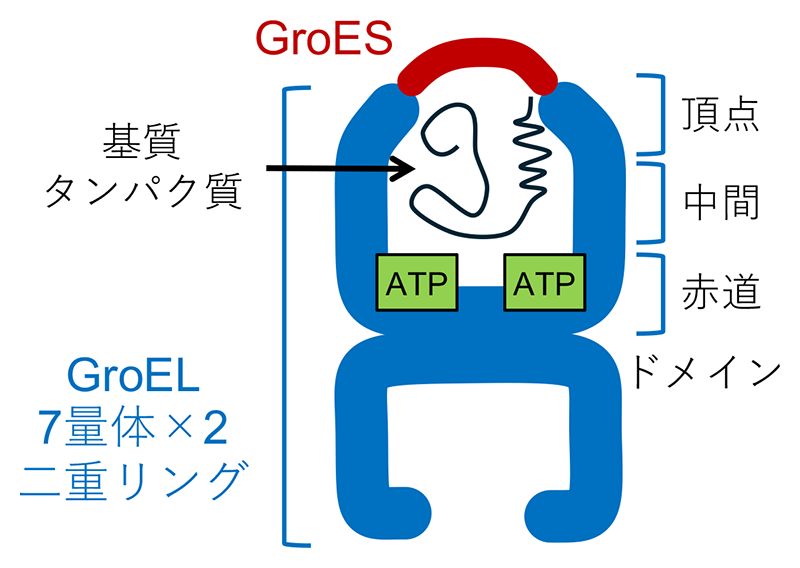
図1 弾丸型のGroEL-GroES複合体の模式図。
2. Hp1シャペロニンのフットボール型複合体
Hp1由来のシャペロニンについては、GroELは培養した水素細菌から直接精製したものを、GroESは大腸菌の組換えタンパク質をそれぞれ用いた。近年ではネガティブ染色法による電子顕微鏡解析を行うことも減っているが、GroELのような大きいタンパク質複合体の場合、クライオ測定のためのサンプル純度を確認するには役に立った。ATP存在下でのHp1 GroEL-GroES複合体をネガティブ染色電顕で観察したところ、明確なコントラストで多数の複合体が目視で確認できた(図2(A))。クライオ測定のためにグリッド作成条件検討を行なった結果、気液界面で複合体粒子が失活していることが判明したため、最終的に界面活性剤を用いることにより、グリッドのホールの周辺部で比較的分厚い氷を作らせて、ホールの中心からやや外れた位置で撮影することにした(図2(B))。
意外なことに、大腸菌のGroEL-GroESでは大部分の粒子が弾丸型複合体として観察されるのに対して、Hp1の場合は90%以上の粒子がフットボール型複合体であった。Hp1 GroELは大腸菌GroELに比べて熱安定性の高いタンパク質であり、そのATPase活性は常温で至適温度(65°C)の半分以下に低下する。そのため、常温で調製した試料において分解能の高いフットボール型複合体の構造解析が可能になったのではないかと考えている。Hp1シャペロニンの電顕グリッドは粒子の均一性が優れており、1,200枚の画像を用いて2.3 Å分解能でフットボール型複合体のマップを構築できた(図2(C、D))。Hp1シャペロニンの全体構造は大腸菌シャペロニンのフットボール型複合体と類似していたが、GroEL/GroESの界面にあるヘリックスH/Iと可動ループの部分は結晶構造と若干違うことが分かった(図2(E、F))。大腸菌シャペロニンでフットボール型複合体の構造を得るにはヌクレオチド結合部位の変異体あるいは加水分解されないATP類似体を用いる必要があったが、Hp1のシャペロニンを用いることにより、野生型タンパク質を用いて、ATPを添加した条件でフットボール型複合体の構造の捕捉に成功したことになる。
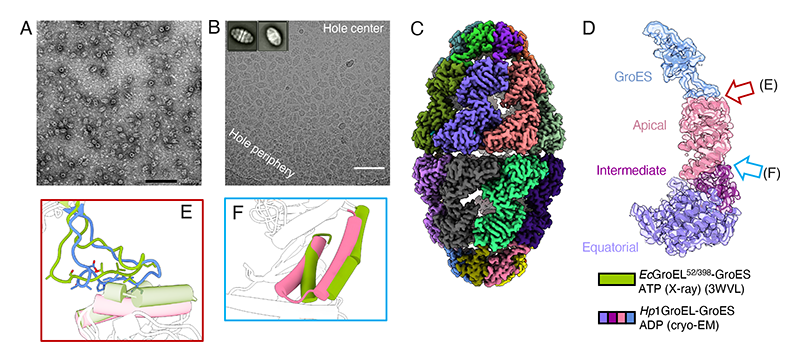
図2 Hp1シャペロニン複合体の電顕解析
(A) Hp1 GroEL-GroESのネガティブ染色電顕画像。(B) クライオ電顕画像と代表的な2D平均像。(C) Hp1フットボール型複合体の電顕マップ。(D) Hp1フットボール型複合体プロトマーのモデルとマップ。(E、F) 既報の結晶構造との構造比較。Hp1 GroEL-GroESをピンクと青で、ヌクレオチド結合部位に変異を導入した大腸菌GroEL-GroESの構造を緑色で表す。
3. Hb6シャペロニンの非対称フットボール型複合体
続いて、Hp1でのノウハウを用いて別種の水素細菌であるHb6由来のシャペロニン複合体の構造解析に挑んだ。しかし、Hb6はHp1よりもさらに菌体生育至適温度が高く、そのGroELは常温においてATPase活性がほとんど見られないことが判明した。大腸菌の組換えタンパク質として調製したHb6のGroEL-GroESを常温で混合して電顕測定を行ったところ、粒子のうちごく一部は複合体を形成しているように見えたが、ヌクレオチド結合部位のATPは未分解状態で、非常に不安定な構造であることが判明した(未発表データ)。グリッド作成前の加熱温度と各種のヌクレオチド類似体を検討した結果、AMP-PNP(非加水分解性ATPアナログ)を添加して45°Cで調製した試料を用いることにした。単粒子解析の結果、興味深いことに、これまで報告されていた弾丸型複合体と半フットボール型複合体に加えて、GroELの両側にGroESが結合しているが明らかに上下非対称の複合体が存在していた(図3(A))。我々はこれを非対称フットボール型(asymmetric football)複合体と名付けて、弾丸型複合体とともに構造精密化を行った(図3(B))。非対称フットボール型複合体のうち、一般的な弾丸型複合体と似た構造を持つ側のリング(ここではcis-ringと定義する)においては、弾丸型と同様の相互作用でGroESが安定に結合していた。一方、その逆側のリング(trans-ring)では、GroELの頂点ドメインからGroESまで非常にフレキシブルな状態であることが分かった。Cis-ringの構造に重ね合わせると、trans-ringではGroELの頂点ドメインとGroESがともに安定な位置からずれていることが分かる(図3(C、D))。
また、赤道ドメインのヌクレオチド結合部位においては、両方のリングでAMP-PNPおよびMg2+イオンと思われる電顕マップが確認できた。Trans-ringではさらにATP加水分解に必要なK+イオンのピークも観測された。それに関連して、trans-ringではS-loopとterminal strandsと呼ばれる部分がcis-ringとは異なるコンフォメーションをとっていることが判明した(図3(E))。これらの部分はGroELのチャンバーに入り込んだ基質と相互作用すると考えられており、trans-ringではcis-ringとチャンバーの大きさも異なっていることが分かった。
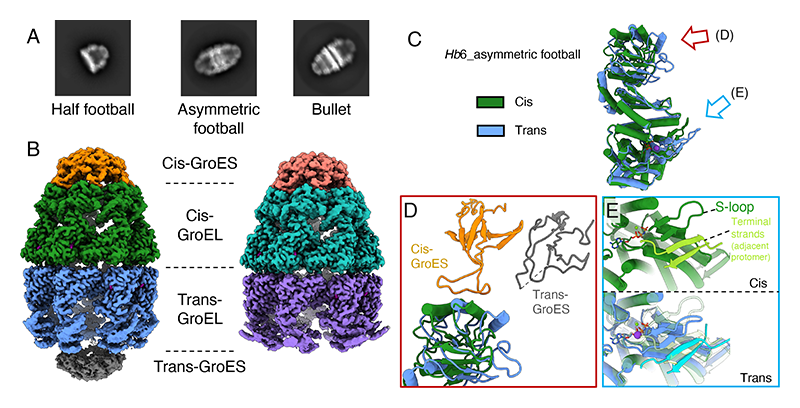
図3 Hb6シャペロニン複合体の電顕解析
(A) 複合体のタイプ別2D平均像。(B) 非対称フットボール型複合体(左)と弾丸型複合体(右)の電顕マップ。(C、D、E) 非対称フットボール型複合体のcis-ringとtrans-ringのプロトマー間の構造比較。
4. Trans-GroESの結合による複合体の構造変化
これまでに大腸菌のGroELとGroESの結合中間状態を捕捉する試みはいくつか行われているが[2, 3][2] N. A. Ranson et al.: Cell 107 (2001) 869-879.
[3] D. K. Clare et al.: Cell 149 (2012) 113-123.、cis-GroESに比べてtrans-GroESの結合-解離平衡を左右する因子はさらに複雑であるため、その詳細はいまだによく理解されていない。シャペロニン複合体を制御するメカニズムの一つとして、cis-ringとtrans-ringの接点である赤道ドメインの上下で交差するGroELプロトマー間の相互作用が重要であることが分かっている。ここには複数の保存された残基が存在しており、その片方のリングにおいてATPの加水分解にともないプロトマーが回転する際に、塩橋や疎水性相互作用を通じて反対側のリングとコミュニケーションすると考えられている。我々の結果では、非対称フットボール型複合体のリング間相互作用のパターンは、Hp1のように一般的な対称型フットボール型複合体とは異なり、むしろ弾丸型に類似していることが分かった(図4(A、B))。非加水分解性のAMP-PNPでは、赤道ドメインの構造変化を引き起こせなかったと思われる。
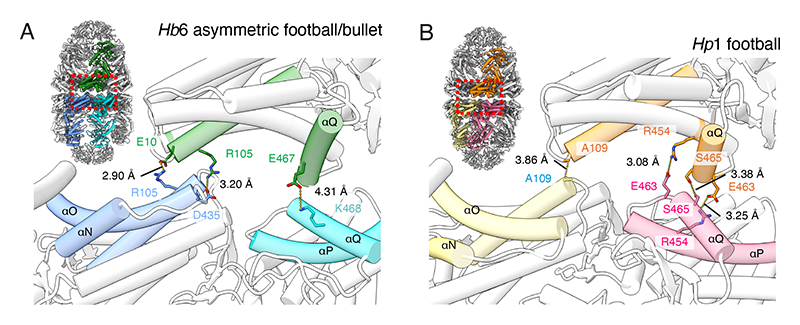
図4 水素細菌シャペロニン複合体のリング間の相互作用の比較
(A) Hb6シャペロニンの非対称フットボール型と弾丸型複合体に共通した相互作用。(B) Hp1の対称フットボール型複合体の相互作用。
一方で、Hb6の複合体のtrans-ring内での構造を比較したところ、意外なことに、非対称フットボール型複合体のtrans-ringの頂点ドメインは弾丸型に比べて外向きに構造変化していた(図5(A、B))。弾丸型のtrans-ringで塩橋を形成してプロトマー同士を安定化していたK336-E257は、非対称フットボール型のtrans-ringでは遠く離れている。これらの結果から、弾丸型複合体のtrans-ringにGroESが結合すると、相互作用界面の構造変化が起こることが推測できた。そのため、GroESの結合が起こっている時点では、trans-ringの体積は膨大する。このようにゆるく蓋を閉めるようなGroESの結合様式は、基質タンパク質の収容に十分なスペースを提供できると予想される。その後、対称型フットボール複合体に構造変化していく段階ではチャンバーのスペースの収縮が起こり、これがタンパク質をフォールディングさせる物理的な力として働いていると推測される。
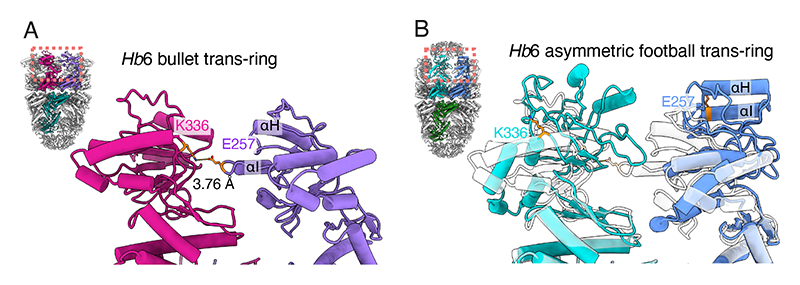
図5 Hb6シャペロニン複合体のtrans-ring内の相互作用の比較
(A) 弾丸型複合体の相互作用。(B) 非対称フットボール型複合体の相互作用。
我々はHb6のシャペロニン複合体の解析途中で、trans-ringに注目してクラス分けを試みたが、GroES結合界面付近のマップはどうしても綺麗に解析できなかった。おそらくプロトマー間のコンフォメーションの非均一性によるものと思われる。なお、symmetry expansion法を用いたプロトマーごとの解析結果は論文中で示している[6][6] Z. Liao et al.: Structure 32 (2024) 679-689.。このようなフレキシビリティも、上記のメカニズムに加え、基質タンパク質の収容に重要だと推測される。
本研究はSPring-8大学院生提案型課題2022A2767、2022B2556、2023A2763、2023B2545により実施しました。またSPring-8での電顕測定に関して重松秀樹先生、Gerle Christoph先生、Chai Gopalasingam先生の協力、助言をいただきました。KEKでの電顕測定は千田俊哉先生、安達成彦先生、川崎政人先生、守屋俊夫先生の協力をいただきました。また、一部のグリッド作成、電顕でのスクリーニングは胡桃坂仁志先生、滝沢由政先生、小笠原光雄先生の協力をいただきました。ディスカッションおよびサンプル提供で荒川孝俊先生、亀谷将史先生、新井博之先生、石井正治先生に協力をいただきました。多くの共同研究者の方々、実験施設のサイエンティストの方々にこの場を借りて感謝申し上げます。
参考文献
[1] K. Braig et al.: Nature 371 (1994) 578-586.
[2] N. A. Ranson et al.: Cell 107 (2001) 869-879.
[3] D. K. Clare et al.: Cell 149 (2012) 113-123.
[4] H. Arai, Y. Shomura, Y. Higuchi and M. Ishii: Microbiol. Res. Announc. 7 (2018) e00857-18.
[5] H. Arai, H. Kanbe, M. Ishii and Y. Igarashi: J. Bacteriol. 192 (2010) 2651-2652.
[6] Z. Liao et al.: Structure 32 (2024) 679-689.
東京大学 大学院農学生命科学研究科
応用生命工学専攻 酵素学研究室
(現所属)Laboratory of Prof. Andreas Martin
Department of Molecular & Cell Biology; QB3 Institute;
Howard Hughes Medical Institute
University of California at Berkeley
570 Stanley Hall
Berkeley, CA 94720-3220
e-mail : zengwei_liao_mark0@berkeley.edu
東京大学 大学院農学生命科学研究科
応用生命工学専攻 酵素学研究室
〒113-8657 東京都文京区弥生1-1-1
TEL : 03-5841-5151
e-mail : asfushi@mail.ecc.u-tokyo.ac.jp








