Volume 27, No.4 Pages 306 - 310
1. 最近の研究から/FROM LATEST RESEARCH
(SPRUC 2022 Young Scientist Award受賞 研究報告)
放射光X線で照らす時計タンパク質KaiCの概日振動メカニズム
Oscillation Mechanism of Clock Protein KaiC Revealed by Synchrotron X-ray Crystallography
自然科学研究機構 分子科学研究所 協奏分子システム研究センター Research Center of Integrative Molecular Systems, Institute for Molecular Science
- Abstract
- シアノバクテリアの概日時計は、時計タンパク質KaiA、KaiB、KaiCによって構成される。ペースメーカーとして働くKaiCは、N末端側のC1ドメインにてATP加水分解反応を触媒して時計システム全体のペースを決定し、C末端側のC2ドメインではリン酸化・脱リン酸化によりサイクル反応を進行させる。C1ドメインとC2ドメインの間には、密接な連携メカニズムが存在すると長年考えられてきたが、リン酸化サイクル反応におけるC2ドメインの構造変化の全容が解明されていなかったために、その仕組みは明らかになっていなかった。
SPring-8において構築したKaiC結晶構造ライブラリーに基づき、リン酸化サイクル反応全体を原子分解能で可視化することによって、リン酸化部位の状態に依存して二次構造が転移するスイッチ機構を見出した。このリン酸化スイッチ機構は、C1ドメインのATP加水分解反応によって生じる構造変化と連携していた。この2つのドメインにまたがって、それぞれのサイクル反応を統合するアロステリックな分子構造変化は、「概日周期をもった振動性」をはじめとする概日時計の機能に必須であった。
1. 原子スケールで時を刻むタンパク質、KaiC
地球の自転による24時間周期の環境変動に対応するため、生命は体内の計時システムを発達させ、細胞活動をリズミックに制御している。これは概日時計と呼ばれ、時計遺伝子、時計タンパク質によって構成される。概日時計に関わる時計遺伝子がショウジョウバエで発見されて以降、シアノバクテリア、昆虫、植物、ヒトを含む哺乳類など、概日時計を支える生体分子が相次いで発見されている。
時計遺伝子が誘起する細胞活動の概日振動性は、転写-翻訳フィードバックループ機構によって説明がなされている。これは、細胞核内の時計遺伝子の転写-翻訳によって生成された時計タンパク質が、核外へと移動して他の関連因子との複合体形成・化学反応を通して状態変化し、核内に戻って自身の転写活性を抑制、しだいに時計タンパク質の濃度が低下するに伴い転写活性の抑制効果がおさまり、ふたたび時計タンパク質の生成量が増加するという一連の振動メカニズムである。
研究の進展に伴い、転写-翻訳フィードバックループ機構の背後で実際に時刻情報を生み出す化学的基盤として、時計タンパク質の活性部位で進行する触媒反応(リン酸化、脱リン酸化、ATP加水分解など)が着目されはじめ、現在では時計タンパク質の動態研究とりわけ立体構造解析が主要な研究の舞台のひとつとなっている。
シアノバクテリアの概日時計[1][1] M. Ishiura et al.: Science 281 (1998) 1519-1523.は、時計タンパク質KaiA、KaiB、KaiCによって駆動されている。2005年には、これら3種のタンパク質とATP(アデノシン三リン酸)を混合するだけで、24時間周期の振動反応が試験管内で再現できることが明らかにされ、転写-翻訳フィードバックループに依存せずとも[2][2] J. Tomita et al.: Science 307 (2005) 251-254.、時計タンパク質のみで概日振動を引き起こせることが実証された[3][3] M. Nakajima et al.: Science 308 (2005) 414-415.。以降、生化学、生物物理学、構造生物学にまたがるin vitro実験が様々な角度から試みられ、KaiA-KaiB-KaiCの離合集散現象が、KaiCのATP加水分解反応および自己リン酸化・自己脱リン酸化反応によって指揮されていることが明らかにされてきた[4-10][4] T. Nishiwaki et al.: EMBO J. 26 (2007) 4029-4037.
[5] K. Terauchi et al.: Proc. Natl. Acad. Sci. USA 104 (2007) 16377-16381.
[6] S. Akiyama et al.: Molecular Cell 29 (2008) 703-716.
[7] J. Abe et al.: Science 349 (2015) 312-316.
[8] Y. Chang et al.: Science 349 (2015) 324-328.
[9] R. Tseng et al.: Science 355 (2017) 1174-1180.
[10] J. Snijder et al.: Science 355 (2017) 1181-1184.。
しかしながらペースメーカーとして働くKaiCの内部で、どのようなメカニズムが働き、時刻情報が生成されているかは謎に包まれたままであった。KaiCは、C1、C2という2つのドメインによって構成され、ATPを結合して六量体を形成し機能する(図1A)。C1ではATP加水分解反応(C1-ATPaseサイクル)が進行し、その反応速度により時計システム全体のペースが規定される[5,6][5] K. Terauchi et al.: Proc. Natl. Acad. Sci. USA 104 (2007) 16377-16381.
[6] S. Akiyama et al.: Molecular Cell 29 (2008) 703-716.。C2ではS431とT432という2つのアミノ酸残基が順にリン酸化・脱リン酸化されるサイクル反応(C2-リン酸化サイクル)が進行し(ST→SpT→pSpT→pST→ST:SはS431、TはT432、pはリン酸化した状態を指す)、これによりKaiCの状態が24時間周期で変動する[4][4] T. Nishiwaki et al.: EMBO J. 26 (2007) 4029-4037.(図1B)。C1とC2のあいだには密接な連携があり、どちらか片方でも反応活性が損なわれると、KaiCは計時機能を失う。シアノバクテリア概日時計の中枢を成すこのC1-C2連携メカニズムは長年明らかにされてこなかった。
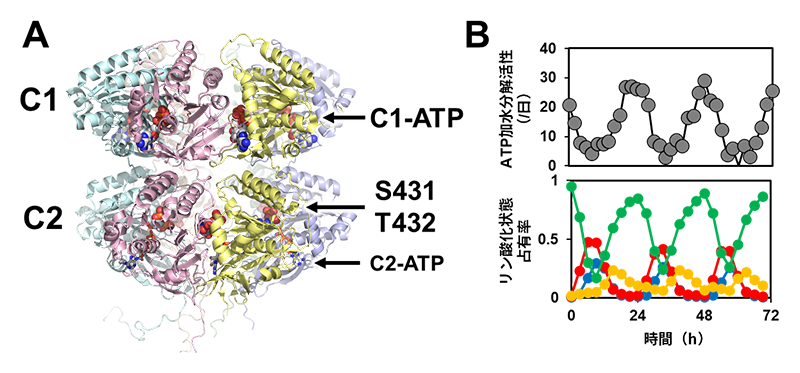
図1 時計タンパク質KaiCの全体構造とATP加水分解活性およびリン酸化状態の概日振動
(A)KaiC六量体の2つのドメイン(C1、C2)と基質ATPおよびリン酸化部位(S431、T432)の配置、(B)ATP加水分解活性およびリン酸化状態(緑:KaiC-ST、赤:KaiC-SpT、青:KaiC-pSpT、黄:KaiC-pST)の振動。
2. 時計タンパク質KaiCのX線結晶構造解析
KaiCがみせる「1日」にわたる構造変化の全容を明らかにするためには、異なるリン酸化状態にあるKaiCを調製する必要があった。あらかじめ溶液中でリン酸化状態をコントロールしたKaiC試料を用いて結晶化に取り組み、広範な条件探索の末、計13種類におよぶKaiC六量体構造を結晶中にて観察することができた。
SPring-8 BL44XUでの回折実験で得られた2.2-3.1 Å分解能の電子密度マップに基づき、KaiC六量体に含まれる全プロトマーのリン酸化状態を逐一同定した。その結果、本研究で構築したKaiC構造ライブラリーには、六量体すべてが脱リン酸化した状態のKaiC-ST、T432をGluに変異させてpT432を模倣したKaiC-SpT(SE)、全てのリン酸化部位が修飾を受けたKaiC-pSpT、T432が脱リン酸化したKaiC-pSTが含まれており、C2-リン酸化サイクル全体をカバーすることができた。他にも、部分的に脱リン酸化が進行したKaiC六量体(KaiC-pSpT/pSTやKaiC-pST/ST)も捉えられており、溶液中でみられるKaiCのリン酸化状態変化の特徴が再現されていた。
また同様に、C1ドメインの活性部位の状態を判別したところ、ATP加水分解反応の結果生じた生成物ADP(アデノシン二リン酸)を結合しているKaiC六量体が構造ライブラリーに含まれていることが分かった。これは、C1とC2の2種類の反応サイクルにおける重要な反応ステップがはじめて同時に捉えられたことを示している。
3. KaiCを駆動する2つのドメインの連携メカニズム
KaiC構造ライブラリーの詳細な観察の結果、C1-C2連携メカニズムを支える3つの構造基盤を明らかにすることができた。
リン酸化部位であるS431とT432は、C2ドメインのプロトマー界面に位置している(図1A)。その近傍に、リン酸化状態の変化に応じて二次構造が転移する領域が存在することがはじめて明らかとなった。KaiC-STでは、この領域はヘリックス構造をとっていた(図2A)。このヘリックス構造は、T432のリン酸化の影響を受けず、KaiC-SpTにおいても同様に観察された。KaiC-pSpTでは、同じ領域に特定の二次構造は存在せず、ほどけた構造がみつかった。これはT432の脱リン酸化によって変化することはなく、KaiC-pSTでも同様のほどけた構造がみられた。このヘリックス構造-ほどけた構造の転移は、S431およびpS431の側鎖体積の違いや周辺との水素結合パターンの差異によって生じており、S431のリン酸化状態のみに依存して切り替わるスイッチ機構(C2-リン酸化スイッチ)として働くことが分かった。
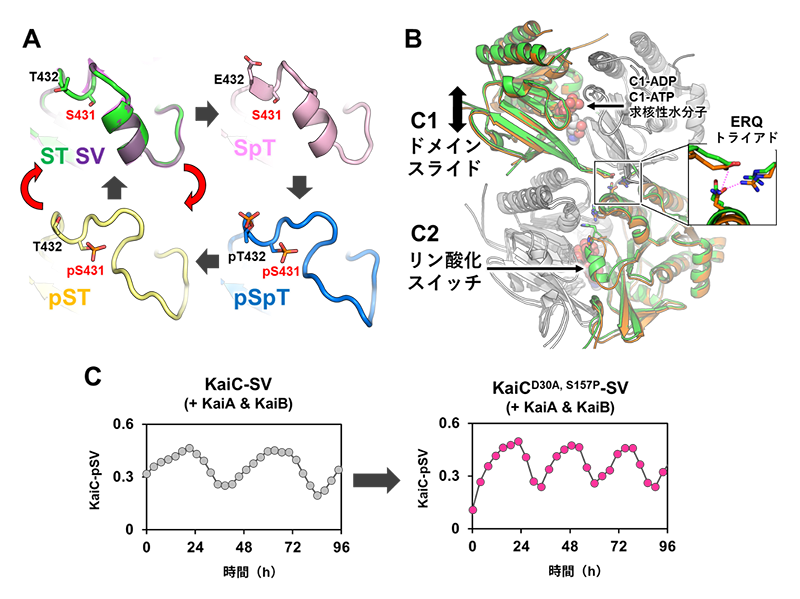
図2 KaiCの概日振動の根幹をなすC2-リン酸化サイクルとC1-ATPaseサイクルのあいだのアロステリックな連携
(A)C2-リン酸化サイクルにともなうヘリックス構造-ほどけた構造の転移(緑:KaiC-ST、赤:KaiC-SpT、青:KaiC-pSpT、黄:KaiC-pST、紫:KaiC-STに重ねて描画したKaiC-SV)、野生型のKaiCは黒色の矢印に沿って4状態を経由するがKaiC-SVは赤色の矢印に沿って2状態のみを経由して概日振動を生み出す、(B)隣り合う2つのプロトマーを六量体の中心(リング内径)からみたときに見出されるC1-C2連携を生み出す構造的要素の配置(緑:C1-ATPと求核性水分子を結合したKaiC-ST、橙:C1-ADPを結合したKaiC-pST)、(C)S431単一リン酸化変異体(KaiC-SV)のKaiA、KaiB存在下での47時間周期振動(灰色)とATPase活性化変異によって短縮化された29時間周期振動(ピンク色)。
生化学的に同定されていた4つのリン酸化状態(ST, SpT, pSpT, pST)が2種類の構造に分類されるという結果は意外なものではあったが、このC2-リン酸化スイッチがC1とのドメイン間連携において重要な役割を果たすこともあわせて明らかになった(図2B)。C1は、プロトマー界面に結合している生成物ADP(C1-ADP)と新たな基質ATP(C1-ATP)の交換時に、隣り合うプロトマー間でドメイン全体が上下方向にシフトする構造変化(C1-ドメインスライド)を起こすこと、そしてC1-ドメインスライドがATP加水分解で反応物となる求核性水分子の配置と対応していることが判明した。
C2-リン酸化スイッチとC1-ドメインスライドの構造変化は、水素結合の切り替え機構によって仲介されていた(図2B挿入図)。C2に由来する中性のQ394が、C1ドメインに属する塩基性のR217と酸性のE214のあいだを行き来できるようにドメイン界面がデザインされていたのである(これら3つのアミノ酸残基をまとめてERQトライアドと名付けた)。
このようにC1-C2連携メカニズムは、たった3つの構造的要素、すなわちC2-リン酸化スイッチ、C1-ドメインスライド、ERQトライアドによる仲介、によって単純化して記述できることが判明した。C1-ATPaseサイクルは、ATPの結合、求核性水分子の配置、リン酸結合の開裂、ADPの解放、といった多数の素過程から成っている。C2-リン酸化サイクルについても、少なくとも4種のリン酸化状態が必須と考えられてきた。KaiCはこうした活性部位における化学反応をアロステリックな運動へと統合的に変換することで、分子全体のリズミックな構造変化すなわち概日振動性を獲得しているのである。
4. S431単一リン酸化変異体によって実証されたKaiC概日振動メカニズム
C2-リン酸化スイッチの発見は、KaiCがリン酸化主要4状態を経由せずとも、S431のリン酸化状態変化のみによってKaiCが概日振動できる可能性を示していた。そこで、T432を非リン酸化アミノ酸であるValに変異させたS431単一リン酸化変異体(KaiC-SV)を調製し、溶液条件中での検証を行った。KaiC-SVはリン酸化によりKaiC-pSVとなるが、KaiAおよびKaiBと混合したうえで、その存在比を電気泳動によって求めたところ、47時間周期の振動がみられた(図2C)。また概日スケールから逸脱して伸長した振動周期は、C1-ATPaseサイクルの速度を操作することで再び短縮できた。ATP加水分解活性を上昇させるアミノ酸変異(D31AとS157P)をC1ドメインに追加導入することで、振動周期は29時間周期になった。しかもこの変異体はシアノバクテリア細胞内でも機能することができた。これにより、C1(ドメインスライド)-C2(リン酸化スイッチ)連動性に基づくS431単一リン酸化変異体が、概日振動体として機能しうることが実証できた。
ヒト、昆虫、植物の時計タンパク質は、配列・構造が異なるものの、いずれもが複数のリン酸化部位を有している。このリン酸化部位の複数性・多重性こそが概日振動を生み出す化学的基盤であるとする考えもある。しかしS431単一リン酸化変異体が概日振動体として機能できるという事実は、個々の全てのリン酸化・脱リン酸化反応が等価に必須機能を担っているというよりは、時計タンパク質の全体構造の連動性のなかでそれぞれが異なる意義を有している可能性を示している。
5. 概日時計の時空間スケールとSPring-8/SACLA
本研究は、KaiCの2つのドメインそれぞれの反応サイクルを成す多数の素過程をできるだけ多く、X線結晶構造解析によって捉えることを目標に開始した。その結果、KaiCの概日振動は原子スケールの機構で動作するC1-C2連携メカニズムによって説明できた[11-13][11] Y. Furuike et al.: Science Advances 8 (2022) eabm8990.
[12] Y. Furuike et al.: Proc. Natl. Acad. Sci. USA 355 (2022) e2119627119.
[13] A. Mukaiyama et al.: Biochem. J. 479 (2022) 1505-1515.。この成果は、SPring-8/SACLAが幅広い時空間スケールの生命現象の解明に有力な性能を備えていることを示している。
SPring-8では、進行速度をうまく制御した秒~分スケールのタンパク質結晶相反応の原子構造追跡研究や一分子回折計測研究が重ねられてきた実績があり、SACLAの供用開始以後には、フェムト秒スケール(10-15秒)に至るタンパク質の超高速動態研究が加速度的に進められている。一方で、概日時計のような1日(105秒)スケールで極端にゆっくり進行する生命現象の解明においてもSPring-8/SACLAは有力であったように、放射光X線を用いた計測によって時間生物学をはじめとする長い時間スケールで進行する原子・分子構造変化の追跡研究が今後進展していくことが見込まれる。
謝辞
本研究は、分子科学研究所の向山厚助教、甲田信一助教、欧陽東彦博士、Damien SIMON博士、秋山修志教授、斉藤真司教授、大阪大学蛋白質研究所の山下栄樹准教授、名古屋大学の伊藤(三輪)久美子特任助教、西脇(大川)妙子准教授、近藤孝男特別教授、立命館大学の寺内一姫教授との共同研究です。的確なご指導、有意義なご議論をいただきありがとうございました。
X線回折実験は、SPring-8のBL44XUにおいて課題番号2017A6700、2017B6700、2018A6700、2018B6700、2019A6700、2019B6700、2020A6700、2020A6500、2017A6702、2017B6702、2018A6802、2018B6802、2019A6902、2019B6902、2020A6502のもとで行いました。BL44XUビームラインスタッフの皆さま、構造生物学ビームラインの関係者の皆さまにお礼を申し上げます。
また本研究は、科研費(17H06165、19K16061、18K06171)、AMED創薬等ライフサイエンス研究支援基盤事業(創薬等先端技術支援基盤プラットフォーム事業)JP20am0101072(support number 0583)等の支援を受けて実施しました。
参考文献
[1] M. Ishiura et al.: Science 281 (1998) 1519-1523.
[2] J. Tomita et al.: Science 307 (2005) 251-254.
[3] M. Nakajima et al.: Science 308 (2005) 414-415.
[4] T. Nishiwaki et al.: EMBO J. 26 (2007) 4029-4037.
[5] K. Terauchi et al.: Proc. Natl. Acad. Sci. USA 104 (2007) 16377-16381.
[6] S. Akiyama et al.: Molecular Cell 29 (2008) 703-716.
[7] J. Abe et al.: Science 349 (2015) 312-316.
[8] Y. Chang et al.: Science 349 (2015) 324-328.
[9] R. Tseng et al.: Science 355 (2017) 1174-1180.
[10] J. Snijder et al.: Science 355 (2017) 1181-1184.
[11] Y. Furuike et al.: Science Advances 8 (2022) eabm8990.
[12] Y. Furuike et al.: Proc. Natl. Acad. Sci. USA 355 (2022) e2119627119.
[13] A. Mukaiyama et al.: Biochem. J. 479 (2022) 1505-1515.
自然科学研究機構 分子科学研究所
協奏分子システム研究センター
総合研究大学院大学
〒444-8585 愛知県岡崎市明大寺町大字西郷中39番地
TEL : 0564-55-7336
e-mail : furuike@ims.ac.jp








