Volume 26, No.2 Pages 109 - 118
1. 最近の研究から/FROM LATEST RESEARCH
長期利用課題報告1
イオンポンプの結晶構造解析
Crystal Structure Analysis of Ion Pumps
東京大学 定量生命科学研究所 Institute for Quantitative Biosciences, The University of Tokyo
- Abstract
- イオンポンプ蛋白質の作動機構を原子構造に基づいて完全に理解することを目標に長期利用課題を遂行している。筋小胞体Ca2+ポンプに関しては、今や変異体の構造決定を行える段階にある。そこで、Ca2+イオン通路の細胞質側ゲートであるアミノ酸残基Glu309をGlnに置換した変異体のE2(Ca2+非存在下でCa2+に対し低親和性)状態の構造を決定した。この構造は、予想に反し、天然蛋白質とは著しく異なっていた。すなわち、Ca2+ポンプは酸素と窒素を区別する能力を持つことが示され、E2→E1(Ca2+に対し高親和性)遷移のメカニズムが明らかになるという予想外の進展をもたらした。
1. はじめに
2018A期からBL41XUにおいて、長期利用課題「イオンポンプの結晶構造解析」を、研究室のメンバー(金井隆太助教、椛島佳樹助教)とともに遂行した。この長期利用課題はイオンポンプ蛋白質の作動機構の原子構造による完全な理解を目指すものである。ここで言うイオンポンプ蛋白質は生体膜に埋め込まれた膜蛋白質であり、生体におけるエネルギーの通貨であるATP(アデノシン三燐酸)がADP(アデノシン二燐酸)と無機燐酸に加水分解されるときに放出されるエネルギーを利用し、濃度勾配に逆らって、生体膜を越えてイオンを運搬する(能動輸送する)蛋白質のことである。ATPを加水分解するという意味でATP水解酵素(ATPase)であり、反応サイクルの途中でATPからポンプ蛋白質へ燐酸が転移される(自己燐酸化)ため(図1)、P型ATPaseとも呼ばれる。「ATPの化学エネルギーを変換して」とはよく言われることだが、モーター蛋白質よりも簡単な系であり、「エネルギー変換とは何か」を考える上で理想的な研究対象と言える。イオンポンプは生体の活動の基盤を作る大変重要な蛋白質群である。例えば、神経の興奮は細胞内にナトリウムイオンが流入することによって起こるが、流入が起こるのはナトリウムイオンの濃度が膜を隔てて大きく違うからであり、その濃度差を作るのがナトリウムポンプの役割である。濃度勾配に逆らった輸送を可能にするためには、ATPの分解、燐酸転移といった化学反応と共役して2つのゲートを順番に開け閉めすることが必要である(2つのゲートが同時に開くことはないというのが肝心な点である)。従って、反応サイクルは多数のステップから成るが(図1)、その全ての反応中間体の原子構造を決定し、それに基づいて作動機構を解明することを目指している。具体的には(i)私達が世界に先駆けて構造決定に成功して以来、20年以上にわたって追求してきた筋小胞体Ca2+ポンプ(Ca2+-ATPase、sarco(endo)plasmic reticulum calcium ATPase(SERCA))の反応サイクル中間体の結晶構造解析、(ii)医学的生物学的にはより重要とも言え、そのために激しい国際競争が続いているナトリウムポンプ(Na+,K+-ATPase)の複数の状態の結晶構造解析、(iii)「結晶中の脂質二重膜の可視化」を行い、膜蛋白質が働く「場」である脂質二重膜とポンプ蛋白質との相互作用を解明すること、の3つのテーマから成る。それぞれ大きな進歩があったが、本報告ではPNAS誌に発表したテーマ(i)に関する論文[1][1] N. Tsunekawa, H. Ogawa, J. Tsueda, T. Akiba and C. Toyoshima: Proc. Natl. Acad. Sci. USA 115 (2018) 12722-12727.について紹介したい。
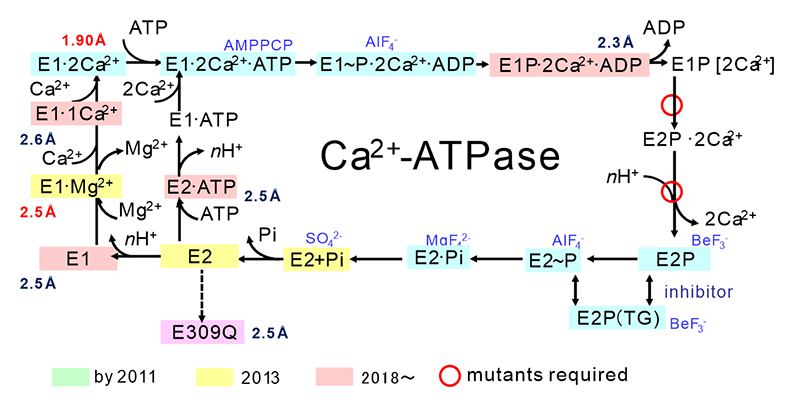
図1 Ca2+ポンプ(SERCA1a)の反応ダイアグラム。Ca2+ポンプはATPの化学エネルギーを利用し、ATP1分子の加水分解当たり2個のCa2+を細胞質から小胞体内腔へと輸送し、逆方向にプロトンを輸送する。Ca2+結合サイトがCa2+に対し高親和性で細胞質側を向いている状態をE1、低親和性で小胞体内腔側を向いている状態をE2と言う。E1P等のPは燐酸化を示す(Piは無機燐酸)。四角で囲った状態は構造決定済み。四角の外にはその状態を安定化するために用いた基質アナログと分解能を示す。nは2~3。
2. 筋小胞体Ca2+ポンプの構造研究の現状
私達は、筋小胞体Ca2+ポンプSERCA1a(速筋由来の成熟型が1aの意味)を対象に、2000年の最初の構造[2][2] C. Toyoshima, M. Nakasako, H. Nomura and H. Ogawa: Nature 405 (2000) 647-655.(E1・2Ca2+)以来、BL41XUを利用して、その反応過程のほぼ全体をカバーする10状態の結晶構造を決定した(図1)。また、4状態に関しては、結晶中の脂質二重膜を解像することによって、脂質二重膜は単に膜蛋白質を浮かべる海のようなものではなく、ポンプ機構の重要な部分として組み込まれていることを示し[3][3] Y. Norimatsu, K. Hasegawa, N. Shimizu and C. Toyoshima: Nature 545 (2017) 193-198.、イオン能動輸送機構の原子構造に基づく理解を推進してきた。ここからさらに理解を進めるためには、変異体の構造決定は避けて通れない課題である。SERCAに関しては、組み換え蛋白質の大量生産技術も確立しているので[4][4] C. Toyoshima, S. Iwasawa, H. Ogawa, A. Hirata, J. Tsueda et al.: Nature 495 (2013) 260-264.、結晶化に取り組むこと自体には大きな問題はない。2021年2月末の時点では欧州グループによる4つの変異体の構造が発表されているが[5-7][5] M. M. G. Geurts, J. D. Clausen, B. Arnou, C. Montigny, G. Lenoir et al.: Proc. Natl. Acad. Sci. USA 117 (2020) 31114-31122.
[6] A. Marchand, A.-M. L. Winther, P. J. Holm, C. Olesen, C. Montigny et al.: J. Biol. Chem. 283 (2008) 14867-14882.
[7] J. D. Clausen, M. Bublitz, B. Arnou, E. Montigny, C. Jaxel et al.: EMBO J. 32 (2013) 3231-3243.、メカニズムの解明に大きく貢献しているとは言い難い。生化学実験の条件と結晶化条件が離れていることも1つの要因であるが、得られた構造の意義を理解することは容易とは限らない。一方で、構造の理解のためには道具立ても重要であり、本稿で紹介する論文[1][1] N. Tsunekawa, H. Ogawa, J. Tsueda, T. Akiba and C. Toyoshima: Proc. Natl. Acad. Sci. USA 115 (2018) 12722-12727.では量子化学計算が必須であった。
3. Ca2+ポンプのイオン通路ゲート残基Glu309とその変異体
SERCA1aはATP1分子の加水分解に対し2個のCa2+を細胞質から小胞体内腔へと濃度勾配に逆らって運搬し、逆方向に2ないし3個のプロトンを輸送する[8][8] S. Zafar, A. Hussain, Y. Liu, S. Lewis and G. Inesi: Arch. Biochem. Biophys. 476 (2008) 87-94.。994残基の単一鎖から成る膜蛋白質であり、3つの良く分離した細胞質ドメイン(A、N、P)と10本の膜貫通ヘリックス(M1-M10)から成る(図2a)。燐酸化される残基はAspでありPドメインに位置する。ATPのアデニン環はPドメインに連結したNドメインに結合する(図2)。2個のCa2+の結合サイトは膜貫通領域にあり、M4-M6、M8にあるアミノ酸残基の酸素原子が配位することによってμMの親和性を生み出す(図3)。Ca2+結合サイトがCa2+に対し高親和性を持ち、細胞質側を向いている状態をE1、低親和性で内腔側を向いている状態をE2と言う(図1)。自己燐酸化(E1P、Pは燐酸を表す)に伴い、Ca2+のゲートを動かすactuatorとして機能するAドメインが~30°膜面に対し傾き、M1-M2ヘリックスを細胞質側に引き上げることで細胞質側通路が閉じられる[9][9] C. Toyoshima: Biochim. Biophys. Acta 1793 (2009) 941-946.。その結果、2個のCa2+は膜内に閉塞される。この状態でADPが外れるとE2P状態になり、内腔側ゲートが開きCa2+は放出される。その後、燐酸が蛋白質から外れた状態がE2(正確にはE2・nH+、上述のようにnは2ないし3)であり、プロトンは自然に外れてCa2+に対し高親和性のE1状態になる。イオンポンプはこのように膜貫通領域の細胞質側と内腔側(細胞外側)にある2つのゲートを、Pドメインで起こる化学反応と共役させて順番に開け閉めし、運搬するイオンに対する結合サイトの親和性を変化させることによって濃度勾配に逆らった輸送を行う。E1とE2の変化は主にM4ヘリックスを(手押しポンプのピストンのように)上下させるとともにM6ヘリックスのほどけている部分を回転させることによって成される(図3、詳細は後述)。
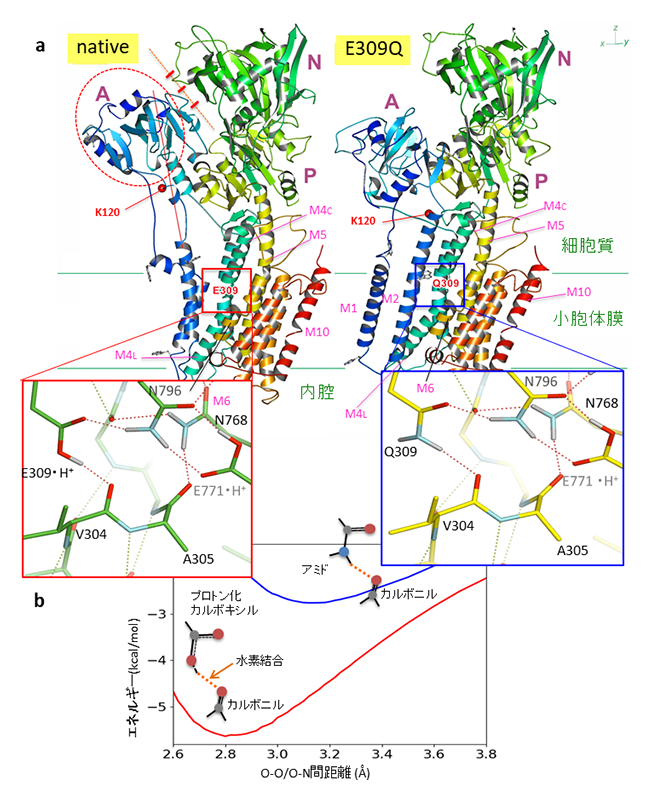
図2 Ca2+ポンプの天然型(native)とGlu309Gln変異体(E309Q)のE2状態の結晶構造(a)とGlu/Gln309-Val304間の水素結合の至適距離(b)。拡大図中のオレンジ色の点線は水素結合を表す。天然型ではA、N、Pの3つのドメインから成る細胞質側頭部はA-N間の7つの水素結合(赤の短い棒)のために閉じているが、変異体では開いている。また、Aドメインが大きく回転しているためにM1、M2ヘリックスの位置も大きく変わっている。M5ヘリックスの湾曲も天然型に比べ著しく小さい。天然型E2状態では蛋白質分解酵素(proteinase K)によりLys120(K120、赤丸)で切断されるが、変異体ではされない。(b)のプロットは量子化学計算(CCSD(T))によるプロトン化カルボキシル−カルボニル間とアミド−カルボニル間の水素結合エネルギーの距離依存性を示す。エネルギー的至適距離はプロトン化カルボキシルの場合2.83 Å、アミドの場合3.16 Åである。
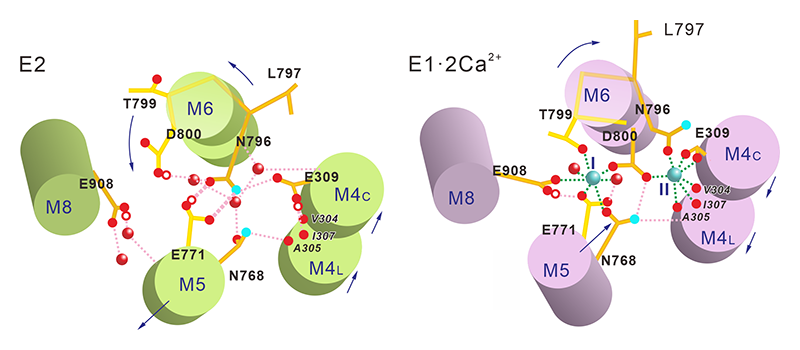
図3 Ca2+結合サイトを構成するアミノ酸残基のE2とE1・2Ca2+状態における配置と量子化学的に最適化された水素結合ネットワーク。細胞質側から見ている。水色の小球はCa2+、赤い小球は水分子、赤丸はプロトンを表す。イタリックの残基番号は主鎖カルボニル基が配位していることを示す。オレンジ色の点線は水素結合を、水色の点線はCa2+の配位を示す。最初にサイトICa2+が結合する。
SERCAの点変異体中最も注目されてきたものの1つにGlu309Gln変異体がある。細胞質にあるCa2+の細胞質側通路の入り口に位置し、イオン通路のゲートとなる。Glu309は2つある高親和性Ca2+結合サイトの2番目を構成する主要なアミノ酸残基であり、Ca2+結合の完成を細胞質ドメインに伝え、次の「ATPからの燐酸転移」(E1・2Ca2+ + ATP → E1P・2Ca2+ + ADP)のステップ(図1)に導く働きを持つ。M4ヘリックス中央のほどけた領域に位置するが(図2、3)、燐酸転移に伴い、その側鎖でイオン通路を塞ぐ役割を持つ。この残基のいかなる変異によってもCa2+の結合がSERCA1分子当たり1個になってしまうことがSERCA研究の初期に示され、Ca2+が1個だけ結合した状態の構造を決定できるかもしれないという希望のもとに構造研究も進められた[7][7] J. D. Clausen, M. Bublitz, B. Arnou, E. Montigny, C. Jaxel et al.: EMBO J. 32 (2013) 3231-3243.。ところが、デンマークグループの結晶構造では、両方のCa2+結合サイトともに埋まっており、しかもCa2+の配位にも大きな変化はなかった[7][7] J. D. Clausen, M. Bublitz, B. Arnou, E. Montigny, C. Jaxel et al.: EMBO J. 32 (2013) 3231-3243.。変異体の生化学的実験結果は、低温(0~4°C)で且つ低Ca2+濃度(μM)で得られたものであり、結晶化に使われる室温且つmM Ca2+の存在下とは大きく異なっていた、ということである。いずれにせよ、当初の期待は満足されず、得られた構造の意義も深く考えられたとはとても言い難い。
一方でGlu309は、Ca2+に対し低親和性のE2状態においては、プロトン化していると考えられてきた。Ca2+の高親和性結合に当たっては、負の荷電を持つ酸素原子がCa2+の周りに集中する(図3)。Ca2+放出後(E2状態)においては4つあるGluやAsp(Glu309、Glu771、Asp800、Glu908)のカルボキシル基はプロトン化し、負電荷の集中による反撥を中和する必要があると考えられる[10][10] K. Obara, N. Miyashita, C. Xu, I. Toyoshima, Y. Sugita et al.: Proc. Natl. Acad. Sci. USA 102 (2005) 14489-14496.。プロトン化は同時に、Ca2+に対する親和性を低下させる役割も担う[9][9] C. Toyoshima: Biochim. Biophys. Acta 1793 (2009) 941-946.。つまり、E2は厳密にはE2・nH+(nは2~3)と記述されるべきであるが、E2→E1遷移において、「4つあるカルボキシル基のどれからプロトンが外れるのか」、「プロトンの放出は構造変化の引き金なのか結果なのか」は分からないし、実験的にどうすればこの問題に迫れるのかも不明であった。
Glu309Gln変異体がE2状態で特異な振舞いをすることは、蛋白質分解酵素(proteinase K)による部分消化実験で示されていた。すなわち、もっと「激しい」置換であるはずのGlu309Ala変異体では切断パタンはE2型であるのに対し、Glu309Gln変異体はE1型であった[11][11] G. Inesi, D. Lewis, C. Toyoshima, A. Hirata and L. de Meis: J. Biol. Chem. 283 (2008) 1189-1196.。その差異は膜貫通ヘリックスM2の細胞質側の端にあるLeu119-Lys120(図2a)間のペプチド結合が蛋白質分解酵素で切断されるのがE2、されないのがE1ということなのだが、切れる切れないの構造上の差異はごく小さいものかもしれない。ともあれ、Glu309Gln変異体をCa2+非存在下で結晶化しようということになり、E2状態を確実なものにするために、SERCAをE2状態に固定する強力阻害剤thapsigargin存在下で結晶化した[1][1] N. Tsunekawa, H. Ogawa, J. Tsueda, T. Akiba and C. Toyoshima: Proc. Natl. Acad. Sci. USA 115 (2018) 12722-12727.。得られた構造(図2aのE309Qと4b)は、Ca2+結合部位を構成する残基の配置(側鎖の位置、配向も含めて)から言えば疑いもなくE2状態を示しているのだが、これまでに得られてきたE2状態(6種ある)の構造とは大きく異なっており、何がE2状態を形成するのに本質的に重要かを明らかにし、さらに、E2→E1遷移はどう起こるかを示すものであった。
4. Ca2+ポンプのE2状態
E2状態の構造(図2aのnativeと4a)の特徴としては、目立つ順に並べると[12][12] C. Toyoshima and H. Nomura: Nature 418 (2002) 605-611.、
(i)3つのドメインから成る細胞質側頭部は閉じてコンパクトな構造である。
(ii)SERCA蛋白質の背骨であるM5ヘリックスはM1方向に大きく湾曲している。
(iii)Ca2+の結合・放出に当たって膜に対し上下運動するM4ヘリックスは下がった位置にある。
(iv)Ca2+に配位する2つの残基(Asn796、Asp800)を含むM6ヘリックスは、Asp800付近でほどけているが、その部分はE2ではE1に比べ、~90°回転している。その結果、Ca2+結合時にはサイトIICa2+に配位するAsn796が、E2ではサイトICa2+に配位するGlu771と水素結合を作っている(図3、4)。
ことが挙げられる。この4つは勿論独立の事象ではない。Ca2+結合部位が低親和性であるのはM4が下がった位置にあるためであり、M5の湾曲はM4を下がった位置に持ってくるため(図5)、またM5の湾曲を保つためには細胞質側頭部は閉じて安定な構造をとることが必須と考えられていた。つまり、(i)−(iii)間の連関は認識されていたが、(iv)の意義は構造の安定性のためくらいの理解であった[10][10] K. Obara, N. Miyashita, C. Xu, I. Toyoshima, Y. Sugita et al.: Proc. Natl. Acad. Sci. USA 102 (2005) 14489-14496.。さらに、(i)に関連して、
(v)Aドメインの位置はE1とは方位角にして110°違う(膜面に対しほぼ垂直な軸の周りに回転している)。
(vi)そのことを反映して、M2ヘリックスは細胞質領域で一部ほどけており(図2a)、長くなっている。その結果、M4ヘリックスは膜に対して下がった位置にあることが可能である[13][13] C. Toyoshima, Y. Norimatsu, S. Iwasawa, T. Tsuda and H. Ogawa: Proc. Natl. Acad. Sci. USA 104 (2007) 19831-19836.。
も、構造的特徴であった。
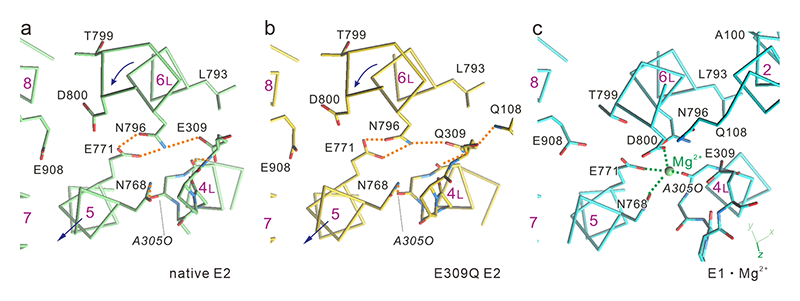
図4 Ca2+に配位するアミノ酸残基の配置とE2→E1遷移に伴う構造変化。(a) 天然型SERCA1aのE2状態。(b) Glu309Gln変異体のE2状態。(c) 天然型のE1・Mg2+状態。細胞質側からほぼ膜面に垂直に見ている。オレンジ色の点線は水素結合を表す。紫色の数字はヘリックスの番号。矢印はE2→E1遷移に伴うM5、M6ヘリックスの動き。M4ヘリックスは紙面に対し手前に(細胞質側に)~5 Å移動する。Ca2+に配位するアミノ酸残基の配置は天然型とGlu309Gln変異体とで同一であることに注意。

図5 M4ヘリックスの上下運動とM5の湾曲の関係。M4とM5ヘリックスはPドメインに上端で固定されている。M5ヘリックスは連続したヘリックスであるが、2つのGlyのために3つのセグメントから成るとみなすことができる。E1→E2遷移においてはM5が図のピンク色から緑色の位置に傾きを変える(湾曲する)。その結果、Pドメインは傾斜し、M4ヘリックスはΔd1だけ下がり、Ca2+の結合に適当であったアミノ酸残基の配位は破壊される。一方で、M4LはよりM5Lに接近する(押し付けられる)。2002年モデル[12][12] C. Toyoshima and H. Nomura: Nature 418 (2002) 605-611.の改訂版。
ところがこのGlu309Gln変異体では、細胞質側頭部は開いている(図2a)。特にAドメインは125°回転し、E1状態の方位角とほぼ一致している。しかし、回転軸が膜に対して傾いている結果、膜表面に~10 Åも接近していた。このAドメインの傾きは、これまでに得られたどの状態の構造とも違っていた。一方、M5ヘリックスはほぼ真っ直ぐであり、標準的E2状態のような大きな湾曲は見られなかった。M2はE1様の連続したヘリックスになっており、蛋白質分解酵素による切断パタン[11][11] G. Inesi, D. Lewis, C. Toyoshima, A. Hirata and L. de Meis: J. Biol. Chem. 283 (2008) 1189-1196.を良く説明するものであったが、大きく膜面に対して傾いていた。これはAドメインの大きな傾斜と相関していると考えられる。M1ヘリックスは、これもまた全くユニークな位置・傾斜であり、しかも、これまでE1・2Ca2+状態でしか見られなかった連続ヘリックス(他の状態ではAsp59のところで大きく折れ曲がり、細胞質側(M1')は両親媒性であることを反映し、膜表面に寝る)であった。一方、(iii)のM4ヘリックスは下がった位置にあり、Ca2+結合に関与するアミノ酸残基の配置は側鎖まで含めて全く同一であり(図4)、従って、プロトン化状態も同じはずである。つまり、このGlu309Gln変異体の構造は間違いなくE2状態にあるが、E2構造の「看板」と目されていた(i)と(ii)は本質ではないことが明らかになってしまったのである。一方、あまり注目されてこなかった、(iv)のM6のヘリックスがほどけている部分はE2構造を保っていた。
5. Glu残基のGln置換で何が起こり得るのか
このように大きな構造変化の原因はGlu309をGlnに置換したことである。一方で、より激しい置換であるはずのGlu309Ala変異体は、蛋白質分解酵素による切断パタンが示すように、標準的E2構造を保っていた。Gluの側鎖の位置には水一分子が同定された。つまり、水分子がプロトン化カルボキシル基の代わりをしていたわけである。一方、Gln変異体ではGlnの側鎖が邪魔をして水は入れない。ともかく、Glu→Glnの置換が大きな構造変化を引き起こしたわけであるから、まずは標準的E2状態においてGlu309が何をしているかを見てみよう。
Glu309はM4中央付近のヘリックスがほどけた部分に位置する(図2、6)。そのカルボキシル基の酸素原子の1つはM4ヘリックスの内腔側(M4L)の上端(細胞質側の端)にあるVal304主鎖カルボニル基の酸素と2.8 Åの距離にある(図6b)。この距離はともに負の荷電を有する酸素原子間の距離としては近過ぎ、一方は(この場合可能なのはGlu309側だけ)プロトン化して水素結合を作っている、としか考えられない。実際、分散を加味した密度汎函数法(DFT-D)を用いて、このX線結晶解析(2.5 Å分解能)による原子モデルの量子化学的な構造最適化を行ってみても、結晶構造からのずれはほぼない。一方、Glu309Gln変異体の結晶構造ではこのN-O間の距離は3.0 Åと僅かに長い。これが意味のある違いなのかは、この結晶構造をいくら眺めていても分からないので、そもそも「量子化学的に期待される水素結合の距離はどんなものか、それはCOO--H+-O間とNH2-O間で違うのか」を調べることにした。
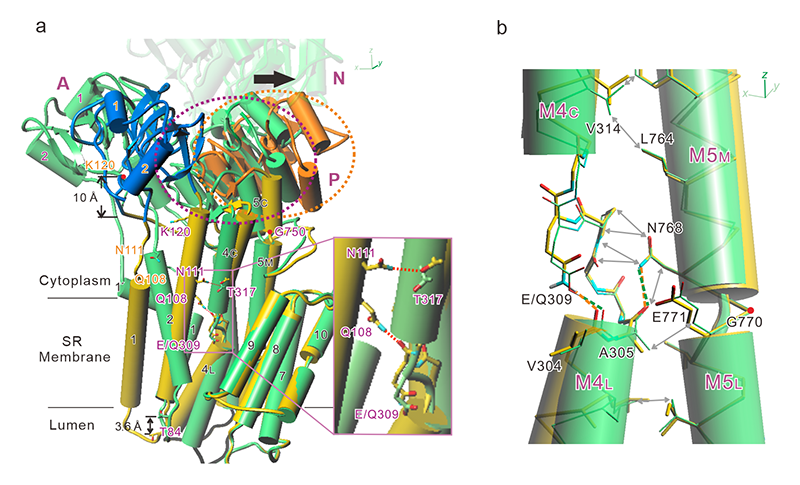
図6 SERCA1aの天然型とGlu309Gln変異体のE2状態の結晶構造の重ね合わせ。緑色、天然型。黄色(青色、Aドメイン。オレンジ色、Pドメイン)、変異体。変異体のAドメインは127°膜に垂直から14°傾いた軸の周りに回転しており、Lys120の位置で10 Å膜面に近づいている。そのため、Aドメインに直結したM2ヘリックスは、傾くだけでなく、部分的にほどけたヘリックスから連続したヘリックスになる(a)。M4Cの傾きの差は~5°。この差はM5Cの傾きの変化で吸収され、M5Mはほとんど傾きを変えない。Pドメインの運動は膜面にほぼ平行である(黒の矢印)。(b) 両頭の矢印はファンデルワールス接触を、破線は水素結合を示す。Glu309のGln置換の結果、Val304カルボニル基との間の水素結合は僅かに長くなり、M4C全体が5°傾くとともに上昇している。M4LはAla305のカルボニル酸素の周りに16°、M5Lから離れる方向に回転していることに注意。(a),(b)ともにM7-M10ヘリックスで重ね合わせてある。
この計算自体は小さい系で可能なので、CCSD(T)(coupled-cluster single-double and perturbative triple)を用いた精度の高い計算を行った[1][1] N. Tsunekawa, H. Ogawa, J. Tsueda, T. Akiba and C. Toyoshima: Proc. Natl. Acad. Sci. USA 115 (2018) 12722-12727.。標準的E2構造におけるGlu309-Val304のgeometryを仮定した時、プロトン化カルボキシル−カルボニル間の水素結合の距離(O-O間のエネルギー的な至適値)は、水素結合の教科書的標準距離である2.83 Åだが、アミド−カルボニル間(N-O間の距離)は3.16 Åであった(図2b)。M3-M6の4つのヘリックスの58残基を含むDFT-Dによるエネルギー最小化の結果は、CCSD(T)の結果と僅かに異なり、Glu→Gln変異によって2.62 Åが2.98 Åに広がるはずとの答えであった。
実際の結晶構造と量子化学的に最適化されたモデルは実に良く一致しており、結晶構造では確かに水素結合距離は2.8 Åから3.0 Åに広がっていた(図6b)。このOがNに置換されることによる僅かな差は、Gln側鎖の回転で十分許容できそうであるが、van der Waals接触を調べてみると、Glu309の側鎖は隣接するM6ヘリックスにあるLeu793、Asn796を中心にしっかり固定されており、角度変化の余地はないことが分かる。実際、Gln309から主鎖も上昇し、M4Cヘリックス全体は標準的E2状態のM4Cから~5°傾いていた。この傾きの変化自体は小さいがM4Cヘリックス(~30 Åある)の反対端(上端)では4.4 Å、Pドメインの上端ではそれがさらに拡大されて9 Åを超える運動を生み出していた(図6a)。この運動は、大きさは違うにせよ、E2→E1遷移で起こる運動と良く似ている。M4CヘリックスはPドメインの一部として組み込まれているから、これだけの大きさの運動があると、N-A間の接触面は保たれるわけもなく、細胞質側頭部は開いてE1様になったし、Aドメインは大きく回転してやはりE1様の位置をとることになったわけである。そうなると、Aドメインに直結しているM2の位置も変わり、Aドメインの傾きのためにE1様ではあるがユニークな位置をとり、膜面に近くなったためにE1様の連続なヘリックスとなったということである。M1に関しても同様であり、連続ヘリックスとなることに積極的意味はあるまい。一方、これだけ大きい構造変化があってもCa2+に配位するアミノ酸残基の配置は少しも揺らがなかった(図4)。しかも、M4Cは持ち上がっているのに、M4Lは下がったままである。何故か。
6. Ca2+ポンプのE2構造の本質
そもそもM4の高さを決めているものは直接的にはPドメインの傾きであり、それを決めているのはM5ヘリックスの湾曲具合のはずであった[9,12][9] C. Toyoshima: Biochim. Biophys. Acta 1793 (2009) 941-946.
[12] C. Toyoshima and H. Nomura: Nature 418 (2002) 605-611.(図5)。E2状態ではM5ヘリックスはM1方向に大きく湾曲し、PドメインのAドメインに近い側を膜面に近づけるからM4も下がり、高親和性Ca2+結合サイトを破壊する。一方、Pドメインが傾く結果、AドメインはPドメインに押され、E1状態とは110°違う方向を向くように回転し、その結果Nドメインと3つの塩橋を含む7つの水素結合を作れるようになって閉じたコンパクトな細胞質側頭部を形成する。これが2002年の理解であった[12][12] C. Toyoshima and H. Nomura: Nature 418 (2002) 605-611.。この時点ではM5の湾曲にもっと深い意味があることを理解していなかった。
その1つはM6のヘリックスがほどけている部分の回転との連関である。このヘリックスがほどけている部分にはThr799とAsp800の2つの残基があり、E2状態においてはThr799のE1の位置にAsp800が来ることになり、Thr799はサイトIから完全に除外される(図3)。そしてサイトIICa2+を配位していたAsn796がプロトン化したGlu771と2つの水素結合を形成し、この構造を安定化する。Glu771はサイトICa2+の配位に重要な残基だから、Thr799とGlu771をCa2+の配位から除外することによってサイトIを二重に破壊したわけである。実はこの、M6のほどけた部分の回転をもたらしているものは、M5の湾曲なのである。Tyr763とLeu764と2つの嵩高い残基がM5の湾曲によってThr799とAsp800の側鎖を押して回転させているのである。一方で、Glu771がプロトン化していない限り、Glu771-Asn796間の水素結合は作れず、この構造は安定化しない。つまりM5の湾曲はGlu771のプロトン化を引き起こす(確認する)ためのメカニズムなのであろう。
もう1つの意味は、この湾曲がM4LをM5、M6にきつく押さえつけているということである。図5に示すようにM5の湾曲(傾斜)はGly770を中心にして起こるため、M5のGly770より下側(M5L)は動かず、E2状態への遷移によってM4は下に行くほどM5、M6との距離は短くなる。従って、Glu309→Glnの置換によってVal304との水素結合は長くなるが、その差をM4Lの傾きの変化で吸収するのは不可能であるし、M5側に移動して吸収することもできない。実際、変異体でM4Lの傾きの変化は起こっている。しかし、その方向は予想とは逆で、Val305はGlu/Gln309により接近するようになっている(図6b)。すなわち、Ala305カルボニル酸素を中心とするM4Lの回転が生じている。このカルボニル酸素はAsn768アミドとの間で水素結合を作っており、その位置はGlu771 CβやAsn768 Cβ等との接触で決まっている。Glu771がAsn796との二重の水素結合でしっかり固定されているため、Asn768も動けない。そのため、M4Lは上昇できず、結局M4Cがその傾斜角を変えることになる。つまり、M4ヘリックスの上昇のためには、M5、M6ヘリックスから離れることが必要なのである。実際、M6ヘリックスのほどけている部分がE1様になれば、Asp800とThr805がM4CにあるVal314を押すことになって、M4CをM5、M6ヘリックスから離し、Ala305もAsn768から離れ、ロックが外れることになる。
一方で、Glu309Gln変異体の構造は、このロック機構には十分な裕度があることを示している。実際、細胞質側頭部は開いているし、M5はかなり真っ直ぐである。それでもこのロックは外れない。M5には2つのGly(750と770)があり、M5ヘリックスはある程度独立した3つのセグメントから成るとも言える(図5)。M4Cの傾きの~5°の違いはM5の上端セグメント(M5C)の傾きの変化で十分吸収できる。但し、M5CはPドメインに組み込まれているから、M5Cの傾きの変化はPドメインの運動を引き起こす(図6aの黒矢印)。この運動は、閉じた細胞質側頭部を開くのには十分な大きさであるが、M5Mの大きな変化を引き起こすほどではない。実際、Glu309Gln置換によるM5Mの傾きの変化は~4°であり、E1・Mg2+に遷移する時には13°である。そこまでいかないとM4は上昇できず、E1には遷移できない。Glu771とAsn796間の二重の水素結合は局所的にM5-M6ヘリックス間を広げ、M5Lを内腔側に押し下げている。Glu771が自由にならない限り、M5Mの大きい変化は起こり得ないのである。すなわち、Glu309Gln変異体の構造はE2構造のロック機構を見事に示してくれたわけである。
7. E2→E1遷移
それでは、この変異体構造は正常のポンプ反応サイクルにおける構造変化とどう関係しているのだろうか。Glu309のカルボキシル基はE2状態ではプロトン化されていると考えられてきた(図3)。上述のように、結晶構造を出発点とし、DFT-Dを用いた量子化学計算によってエネルギー最小化を行ってみると、プロトン化しているならば大きな構造変化は起こらない。問題はプロトンが外れた時に何が起こるか、である。これもDFT計算によってエネルギー最小化を図ってみる。Glu309カルボキシル基の酸素原子とVal305カルボニル基の酸素原子間には今や水素結合は形成され得ないので静電的反発が起こる。それによって、M4CとM4L間は拡大する(図7b)。これは、Glu309Gln変異体の構造で見たように、E2状態の閉じてコンパクトな細胞質側頭部を開き、M5の湾曲に対する制限を外すことになる。肝心なことは、それと同時に、Glu309カルボキシル基の負電荷を中和すべく、M6のAsn796のアミド基がGlu771のカルボキシル基との水素結合を切って、Glu309側に移動することである。その結果、M6は自由になり、ヘリックスがほどけていた部分はM5の湾曲によって回転させられていたのと逆方向へ回転する。これはM5の湾曲を戻し、M4CをM5、M6から離れる方向へ押すことになる。M5の湾曲が戻る結果、M4Lを止めていたAsn768はM4から離れ、Ala305との間の水素結合も切れる結果、M4は自由になる。つまり、M4全体が上昇することが許されることになり、E1状態へ遷移する。これがE2・nH+→E1遷移のシナリオであろう。この遷移によって、Glu771、Asp800ともにプロトンを放出しているはずである。従って、金属イオンに対し高親和性になり、Ca2+が結合する前にはCa2+の1万倍は細胞質中に多く存在するK+や100倍は多いMg2+を結合して構造を安定化するのである[4][4] C. Toyoshima, S. Iwasawa, H. Ogawa, A. Hirata, J. Tsueda et al.: Nature 495 (2013) 260-264.。
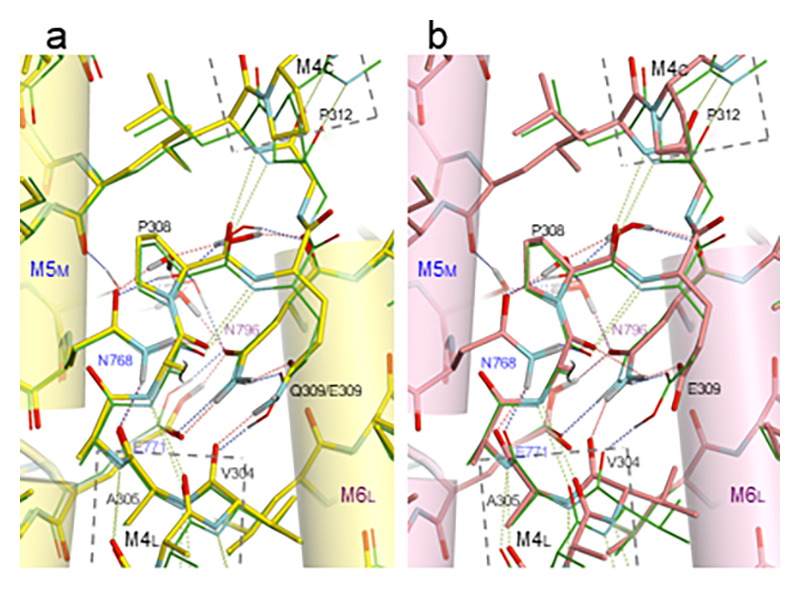
図7 SERCA1aのE2状態におけるGlu/Gln309周辺の水素結合ネットワークの詳細。(a) 天然SERCA1aの原子モデル(緑色の細い線)とGlu309Gln変異体の原子モデル(黄色の棒と円筒)を重ねたもの。(b) 天然SERCA1aにおいてGlu309がプロトン化されている時(緑色の細い線)と脱プロトン化された時(ピンク色の棒と円筒)。点線は水素結合を表す。原子モデルは全て量子化学的に最適化されており(但し、各残基のCα原子は空間固定)、膜面にほぼ平行に、図2の左側から見ている。M5とM6ヘリックス上の残基が最大限一致するように重ね合わせた。
8. charge movement測定実験
この結果はcharge movementの実験結果[14][14] Y. Liu, R. Pilankatta, D. Lewis, G. Inesi, F. T.-Buoninsegni et al.: J. Mol. Biol. 391 (2009) 858-871.を非常に良く説明するものであった。E2・nH+→E1がプロトンの放出を伴うことから予想されるように、pHを弱酸性から弱アルカリ性に上げるとE2·nH+→E1遷移が起こる。そこで放出されたプロトンによる一過性の電流を測定すればこの過程をモニターできる。このプロトン放出は協働的で、天然のSERCA1aならpH6.4付近で明瞭な遷移が観測される。一方、Glu309Gln変異体では遷移が観測されるのはpH7.8付近である。このことはGlu309以外のGlu/Aspからのプロトン放出はpH7.0付近では起こらないことを意味する。一方、天然のSERCA1aではプロトンの放出は一段で協働的に起こることから考えて、Glu309がまずプロトンを放出し、それに伴う構造変化によって他のGlu/Aspからの放出が起こるのだろう。実際Glu/Gln309-Val304間の水素結合はGln309の方が安定なはずで(蛋白質分解酵素による切断パタンはE1様であるが)、高いpHまでE2状態を保つだろうと考えられる。
それではGlu309Ala変異体ではどうかというと、この変異体の結晶構造においてはGlu309のカルボキシル基は水分子で置換されているのであった。であるならば、プロトンを放出したGlu309のカルボキシル基の負電荷をAsn796アミド基が中和する必要はなくなるので、Glu309のプロトン放出が引き起こす構造変化はやはりM6、M5には伝わらないと考えられる。だから、charge movementに関してはどちらの変異体も同じ挙動を示すはずである。実験結果[14][14] Y. Liu, R. Pilankatta, D. Lewis, G. Inesi, F. T.-Buoninsegni et al.: J. Mol. Biol. 391 (2009) 858-871.はまさにその通りであった。一方、天然型ではGlu309の側鎖が邪魔をして、水分子は入れないのである。この結果は2009年に発表されたが、現象の記述に終わっていた。構造を理解して初めてその意味が理解されたわけである。
9. Ca2+ポンプの反応サイクルは複雑である
さて、ここまでは、ポンプ機構の理解が最も容易と考えられる単一の経路(図1の最も外側の経路)を仮定して進めてきたが、ポンプサイクルはポンプ蛋白質の自己燐酸化と共役しており、そのためには基質であるATPとCa2+との複合体が形成されれば良い。その意味では、最も重要な中間体はE1・ATP・2Ca2+(結合したCa2+を膜内に閉じ込める直前の)状態であり(図1)、それに至る経路は複数あり得る。実際、SERCAはE2状態でもATPに対する高い親和性(10 μM以下)を持ち、細胞中にはmMのATPが常に存在するから、Ca2+結合後にATPが結合するというE2→E1→E1・2Ca2+→E1・ATP・2Ca2+という経路を通るよりも、E2→E2・ATP→E1・2Ca2+・ATP或いはE2→E1→E1・ATP→E1・ATP・2Ca2+という経路をたどる可能性の方が高いであろう。また、生理的pHではE2よりもE1(Ca2+に対し高親和性)状態にあると考えられるが、どちらの経路をたどるかはE2→E1遷移とE2に対するATPの結合速度に依存するであろう。いずれにせよ、そのような異なった経路上の中間体の構造を決定すれば、構造の意味(どうしてポンプ蛋白質の構造はそうでなければならないのか)をより深く理解できるはずである。手始めはE2・ATP状態である。燐酸化反応は起こらないはずだからATP結合そのものがどのような構造変化を引き起こすのかを理解できる、と期待された。実際、このE2・ATP構造[15][15] Y. Kabashima, H. Ogawa, R. Nakajima and C. Toyoshima: Proc. Natl. Acad. Sci. USA 117 (2020) 18448-18458.は予想外のbreak throughであったので、解説を書こうと思ったのだが、与えられた字数を既に大幅に超過している。稿を改めたい。
10.おわりに
20年以上も同じ蛋白質の構造を眺めていると、「構造を理解するということはとんでもなく時間のかかるもの」であり、「如何に自分の理解は浅かったか」を痛感することになる。「SERCAは何のためにプロトンを対向輸送するのか」に対する答えは随分変わった。「せっかくエネルギーを消費してプロトンを運んでも、小胞体膜はプロトンを含む1価のイオンに対し透過性であるから、プロトンは元に戻る。どうして、そんな無駄なことをするのだろう」が疑問だった。最初は、「Ca2+結合サイトの構造安定性のために無駄だけれど運ぶのだ」が答えであった(2005年)[10][10] K. Obara, N. Miyashita, C. Xu, I. Toyoshima, Y. Sugita et al.: Proc. Natl. Acad. Sci. USA 102 (2005) 14489-14496.。比較的すぐにそれは浅はかで「Ca2+に対する親和性を下げ、また高親和性に戻すためである。小胞体膜がプロトンに対しleakyなのは、Ca2+輸送を維持するためである。無駄なのに運ぶわけではない。その逆で、運んだままにしておくと駄目になるから元に戻すのだ」ということに気が付いた[9][9] C. Toyoshima: Biochim. Biophys. Acta 1793 (2009) 941-946.。今回は「いんや、それではまだ足りんよ」と宣告されたわけである。
ここに紹介したように、Ca2+ポンプの構造はM4Cの傾きに非常にsensitiveである。Ca2+ポンプは実に、酸素原子と窒素原子の差を認識できるのである!ヘリックスは剛体であろうから、その一端における小さな運動(傾きの変化)は他端では大きな構造変化と成り得る。これは、蛋白質が構造変化を伝える常套手段であるが、この構造解析から、ヘリックスの傾きを変える手段として2つあることを学んだ。すなわち、M4CとM4Lの2つの部分を継ぐ水素結合の長さを長くすることであり、もう1つは電荷を導入しそれによって生じる反発(或いは引力)を利用することである。前者はまさにGlu309Glu変異体がやっていたことである。反応サイクル中で変異を起こすことは不可能であるから、蛋白質ができることは後者でしかあり得ない。一方、電荷の導入による効果は周囲にあまねく及ぶわけだから、複数のeventsを引き起こし得る。つまりこのGlu309Gln変異体はそのうちの1つだけ(水素結合の長さを長くすること)を選択的に取り出して、何が起こるかを見せてくれたことになっていたのである。そんなことは実験をやる前から予想できるわけがない(少なくとも筆者には)。
だから「実験はやってみるものだ」し、そして「答えはいつも思いがけない方向から飛んでくる」のである[16][16] C. Toyoshima: Phys. Scripta 91 (2016) 042501.。「1つのことが分かるとがらがらと色々なことが解決してしまう」というのも「蛋白質は1つのeventで幾つものことを同時に片付ける」というのもこの論文[1][1] N. Tsunekawa, H. Ogawa, J. Tsueda, T. Akiba and C. Toyoshima: Proc. Natl. Acad. Sci. USA 115 (2018) 12722-12727.を書いていてまたまた痛感したことであった。「蛋白質は実に良くできている」のである。
謝辞
本研究はSPring-8の長期利用課題(2016A0133と2018A0144)の一部としてなされたものである。量子化学計算は研究室の恒川直樹博士が担当した。回折データ収集に当たってはSPring-8 BL41XUの担当者、特に長谷川和也博士の絶大なご支援をいただいた。ここに記して御礼申し上げたい。また、結晶を作製してくれた杖田淳子さん、データ収集を手伝ってくれた研究室のメンバーに感謝したい。
参考文献
[1] N. Tsunekawa, H. Ogawa, J. Tsueda, T. Akiba and C. Toyoshima: Proc. Natl. Acad. Sci. USA 115 (2018) 12722-12727.
[2] C. Toyoshima, M. Nakasako, H. Nomura and H. Ogawa: Nature 405 (2000) 647-655.
[3] Y. Norimatsu, K. Hasegawa, N. Shimizu and C. Toyoshima: Nature 545 (2017) 193-198.
[4] C. Toyoshima, S. Iwasawa, H. Ogawa, A. Hirata, J. Tsueda et al.: Nature 495 (2013) 260-264.
[5] M. M. G. Geurts, J. D. Clausen, B. Arnou, C. Montigny, G. Lenoir et al.: Proc. Natl. Acad. Sci. USA 117 (2020) 31114-31122.
[6] A. Marchand, A.-M. L. Winther, P. J. Holm, C. Olesen, C. Montigny et al.: J. Biol. Chem. 283 (2008) 14867-14882.
[7] J. D. Clausen, M. Bublitz, B. Arnou, E. Montigny, C. Jaxel et al.: EMBO J. 32 (2013) 3231-3243.
[8] S. Zafar, A. Hussain, Y. Liu, S. Lewis and G. Inesi: Arch. Biochem. Biophys. 476 (2008) 87-94.
[9] C. Toyoshima: Biochim. Biophys. Acta 1793 (2009) 941-946.
[10] K. Obara, N. Miyashita, C. Xu, I. Toyoshima, Y. Sugita et al.: Proc. Natl. Acad. Sci. USA 102 (2005) 14489-14496.
[11] G. Inesi, D. Lewis, C. Toyoshima, A. Hirata and L. de Meis: J. Biol. Chem. 283 (2008) 1189-1196.
[12] C. Toyoshima and H. Nomura: Nature 418 (2002) 605-611.
[13] C. Toyoshima, Y. Norimatsu, S. Iwasawa, T. Tsuda and H. Ogawa: Proc. Natl. Acad. Sci. USA 104 (2007) 19831-19836.
[14] Y. Liu, R. Pilankatta, D. Lewis, G. Inesi, F. T.-Buoninsegni et al.: J. Mol. Biol. 391 (2009) 858-871.
[15] Y. Kabashima, H. Ogawa, R. Nakajima and C. Toyoshima: Proc. Natl. Acad. Sci. USA 117 (2020) 18448-18458.
[16] C. Toyoshima: Phys. Scripta 91 (2016) 042501.
東京大学 定量生命科学研究所
〒113-0032 東京都文京区弥生1-1-1
TEL : 03-5841-8492
e-mail : ct@iqb.u-tokyo.ac.jp








