Volume 23, No.4 Pages 321 - 325
1. 最近の研究から/FROM LATEST RESEARCH
(SPRUC 2018 Young Scientist Award受賞 研究報告)
微小結晶タンパク質X線結晶構造解析を加速する自動データ処理システムの開発
Facilitating Structure Analyses from Protein Microcrystals by Automated Data Processing
東京大学 大学院理学系研究科 Graduate School of Science, The University of Tokyo
- Abstract
- X線結晶構造解析によるタンパク質立体構造決定では、十分な大きさと回折能を持ったタンパク質結晶を得る過程がボトルネックになっている。特に、膜タンパク質や不安定な複合体などは難度が高く、数から数十マイクロメートルオーダーの小さな結晶しか得られないことが多い。そのような微小結晶に対しては、マイクロビームX線を利用することで高S/N比でのデータ収集が可能になるが、放射線損傷の問題から、1つの結晶から高分解能かつ完全なデータを得ることは難しい。よって、構造解析には複数の微小結晶を用いる必要がある。筆者らは、SPring-8のマイクロビームビームラインBL32XUにおいて多数の微小結晶からデータ収集・処理を行うワークフローを整備し、自動化に成功した。これらの開発によって、微小結晶を用いた構造解析が劇的に容易になり、多くの成果が創出された。
1. はじめに
タンパク質や核酸などの生体高分子の多くは、固有の立体構造を形成することで、それぞれ固有の機能を発揮する。X線結晶構造解析は、複雑な生体高分子の立体構造を数オングストロームの高い分解能で決定できる強力な手法であり、生命現象の理解に大きく貢献してきた。通常、50から200 µm程度のサイズの結晶が1つあれば、一般的なタンパク質結晶用の放射光ビームラインで簡便に回折強度データ収集が可能である。しかしながら、膜タンパク質や不安定な複合体などは試料調製の難度が高く、十分なサイズの結晶を得ることが極めて困難である。特に、近年膜タンパク質の結晶化に非常によく利用される脂質メソフェーズ法[1][1] M. Caffrey: Acta Crystallographica Section F 71 (2015) 3-18.(lipidic mesophase method)では、品質は高いものの大きな結晶に育たないということがよく起こる。微小結晶は体積が小さいため回折強度が弱く、さらに試料周囲の物質による散乱バックグラウンドも相対的に高くなるため、微結晶に選択的にX線を照射できるマイクロビームの利用が必須である。マイクロビームが利用できるようになって初めて、微小結晶からのデータ収集は現実的になった。しかしながら、体積の小さな微小結晶から高分解能の回折強度を得ようとして高強度のX線を用いると、簡単に吸収線量の限界(~10 MGy)を超え、得られる分解能が劣化してしまう。すなわち、微小結晶1つから高分解能かつ完全な回折データを得ることは困難である。解決方法として、1つは近年利用可能になったX線自由電子レーザー(X-ray free-electron laser: XFEL)が挙げられる。大強度のフェムト秒パルスX線であるXFELを用いると放射線損傷の発生よりも前に回折データを収集できる(diffraction before destruction[2][2] R. Neutze, R. Wouts, D. van der Spoel et al.: Nature 406 (2000) 752-757.)ため、この原理の適用範囲内においては損傷を気にすることなく大強度X線による高分解能データ収集が可能になる。ただし、XFEL照射直後に試料は損壊するため、結晶1つからは静止写真が1枚しか得られず、構造解析には多くの場合、数千枚以上の回折パターンが必要になる。さらに、ビームタイムも非常に限られているため、静的な結晶構造を得るだけの目的にはやや利用しづらいのが現状である。一方、従来の光源では放射線損傷を完全に回避することはできないが、露光中の試料の回転によって多数の回折点の積分反射強度を測定できるので、より少数の結晶からでも高精度な回折強度を得ることができる。具体的には、クライオ条件下で1結晶あたり5–10°程度の狭い振動範囲(small-wedge)だけ測定することで、放射線損傷が重篤にならない範囲で高分解能の回折データが得られる。この場合、通常は数十から数百個程度の結晶だけで十分な質のデータが得られる。結晶にビームを当てる際、クライオループなどの試料ホルダに複数の結晶がランダムな方位で収められていれば、同一の振動範囲で各結晶からデータ収集を行えるため、正面から見た結晶の位置を決めるだけでよいという利点もある。すなわち、このスキームでは、まずループ全域から結晶位置を検出し、各位置から振動写真データを収集することを繰り返す(図1)。SPring-8のマイクロビームビームラインBL32XUにおいても、このスキームの確立および自動化が2015年頃から進められ、ZOO[3][3] K. Hirata, K. Yamashita, G. Ueno et al.: submitted (2018).システムとして現在活発に利用されている。同時期に仏ESRFからMeshAndCollect[4][4] U. Zander, G. Bourenkov, A. N. Popov et al.: Acta Crystallographica Section D 71 (2015) 2328-2343.が発表されたほか、世界的にも開発が進められている。筆者はZOOのうち、特に回折スキャンによる結晶位置検出と、回折強度データの自動処理に携わったので、本稿ではその2つの事柄について記述し、いくつか実例を紹介する。
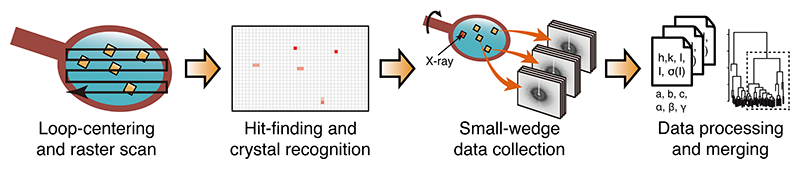
図1 多数の微小結晶を用いたsmall-wedgeデータ収集スキーム。X線を用いた回折スキャン(収集する角度領域は数度程度なので図の方位のみ)によって結晶位置を特定後、個別にデータを収集、マージ処理を行う。総説[5][5] M. Yamamoto, K. Hirata, K. Yamashita et al.: IUCrJ 4 (2017) 529-539.より転載(CC-BY 2.0: https://creativecommons.org/licenses/by/2.0/)。
2. 回折スキャンによる結晶センタリング
微小結晶に確実にX線を照射し回折データを収集するには、高い精度で結晶位置を決める必要がある。加えて、特に脂質メソフェーズ法による結晶は顕微鏡下で視認できないことが多いため、X線によって試料ホルダを2次元走査し、回折斑点の有無から結晶位置を決める方法が取られる。このとき損傷を与えない程度の低線量に抑える必要があるが、結晶位置判定には強度の大きい低分解能領域の回折斑点のみを用いるため、データ収集に値するような結晶を見出すにはそれで十分である。スキャン領域は試料ホルダの大きさに依存するが、多いときは1万箇所(たとえば10 × 10 µm2のビームサイズで1 × 1 mm2を走査)以上をスキャンして結晶位置を探索する。高速に生み出される大量のイメージを即座に解析して結晶位置を提示するため、筆者はプログラムSHIKA(Spot-wo Hirotte Ichiwo Kimeru Application)を開発した。SHIKAは並列計算によってイメージを高速処理し、GUI上に結果を表示する(図2)。BL32XUでは検出器EIGERのstreaming modeを活用し、並列計算によってリアルタイムのイメージ処理を行っている。SHIKAでは、もともとSFX実験のために開発されたプログラムCheetah[6][6] A. Barty, R. A. Kirian, F. R. N. C. Maia et al.: Journal of Applied Crystallography 47 (2014) 1118-1131.のピークサーチ機能を移植して利用している。ピークサーチ領域は分解能5 Å以下の低角領域のみを対象としており、氷由来のピークが混入しないようにして偽陽性率を抑えている。画像処理時はピークサーチに加えて、GUIやレポートHTMLで表示するためのサムネイル画像の作成も行う。レポートHTMLファイルも生成され、ユーザーは持ち帰ったデータからもWebブラウザを利用して結果を確認できる。SHIKA GUIで選択した結晶の座標は、損傷を考慮して露光条件を設定するプログラムKUMA(Kessho-wo Ugokashitari Mawashitari-suru Application)に転送できるようになっている。

図2 SHIKA GUI。スポット数のヒートマップから選択した座標の実際の回折画像が確認できる。KUMAとはネットワーク経由で通信を行う。
結果の評価は、スポットの平均S/N比やスポットの数などの評価基準があるが、現在のところスポットの数を最も有用な指標として利用している。GUIから評価基準を選択し、スポット数のしきい値・結晶間の最小距離を入力することで、自動的に結晶の可能性の高い座標を選択できる。選択した複数の座標は一度にKUMAに転送し、small-wedgeデータ収集のスケジュールを即座に構築可能である。
3. Small-wedgeデータの自動処理
多数の微小結晶を構造解析に用いる場合、データ処理はいくらか複雑になる。データセット1つあたりがカバーする逆空間領域が狭いため、回折強度の正しい対称性を決めるのも単純ではない。また、強度の対称性よりも格子の対称性が高い場合(たとえばP4など)、反射の指数の付け方に幾何学的に等価な複数の可能性が生じる(indexing ambiguity)が、結合(マージ)する前に指数の付け方を合わせる必要がある。データをマージする際は、同型な、つまり同一の格子定数・構造に由来するデータのみを結合する必要がある。これらの要素技術の多くはすでに様々開発が行われているものの、微小結晶から収集したデータを構造解析に利用できる状態へ自動的に処理する仕組みは我々の知る限り存在していなかった。自動データ処理は、ハイスループット構造解析には非常に重要であり、またビームライン実験へのリアルタイムなフィードバックにも有用なものとなる。そこで筆者らはプログラムKAMO(Katappashikara Atsumeta data-wo Manual yorimoiikanjide Okaeshisuru)を開発し、small-wedgeデータ処理の自動化を実現した[7][7] K. Yamashita, K. Hirata and M. Yamamoto: Acta Crystallographica Section D 74 (2018) 441-449.。KAMOはビームライン外でも自由に利用できるよう、GitHubにてNew BSD Licenseで公開されている(https://github.com/keitaroyam/yamtbx)。KAMOは、XDS[8][8] W. Kabsch: Acta Crystallographica Section D 66 (2010) 125-132.や、DIALS[9][9] D. G. Waterman, G. Winter, J. M. Parkhurst et al.: CCP4 Newsletter on Protein Crystallography 49 (2013) 16-19.などのCCP4[10][10] M. D. Winn, C. C. Ballard, K. D. Cowtan et al.: Acta Crystallographica Section D 67 (2011) 235-242.のプログラムを用いて、自動処理を行う。また依存関係としてCCTBXライブラリ[11][11] R. W. Grosse-Kunstleve, N. K. Sauter, N. W. Moriarty et al.: Journal of Applied Crystallography 35 (2002) 126-136.を必要とする。KAMOを開発したことによって、大量の微小結晶を用いてより高分解能・高精度のデータが容易に得られるようになった。
KAMOには、個別データセットの自動処理機能に加え、同一格子に属するデータセットの選択、格子定数または強度相関に基づくデータセットのクラスタリング、異常値を示す結晶・回折像の除去とマージ、レポート作成の機能が備わっている。KAMOを用いた処理の流れを図3に示した。KAMOはもともとSPring-8ビームライン、特にZOOの一部として利用するために開発したが、現在ではその他の施設でも利用可能である。SPring-8ビームラインで利用する場合は、タンパク質結晶ビームライン標準のデータ収集プログラムであるBSS[12][12] G. Ueno, H. Kanda, T. Kumasaka et al.: Journal of Synchrotron Radiation 12 (2005) 380-384.のログを監視することでデータ収集と並行した自動処理ができるようになっている。
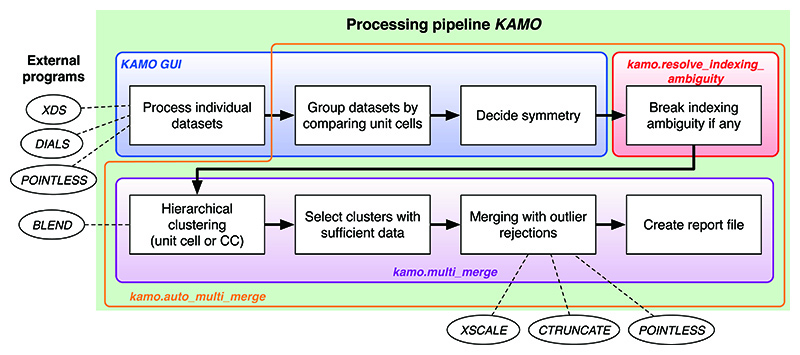
図3 KAMOプログラム利用の流れ。
論文[7][7] K. Yamashita, K. Hirata and M. Yamamoto: Acta Crystallographica Section D 74 (2018) 441-449.より転載(CC-BY 2.0: https://creativecommons.org/licenses/by/2.0/)。
4. 利用成果
BL32XUにおいて、SHIKAおよびKAMOはすでに多数の構造解析に利用されている。KAMOのドキュメント(https://github.com/keitaroyam/yamtbx/blob/master/doc/kamo-ja.md)にはKAMOを構造解析に用いた文献の一覧を載せている。このうち多くは生データも公開されており、https://github.com/keitaroyam/yamtbx/wikiには生データのURLとデータ処理方法を掲載している。興味を持った方はぜひ参照されたい。代表的には、多角体タンパク質[13][13] S. Abe, H. Tabe, H. Ijiri et al.: ACS Nano 11 (2017) 2410-2419.、リゾホスファチジン酸受容体LPA6[14][14] R. Taniguchi, A. Inoue, M. Sayama et al.: Nature 548 (2017) 356-360.、エンドセリンB型受容体のボセンタン結合型構造[15][15] W. Shihoya, T. Nishizawa, K. Yamashita et al.: Nature Structural & Molecular Biology 24 (2017) 758-764.、トリオースリン酸/リン酸輸送体(TPT)[16][16] Y. Lee, T. Nishizawa, M. Takemoto et al.: Nature Plants 3 (2017) 825-832.、真核生物由来MATEトランスポーター[17][17] H. Miyauchi, S. Moriyama, T. Kusakizako et al.: Nature Communications 8 (2017) 1633.、ヒト由来オレキシン2受容体[18][18] R. Suno, K. T. Kimura, T. Nakane et al.: Structure 26 (2017) 7-19.、ロイコトリエンB4受容体BLT1[19][19] T. Hori, T. Okuno, K. Hirata et al.: Nature Chemical Biology 14 (2018) 262-269.、鉄還元酵素Dcytb[20][20] M. Ganasen, H. Togashi, H. Takeda et al.: Communications Biology 1 (2018) 120.、アニオンチャネルロドプシンiC++[21][21] H. E. Kato, Y. S. Kim, J. M. Paggi et al.: Nature 561 (2018) 349-354.およびGtACR1[22][22] Y. S. Kim, H. E. Kato, K. Yamashita et al.: Nature 561 (2018) 343-348.が挙げられる。いずれも1結晶あたり5–10°程度のデータを数十から数百個の結晶から収集し、構造解析に成功している。利用成果の多くは脂質メソフェーズ法によって結晶化された膜タンパク質だが、本稿で述べた手法の適用範囲は膜タンパク質に限らない。最初に挙げた多角体タンパク質は細胞内結晶化によって得られた可溶性タンパク質試料であるし、また、同じく可溶性タンパク質で、恐らく砕けた状態の針状結晶を含んだ1ループのみから構造決定に成功した例もある(unpublished data)。マイクロビームによって試料の質の良い部分からデータを得ることが可能になったが、今回開発した多数の試料からデータを集める方法と組み合わせることで、それが現実的なものになったと言えるだろう。微小結晶や針状結晶でお困りの方は、ぜひ利用を検討していただきたい。
5. おわりに
タンパク質結晶構造解析では、良質かつ大きな結晶を得ることが非常に大きなボトルネックになっている。マイクロビーム技術の登場によって微小結晶を用いた構造解析が技術的に可能になり、これまで大きく良質な結晶が得られず構造解析できなかったような試料も解析対象になった。SHIKA・KAMOを含む微小結晶のためのワークフローおよびソフトウェア環境整備と、その自動化システムZOOの開発によって、そのような微小結晶を用いた構造解析が非常に簡易・高速になった。Small-wedgeデータ収集では、なるべく多くのデータを集め、適切にマージを行うことで、より高分解能かつ高精度のデータが得られることが期待される。KAMOは大量のデータセットの処理を大幅に省力化し、これによって処理方法について心配することなく大量のデータ収集を行えるようになった。
KAMOはBL32XU以外のSPring-8ビームラインでも利用可能なほか、海外の放射光施設の一部でもすでに導入されている。今後も、機能の高度化を続けていきたい。特に、データ収集中にオンザフライでマージおよび構造解析を自動的に行う機能が求められている。これによって、ある試料についてデータ収集を継続する必要があるかどうかの判断が自動的に可能になり、全自動データ収集がさらに効率化されるだろう。
謝辞
本研究は、日本医療研究開発機構(AMED)「創薬等支援技術基盤プラットフォーム事業」および日本学術振興会(JSPS)科学研究費補助金研究活動スタート支援「多数の微小結晶を用いた結晶構造解析手法および自動化システムの開発」などの支援を受けて行われました。関係各所に深く感謝します。また本研究の遂行にあたり、SPring-8構造生物学ビームライン関係者の方々、特に理化学研究所放射光科学研究センターの山本雅貴部長・平田邦生専任技師には大変お世話になりました。さらに、SPring-8 BL32XUのユーザーの方々には実サンプルの提供やフィードバックを通じて多大なご協力をいただきました。この場を借りてお礼申し上げます。
参考文献
[1] M. Caffrey: Acta Crystallographica Section F 71 (2015) 3-18.
[2] R. Neutze, R. Wouts, D. van der Spoel et al.: Nature 406 (2000) 752-757.
[3] K. Hirata, K. Yamashita, G. Ueno et al.: submitted (2018).
[4] U. Zander, G. Bourenkov, A. N. Popov et al.: Acta Crystallographica Section D 71 (2015) 2328-2343.
[5] M. Yamamoto, K. Hirata, K. Yamashita et al.: IUCrJ 4 (2017) 529-539.
[6] A. Barty, R. A. Kirian, F. R. N. C. Maia et al.: Journal of Applied Crystallography 47 (2014) 1118-1131.
[7] K. Yamashita, K. Hirata and M. Yamamoto: Acta Crystallographica Section D 74 (2018) 441-449.
[8] W. Kabsch: Acta Crystallographica Section D 66 (2010) 125-132.
[9] D. G. Waterman, G. Winter, J. M. Parkhurst et al.: CCP4 Newsletter on Protein Crystallography 49 (2013) 16-19.
[10] M. D. Winn, C. C. Ballard, K. D. Cowtan et al.: Acta Crystallographica Section D 67 (2011) 235-242.
[11] R. W. Grosse-Kunstleve, N. K. Sauter, N. W. Moriarty et al.: Journal of Applied Crystallography 35 (2002) 126-136.
[12] G. Ueno, H. Kanda, T. Kumasaka et al.: Journal of Synchrotron Radiation 12 (2005) 380-384.
[13] S. Abe, H. Tabe, H. Ijiri et al.: ACS Nano 11 (2017) 2410-2419.
[14] R. Taniguchi, A. Inoue, M. Sayama et al.: Nature 548 (2017) 356-360.
[15] W. Shihoya, T. Nishizawa, K. Yamashita et al.: Nature Structural & Molecular Biology 24 (2017) 758-764.
[16] Y. Lee, T. Nishizawa, M. Takemoto et al.: Nature Plants 3 (2017) 825-832.
[17] H. Miyauchi, S. Moriyama, T. Kusakizako et al.: Nature Communications 8 (2017) 1633.
[18] R. Suno, K. T. Kimura, T. Nakane et al.: Structure 26 (2017) 7-19.
[19] T. Hori, T. Okuno, K. Hirata et al.: Nature Chemical Biology 14 (2018) 262-269.
[20] M. Ganasen, H. Togashi, H. Takeda et al.: Communications Biology 1 (2018) 120.
[21] H. E. Kato, Y. S. Kim, J. M. Paggi et al.: Nature 561 (2018) 349-354.
[22] Y. S. Kim, H. E. Kato, K. Yamashita et al.: Nature 561 (2018) 343-348.
東京大学 大学院理学系研究科
〒113-0033 東京都文京区本郷7-3-1
TEL : 03-5841-4391
e-mail : keitaro.yamashita@bs.s.u-tokyo.ac.jp








