Volume 22, No.1 Pages 26 - 29
1. 最近の研究から/FROM LATEST RESEARCH
(SPRUC 2014 Young Scientist Award受賞 研究報告)
CRISPR-Casヌクレアーゼの結晶構造
Crystal Structures of CRISPR-Cas Nucleases
東京大学大学院 理学系研究科 Graduate School of Science, The University of Tokyo
- Abstract
- 原核生物のもつCRISPR-Cas系は外来核酸に対する獲得免疫機構としてはたらく。CRISPR-Cas系にかかわるRNA依存性ヌクレアーゼであるCas9やCpf1はガイドRNAと相補的な二本鎖DNAを特異的に切断する。近年、この性質を応用したゲノム編集技術の登場により、様々な生物のゲノム情報(生命の設計図)を「書き換える」ことが可能になってきた。これまでに我々はCas9およびCpf1の結晶構造を決定し、そのRNA依存的なDNA切断機構を解明してきた。また、Cas9とCpf1の構造比較から、CRISPR-Casヌクレアーゼの作動機構の共通性および多様性が明らかになった。さらに、これらの構造情報は新規のゲノム改変ツールの開発にも大きく貢献してきた。
1. CRISPR-Cas系
細菌などの原核生物のもつCRISPR-Cas(clustered regularly interspaced short palindromic repeat-CRISPR-associated)系はウイルスなどの外来核酸からの防御を担う獲得免疫機構としてはたらく[1][1] R. Barrangou et al.: Science 315 (2007) 1709-1712.。CRISPR-Cas系は、(1)Adaptation、(2)Expression、(3)Interferenceの3つのステップからなり、crRNA(CRISPR RNA)と複数のCasタンパク質が関与する。Interferenceのステップにおいて、特定のCasタンパク質とcrRNAがCas-crRNA複合体を形成し、crRNAと相補的な外来核酸を認識・切断する。Cas-crRNA複合体の構造にもとづき、CRISPR-Cas系は2つのクラスに分類される[2,3][2] K. S. Makarova et al.: Nat Rev Microbiol 13 (2015) 722-736.
[3] H. Nishimasu and O. Nureki: Curr Opin Struct Biol 43 (2016) 68-78.。クラス1 CRISPR-Cas系には複数のCasタンパク質からなる複合体が関与する一方、クラス2 CRISPR-Cas系には単一のCasタンパク質からなる複合体が関与する。クラス2 CRISPR-Cas系はII型、V型、VI型に分類される。
II型CRISPR-Cas系においてはCas9タンパク質が2種類のガイドRNA(crRNAおよびtracrRNA(trans-activating crRNA))とCas9-crRNA-tracrRNA複合体を形成し標的二本鎖DNAを切断する[4][4] M. Jinek et al.: Science 337 (2012) 816-821.(図1A)。Cas9は2つのヌクレアーゼドメイン(RuvC/HNH)をもち、HNHドメインは標的DNAのうちガイドRNAと相補的なDNA鎖(相補鎖)を切断する一方、RuvCドメインはもう一方のDNA鎖(非相補鎖)を切断する。標的DNAの認識にはガイドRNAとの相補性に加え、PAM(protospacer adjacent motif)とよばれる特定の塩基配列が標的配列の近傍に存在する必要がある。異なる生物種に由来するCas9のアミノ酸配列は多様であり、認識するガイドRNAやPAMの配列が異なる。たとえば、Streptococcus pyogenes由来Cas9(SpCas9)[4][4] M. Jinek et al.: Science 337 (2012) 816-821.、Staphylococcus aureus由来Cas9(SaCas9)[5][5] F. A. Ran et al.: Nature 520 (2015) 186-191.、Francisella novicida由来Cas9(FnCas9)[6][6] H. Hirano et al.: Cell 164 (2016) 950-961.はそれぞれNGG(Nは任意の塩基)、NNGRRT(RはAまたはG)、NGGという配列をPAMとして認識する。ガイドRNAのガイド配列(20塩基)は変更可能であるため、Cas9-sgRNA複合体はPAMをもち、ガイド配列と相補的な標的二本鎖DNAを特異的に切断することができる。さらに、crRNAとtracrRNAを連結したsgRNA(single-guide RNA)も同様に機能する[4][4] M. Jinek et al.: Science 337 (2012) 816-821.。したがって、Cas9-sgRNAは効率的なゲノム編集ツールとして急速に普及してきた[7][7] L. Cong et al.: Science 339 (2013) 819-823.。さらに、DNA切断活性をもたないCas9変異体(dCas9)はRNA依存的にゲノムDNAの狙った位置に結合するため、この性質を利用した様々な新規技術も開発されている。
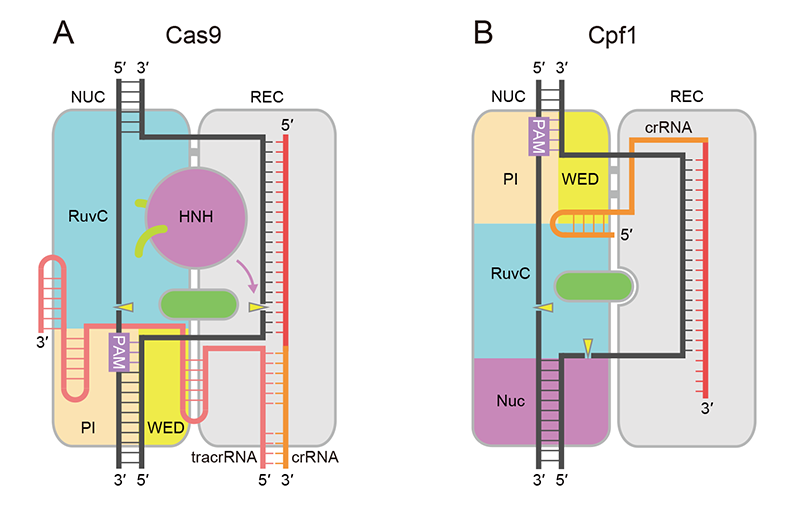
図1 Cas9とCpf1の作動機構
V型CRISPR-Cas系においてはCpf1(CRISPR from Prevotella and Francisella)がcrRNAと複合体を形成し標的二本鎖DNAを切断する[8][8] B. Zetsche et al.: Cell 163 (2015) 759-771.(図1B)。Cpf1は、(1)crRNAのみを利用しtracrRNAを必要としない、(2)TTTV(VはT以外の塩基)という配列をPAMとして認識する、(3)PAMから離れた位置で標的DNAを切断し突出末端をつくる、(4)RuvCドメインをもつがHNHドメインをもたない、などCas9と異なる特徴をもつ。哺乳類細胞において活性を示すAcidaminococcus sp.由来Cpf1(AsCpf1)やLachnospiraceae bacterium由来Cpf1(LbCpf1)は、Cas9と異なる特性をもつゲノム編集ツールとして注目されている。
2. SpCas9の結晶構造
2012年、Cas9は新規のRNA依存性ヌクレアーゼであることが報告されたが[4][4] M. Jinek et al.: Science 337 (2012) 816-821.、その作動機構は不明であった。そこで、Cas9によるDNA切断機構の解明を目指し、SpCas9-sgRNA-DNA複合体の結晶構造を決定した[9][9] H. Nishimasu et al.: Cell 156 (2014) 935-949.(図2A)。結晶構造から、SpCas9は2つのローブから構成されることが明らかになった。一方のローブはαヘリックスからなる新規フォールドをもち、sgRNA-DNAの認識に関与していた。そこでREC(recognition)ローブと命名した。もう一方のローブは2つのヌクレアーゼドメイン(RuvC/HNH)とC末端ドメインから構成されていた。そこでNUC(nuclease)ローブと命名した。C末端ドメインは新規フォールドをもちPAMと相互作用するのに適した位置に存在していた。構造情報をもとにした変異体解析の結果から、C末端ドメインはPAMの認識にかかわることが示唆された。そこでC末端ドメインをPI(PAM-interacting)ドメインと命名した。後日報告されたSpCas9-sgRNA-DNA(PAMを含む)複合体の結晶構造から、PIドメインのArg1333とArg1335がNGG PAMを認識していることが明らかになった[10][10] C. Anders, O. Niewoehner, A. Duerst and M. Jinek: Nature 513 (2014) 569-573.。RuvCドメインは非相補鎖DNAを切断するのに適した位置に存在していた一方、HNHドメインは相補鎖DNAと離れた位置に存在していた。したがって、DNA切断の際、HNHドメインは構造変化を起こすことが示唆された。sgRNAのガイド配列は相補鎖DNAとRNA-DNAヘテロ二本鎖を形成し、2つのローブの間に結合していた。一方、sgRNAの他の領域は特徴的な立体構造をとり、Cas9と広範囲に相互作用し複合体形成に貢献していた。以上の結果から、Cas9のRNA依存性DNA切断機構が説明された。

図2 Cas9とCpf1の結晶構造
(A) SpCas9-sgRNA-DNA複合体
(B) VQR改変体-sgRNA-DNA複合体
(C) SaCas9-sgRNA-DNA複合体
(D) FnCas9-sgRNA-DNA複合体
(E) AsCpf1-crRNA-DNA複合体
SpCas9-sgRNA-DNA複合体の結晶構造から、sgRNAの2つのループ領域は溶媒側に露出していることが明らかになった[9][9] H. Nishimasu et al.: Cell 156 (2014) 935-949.。この2つのループ領域にMS2ファージ由来コートタンパク質を特異的に認識するRNA配列を融合した改変型sgRNAを作製し、(1)dCas9、(2)改変型sgRNA、および、(3)転写活性化因子を融合したMS2コートタンパク質を哺乳類細胞に共発現させることにより、sgRNA依存的に標的遺伝子を活性化することに成功した[11][11] S. Konermann et al.: Nature 517 (2015) 583-588.。
3. SpCas9改変体の結晶構造
SpCas9-sgRNA-DNA複合体の構造情報は、DNA切断精度の高いSpCas9改変体[12,13][12] B. P. Kleinstiver et al.: Nature 529 (2016) 490-495.
[13] I. M. Slaymaker et al.: Science 351 (2016) 84-88.や異なるPAM配列を認識するSpCas9改変体[14][14] B. P. Kleinstiver et al.: Nature 523 (2015) 481-485.の開発にも貢献してきた。野生型SpCas9はNGG PAMを認識する一方、VQR改変体(D1135V/R1335Q/T1337R)、EQR改変体(D1135E/R1335Q/T1337R)、VRER改変体(D1135V/G1218R/R1335E/T1337R)はそれぞれNGA、NGAG、NGCGをPAMとして認識する[14][14] B. P. Kleinstiver et al.: Nature 523 (2015) 481-485.。SpCas9改変体によるPAM認識機構を解明するため、3種類のSpCas9改変体に関して、SpCas9-sgRNA-DNA複合体の結晶構造を決定した[15][15] S. Hirano, H. Nishimasu, R. Ishitani and O. Nureki: Mol Cell 61 (2016) 886-894.(図2B)。野生型SpCas9においてPAMの3文字目のGはArg1335によって認識される一方、VQR/EQR改変体においてPAMの3文字目のAはR1335Qによって認識されていた。VRER改変体においてはPAMの3文字目のCはR1335Eによって認識されていた。野生型SpCas9とSpCas9改変体の構造比較から、R1335Q/R1335E以外のアミノ酸置換は、PAM近傍の二本鎖DNAの糖−リン酸骨格の構造変化を誘起し、PAMの3文字目の塩基とR1335Q/R1335Eとの間の直接的な水素結合を可能にしていることが明らかになった。
4. SaCas9の結晶構造
SaCas9(1053残基)はSpCas9(1368残基)に比べて小型であるため、ウイルスベクターへの導入効率の高いゲノム編集ツールとして注目されている[5][5] F. A. Ran et al.: Nature 520 (2015) 186-191.。SaCas9の作動機構の解明を目指し、SaCas9-sgRNA-DNA複合体の結晶構造を決定した[16][16] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.(図2C)。SaCas9はSpCas9と同様に、2つのローブからなる構造をもち、RNA-DNAヘテロ二本鎖は2つのローブの間に結合していた。したがって、RNA依存的なDNA認識機構は両者において高度に保存されていることが示唆された。一方、SpCas9とSaCas9のPAM特異性の違いと一致して、SaCas9はPIドメインのAsn985、Asn986、Arg991、Arg1015を用いてNNGRRT PAMを認識していた。SpCas9とSaCas9の構造比較から、両者の間のドメイン構造の違いも明らかになった。SpCas9と比較し、SaCas9はコンパクトなRECローブをもっていた。さらに、SpCas9とSaCas9において二本鎖DNAとガイドRNAの間に結合するドメイン(WEDドメイン)は大きく構造が異なっていた。これらの比較から、SpCas9とSaCas9は構造の異なるRECドメインおよびWEDドメインを用いてsgRNAの構造の違いを特異的に認識していることが明らかになった。さらに、構造情報をもとに、SaCas9を用いた転写活性化ツールおよび、誘導型SaCas9の作製に成功した[16][16] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.。
5. FnCas9の結晶構造
FnCas9(1629残基)はCas9タンパク質の中でも最もサイズが大きく、SpCas9やSaCas9との配列相同性も低い。FnCas9の作動機構を解明するために、FnCas9-sgRNA-DNA複合体の結晶構造を決定した[6][6] H. Hirano et al.: Cell 164 (2016) 950-961.(図2D)。FnCas9はSpCas9やSaCas9と類似のRuvC/HNHドメインをもつ一方、FnCas9のREC/WEDドメインは新規フォールドをとっていた。REC/WEDドメインの構造の違いと一致して、sgRNAの構造も大きく異なっていた。結晶構造から、NGG PAMはPIドメインのArg1556、Arg1585によって認識されることが明らかになった。SpCas9とFnCas9の構造比較から、両者のNGG PAM認識機構は異なることが明らかになった。さらに、構造情報をもとにFnCas9に3つの変異を導入し、YG(YはTまたはC)という配列をPAMとして認識するFnCas9改変体の作製に成功した[6][6] H. Hirano et al.: Cell 164 (2016) 950-961.。
6. AsCpf1の結晶構造
Cpf1はRuvCドメインを除きCas9を含む既知タンパク質と相同性をもたないため、そのDNA切断機構は不明だった。Cpf1によるDNA切断機構を解明するために、AsCpf1-crRNA-DNA複合体の結晶構造を決定した[17][17] T. Yamano et al.: Cell 165 (2016) 949-962.(図2E)。結晶構造から、Cpf1は2つのローブ(REC/NUC)からなることが明らかになった。RECローブは2つのドメイン(REC1/REC2)から構成され、NUCローブは4つのドメイン(RuvC/WED/PI/Nuc)から構成されていた。RuvCドメインを除く5つのドメインは新規フォールドをもっていた。crRNAの5′末端領域はシュードノット構造をとりWED/RuvCドメインによって認識されていた。一方、crRNAのガイド配列は標的DNAとRNA-DNAヘテロ二本鎖を形成し、2つのローブの間に収容されていた。TTTA PAMを含む二本鎖DNAは歪んだ二重らせん構造をとり、WED/REC1/PIドメインにより構造・配列特異的に認識されていた。NucドメインはRuvCドメインの近傍に位置し、相補鎖DNAの切断に関与することが明らかになった。RuvCドメインを除きCas9とCpf1は配列相同性をもたないが、両者は2つのローブから構成される全体構造を共通してもっていた。一方、PAM認識・DNA切断機構は大きく異なっていた。これらの比較から、Cas9とCpf1の間の機能収斂が明らかになった。
7. おわりに
近年の構造機能研究の進展により、Cas9やCpf1によるDNA切断機構の大枠は解明された。しかし、DNA切断におけるダイナミクスや触媒反応の詳細は不明である。さらに、最近の研究から、新規のCRISPR-Casヌクレアーゼも発見されている[18,19][18] O. O. Abudayyeh et al.: Science 353 (2016) aaf5573.
[19] D. Burstein et al.: Nature doi:10.1038/nature21059 (2016).。今後の研究により、多様なCRISPR-Casヌクレアーゼの作動機構の全容解明、および、新規のゲノム改変ツールの開発が期待される。
謝辞
本研究は、MITのFeng Zhang博士、東京大学大学院理学系研究科の石谷隆一郎准教授、濡木理教授との共同研究として行った。
X線回折実験は、SPring-8のBL32XUおよびBL41XU(課題番号2014A1356、2014B1223、2015A0119、2015B0119、2016A0119、2016B0119)において行った。
本研究は、JST戦略的創造研究推進事業(さきがけ)、JSPS科研費(26291010、15H01463)、文部科学省及び国立研究開発法人日本医療研究開発機構 創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業)の支援を受けて行った。
参考文献
[1] R. Barrangou et al.: Science 315 (2007) 1709-1712.
[2] K. S. Makarova et al.: Nat Rev Microbiol 13 (2015) 722-736.
[3] H. Nishimasu and O. Nureki: Curr Opin Struct Biol 43 (2016) 68-78.
[4] M. Jinek et al.: Science 337 (2012) 816-821.
[5] F. A. Ran et al.: Nature 520 (2015) 186-191.
[6] H. Hirano et al.: Cell 164 (2016) 950-961.
[7] L. Cong et al.: Science 339 (2013) 819-823.
[8] B. Zetsche et al.: Cell 163 (2015) 759-771.
[9] H. Nishimasu et al.: Cell 156 (2014) 935-949.
[10] C. Anders, O. Niewoehner, A. Duerst and M. Jinek: Nature 513 (2014) 569-573.
[11] S. Konermann et al.: Nature 517 (2015) 583-588.
[12] B. P. Kleinstiver et al.: Nature 529 (2016) 490-495.
[13] I. M. Slaymaker et al.: Science 351 (2016) 84-88.
[14] B. P. Kleinstiver et al.: Nature 523 (2015) 481-485.
[15] S. Hirano, H. Nishimasu, R. Ishitani and O. Nureki: Mol Cell 61 (2016) 886-894.
[16] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.
[17] T. Yamano et al.: Cell 165 (2016) 949-962.
[18] O. O. Abudayyeh et al.: Science 353 (2016) aaf5573.
[19] D. Burstein et al.: Nature doi:10.1038/nature21059 (2016).
※用語解説
・ゲノム編集
細胞の中でゲノムDNAが切断されると、修復機構により切断部位の再結合が起こる。この際、塩基の欠損や挿入などの変異が偶発的に生じる。ゲノム編集技術は、ゲノムDNAの狙った部位を人為的に切断し、修復過程における偶発的な変異を利用することによりゲノム情報を「書き換える」技術である。制限酵素は6塩基程度を認識し二本鎖DNAを切断するのに対し、ZFN(zinc finger nuclease)やTALEN(transcription activator-like effector nuclease)などの人工ヌクレアーゼは20塩基以上の配列を認識し二本鎖DNAを切断する。したがって、人工ヌクレアーゼを用いることによりゲノムDNAの標的部位を選択的に切断しゲノム編集を行うことが可能である。しかし、ZFNやTALENはタンパク質モジュールにより塩基配列を認識するため、標的配列ごとにタンパク質を設計しなければならないという問題点があった。一方、Cas9はsgRNAのもつガイド配列(20塩基)と相補的な二本鎖DNAを切断し、sgRNAのガイド配列は変更することができるため、簡便で迅速なゲノム編集ツールとして広く普及した。
東京大学大学院 理学系研究科
〒113-0032 東京都文京区弥生2-11-16
TEL : 03-5841-4391
e-mail : nisimasu@bs.s.u-tokyo.ac.jp








