Volume 21, No.1 Pages 2 - 4
1. 最近の研究から/FROM LATEST RESEARCH
時計タンパク質KaiCの「遅さ」が刻み込まれた原子構造
Slow ATP Hydrolysis Reaction Designed in the Atomic Structure of Circadian Clock Protein KaiC
分子科学研究所 協奏分子システム研究センター Research Center of Integrative Molecular Systems, Institute for Molecular Science
- Abstract
- シアノバクテリアの生物時計はKaiA、KaiB、KaiCの3種類のタンパク質から構成され、これらのタンパク質とATPを混ぜることで試験管内に約24時間周期の反応を再現することができる。近年、KaiCのATP加水分解活性が生物時計の周期決定に深く関与していることが指摘された。しかしながら、一般に加水分解を引き起こすタンパク質の運動はピコ秒~秒の時間域で起こり、KaiCがどのように約24時間周期の遅い反応を作り出しているのか現在まで解明されていなかった。我々は構造生物学、生化学、計算科学を駆使することで24時間の遅さと周期性がKaiCに実装されていることを原子レベルで解明した。
1. これまでのシアノバクテリアの生物時計研究
太古の時代から生物は、地球の自転によって生じる周期的な環境の変化に適応するため、自らの生理機能を周期的に変化させる仕組み(生物時計)を保持している。生物時計は約24時間周期の遅い反応であり、その発振機構として時計遺伝子の転写・翻訳フィードバック機構が提唱されている。これは、時計遺伝子から翻訳された時計タンパク質が自身の発現を抑制し、発現とその抑制との間に時間的なずれが生じることで発振するというモデルである。このモデルは、哺乳類などの高等生物において広く受け入れられている。
生物時計を持つ最も下等な生物としてシアノバクテリアがある。時計遺伝子(kaiA、kaiB、kaiC)の同定、それによる転写・翻訳フィードバック機構の提唱[1][1] M. Ishiura et al.: Science 281 (1998) 1519-1523.からわずか7年後、転写・翻訳を停止させても生物時計が働くことが明らかとなり、翻訳産物であるKaiタンパク質(KaiA、KaiB、KaiC)が時計の実体であることが証明された[2][2] J. Tomita et al.: Science 307 (2005) 251-254.。これとほぼ同時期に、3種類のKaiタンパク質とアデノシン三リン酸(ATP)を混合すると、試験管内で生物時計を再構成できることが発見された[3][3] M. Nakajima et al.: Science 308 (2005) 414-415.。これら一連の発見を皮切りに、タンパク質のみで働く生物時計の研究が精力的に行われるようになった。
近年、KaiCのATP加水分解(ATPase)活性と生物時計の周期が強く相関していることが示された[4][4] K. Terauchi et al.: Proc. Natl. Acad. Sci. USA 104 (2007) 16377-16381.。このATPase活性は非常に低く(1日に12個のATPしか加水分解されない)、温度の影響をほとんど受けない。これらの実験結果からKaiCの加水分解反応が他のATPaseに比べて非常に遅く、その遅さが時計の時間を決定していると推測できる。我々はKaiCの加水分解反応の詳細を理解するため、X線結晶構造解析を主軸とし、生化学的解析を併用することで多角的な視点から研究を行ってきた[5][5] J. Abe et al.: Science 349 (2015) 312-316.。
2. 「24時間」の遅さを実装したKaiC
KaiCはN末端リング(C1)とC末端リングから構成されている。C1のATPase活性は細胞内、試験管内それぞれの生物時計の周期と強く相関することが示された(図1A)。このことは、C1のATPaseが「24時間」の遅さを生み出す重要な反応であることを示している。
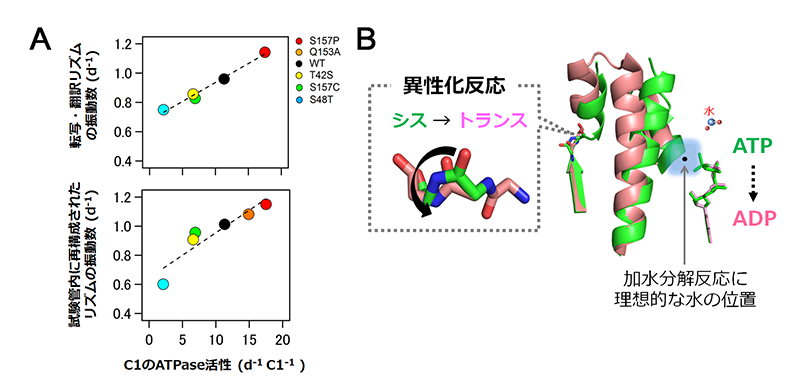
| 図1 | (A) C1のATPase活性が細胞内の生物時計の周期を決定する。 C1のATPase活性は、細胞内の転写・翻訳リズム(上段)、および試験管内で再構成されたリズム(下段)と相関している。図は文献5より引用。 |
| (B) 「遅さ」を生み出す構造的要因 加水分解と連動して、シス型からトランス型へ主鎖の異性化反応が起こる。青色の網掛けは立体障害の領域を模式的に示している。 |
C1のATPaseの反応機構を解明する契機となったのは、ATPとアデノシン二リン酸(ADP)を結合したサブユニットが同一リング内に混在した状態で構造解析されたことである。その結晶構造には、ATP加水分解の反応速度を「24時間」の時間スケールまで遅くし得る2つの構造的要因が見出された(図1B)。1つ目は、ATPを攻撃する水分子の位置である。C1では、ATPのγリン酸基近傍がヘリックスの存在により混み合っていた。これが立体障害として働き、水分子がリン酸基へ接近しにくい(効率よく加水分解反応が起こりにくい)配置になっていた。
2つ目は、上述の立体障害が容易に解消されないようにする仕組みである。水分子がリン酸基へ接近するためには立体障害の解消が必須となるが、それが容易におこらないようペプチド主鎖の異性化反応によって抑制制御されていた。一般にペプチドの異性化反応は生体内で起こる最も遅い反応のひとつとして知られている。計算機シミュレーションからも、異性化反応を伴いつつ加水分解反応を進行させるためには大きな自由エネルギー障壁を越えなければならないことが示唆された。
3. 「24時間」の周期性を実装したKaiC
KaiCのATPase活性はKaiAとKaiBの共存下で発振する(図2A)。定常状態にあるKaiCのATPase活性(12 ATP d-1 KaiC-1)をほぼ中央値として、より高く(28 ATP d-1 KaiC-1)なったり、低くなったり(5 ATP d-1 KaiC-1)を繰り返す。KaiCの視点に立てば、発振中は常に自身の定常状態から逸脱していること(非定常状態)になる。
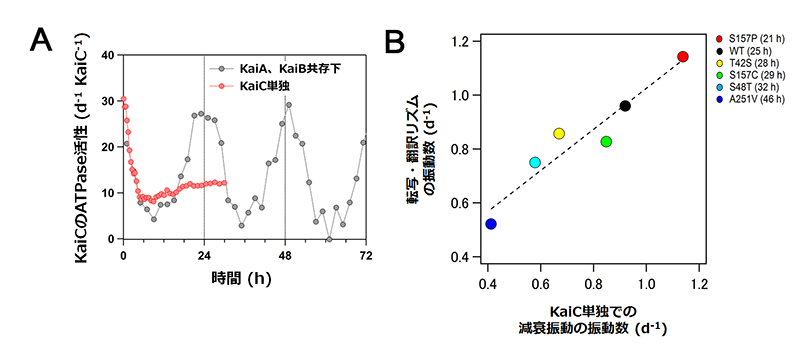
| 図2 | (A) KaiCは単独で減衰振動を示す。 KaiC単独のATPase活性は減衰振動(赤色)を示す(振動数は0.91 d-1)。KaiAとKaiBが共存すると(灰色)、KaiCのATPase活性は安定に振動する。 |
| (B) 「24時間」の周期性を実装したKaiC 周期変異体(短周期型、長周期型)のATPase活性は野生型と同様に減衰振動を示し、その振動数は細胞内の転写・翻訳リズムと相関する。 括弧内の数字は生物時計の周期を示す。 |
|
| (A)と(B)の図は文献5より引用。 |
非定常状態でのATPase活性を詳しく調べるため前定常状態解析を行った。この解析法は、ATPと結合していないKaiCが「ATPの結合、加水分解、生成物であるADPの放出」という反応サイクルを繰り返し、最終的に定常状態へと至る過程[6][6] A. Mukaiyama et al.: BIOPHYSICS 11 (2015) 79-84.を観察することである。このATPase活性の時間変化を測定すると、KaiC単独であっても、ゆっくりとした減衰型の振動成分が確認され(図2A)、その振動数(0.91 d-1)は細胞内における時計の振動数(生物時計の周期の逆数)とよく一致していた。この一致は、24時間よりも短い(短周期型)および長い周期(長周期型)を示すKaiC変異体においても確認された(図2B)。KaiCには「24時間」の周期性が実装されており、その時間スケールはATPase活性の高低によって制御されている。
4. おわりに
本研究は、タンパク質が運動や反応の効率を抑制制御(負帰還制御)することで、「24時間」を実装できることを明らかにした例と言ってよい。また、その抑制制御機構に水分子、ATP、ポリペプチド鎖といった生物が普遍的に持っている物質が使用されていることから、他の生物種においても類似の分子機構の存在を想像させる。実際、高等生物でも転写・翻訳のフィードバック機構によらないリズムが報告されつつある[7][7] J. S. O'Neill and A. B. Reddy: Nature 469 (2011) 498-503.。今後、時計タンパク質が担う化学反応(本研究ではATPase)を対象に、「遅さ」や「周期との相関」に着目した構造生物学的解析を行うことで、生物時計の構成原理により深く切り込むことが可能となるであろう。
謝辞
本研究は、分子科学研究所理論・計算分子科学研究領域の森俊文助教、斉藤真司教授、大阪大学蛋白質研究所の山下栄樹助教、名古屋大学大学院理学研究科のSeyoung Son博士、近藤孝男教授との共同研究である。
X線回折像の取得は、SPring-8のBL44XUにおいて課題番号2009A6902、2009B6902、2010A6502、2010B6502、2011A6602、2011B6602、2012A6702、2012B6702、2013B6700で行った。
本研究は外部資金として、科研費、文部科学省 創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業)、岡崎オリオンプロジェクトの一部の支援を受けて実施した。
参考文献
[1] M. Ishiura et al.: Science 281 (1998) 1519-1523.
[2] J. Tomita et al.: Science 307 (2005) 251-254.
[3] M. Nakajima et al.: Science 308 (2005) 414-415.
[4] K. Terauchi et al.: Proc. Natl. Acad. Sci. USA 104 (2007) 16377-16381.
[5] J. Abe et al.: Science 349 (2015) 312-316.
[6] A. Mukaiyama et al.: BIOPHYSICS 11 (2015) 79-84.
[7] J. S. O'Neill and A. B. Reddy: Nature 469 (2011) 498-503.
分子科学研究所 協奏分子システム研究センター
〒444-8585 愛知県岡崎市明大寺町字西郷中38番地
TEL : 0564-55-7366
e-mail : jabe@ims.ac.jp
分子科学研究所 協奏分子システム研究センター
〒444-8585 愛知県岡崎市明大寺町字西郷中38番地
TEL : 0564-55-7366
e-mail : amukai@ims.ac.jp
分子科学研究所 協奏分子システム研究センター
〒444-8585 愛知県岡崎市明大寺町字西郷中38番地
TEL : 0564-55-7363
e-mail : akiyamas@ims.ac.jp








