Volume 09, No.5 Pages 367 - 371
2. 最近の研究から/FROM LATEST RESEARCH
藍色細菌の時計タンパク質KaiAの構造と機能 機能ドメイン、X線結晶構造解析及び構造─機能相関の解明
Role of Cyanobacterial Circadian Clock Protein KaiA as Revealed by Crystal Structure
[1]名古屋大学 遺伝子実験施設 Center for Gene Research, Nagoya University、[2]京都大学大学院 薬学研究科 Graduate School of Pharmaceutical Sciences, Kyoto University、[3]京都大学大学院 薬学研究科 Graduate School of Pharmaceutical Sciences, Kyoto University、[4]名古屋大学 遺伝子実験施設 Center for Gene Research, Nagoya University
- Abstract
- KaiA, KaiB, and KaiC constitute the circadian clock machinery in cyanobacteria. KaiA activates kaiBC expression while KaiC represses it. Here we demonstrated that KaiA is composed of three functional domains : the N-terminal amplitude-amplifier domain, the central period-adjuster domain, and the C-terminal clock-oscillator domain. The C-terminal domain is responsible for dimer formation, binding to KaiC, enhancing KaiC phosphorylation, and generating circadian oscillations. The 1.8 Å X-ray crystal structure of the C-terminal clock-oscillator domain of KaiA from the thermophilic cyanobacterium Thermosynechococcus elongatus BP-1 shows that residue His270, located at the center of a KaiA dimer concavity, is essential to KaiA function. KaiA binding to KaiC likely occurs via the concave surface. Based on the structure, we could predict the structural roles of the residues that affected circadian oscillations.
1.はじめに
多くの生物は地球上の昼夜の交替に合わせて様々な生理活性を24時間周期で変動させている。植物では葉の就眠運動や光合成活性の日周変動が、動物では昼行性、夜行性などの行動リズム、睡眠覚醒やホルモン代謝、細胞分裂等のリズムが知られている[1][1]E. Bünning : The Physiological clock, 3rd ed. Springer-verlag, New York (1973).。これらのリズムは光や温度が一定な恒常環境下においても、まるで昼夜があるかの様に約24時間周期で継続する。このリズムは概日リズムと呼ばれ、このリズムを制御している細胞内の分子機構が生物時計(概日時計)である。現在までに様々な生物種で生物時計の分子遺伝学的解析が行われており、藍色細菌やアカパンカビ、ショウジョウバエ、マウス等で時計遺伝子がクローニングされている[2][2]M. W. Young and S. A. Kay : Nature Rev. Genet. 2 (2001) 702-715.。藍色細菌は、生物時計の存在が知られている最も原始的な生物である。石浦らは1998年に常温性藍色細菌Synechococcus sp. strain PCC 7942(以下Synechococcus)から、時計遺伝子クラスターkaiABCをクローニングし、分子遺伝学的解析によりkaiABCが藍色細菌の生物時計本体の遺伝子であることを明らかにした[3][3]M. Ishiura et al. : Science 281 (1998) 1519-1523.。kaiABCは2つのオペロンより構成されており、時計タンパク質KaiAはkaiBCオペロンの発現を促進し、もう一つの時計タンパク質KaiCはkaiBCオペロンの発現を抑制する。さらに、時計機能を修飾する時計関連タンパク質SasAはkaiBCの発現を促進し、時計の発振を増幅する[4][4]H. Iwasaki et al. : Cell 101 (2000) 223-233.。これらの事実に基づいて、時計発振のメカニズムとして遺伝子発現のフィードバックモデルを提唱した(図1)[3][3]M. Ishiura et al. : Science 281 (1998) 1519-1523.。しかしながら、時計タンパク質がどの様な分子機構で時計を発振させ、周期を24時間に調節しているのかはまだ分かっていない。
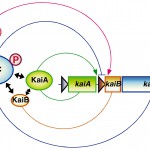
図1 藍色細菌の生物時計のフィードバック制御モデルの概念図
我々は、生物時計を時計タンパク質や時計関連タンパク質から構成される精巧な分子装置と捉え、まず時計装置の構成タンパク質のドメイン構造や原子構造、それらの複合体の原子構造を解明することにした。次に、原子構造に基づいてin vivoリズム解析及びin vitro生化学解析を行い、各タンパク質の機能ドメインや機能モチーフ、推定される重要アミノ酸残基の役割を解明することにした。我々は、各構成タンパク質の原子構造と時計発振機能との相関を解明することが、生物時計装置の作動原理を原子レベルで解明することにつながると考えている。本年、我々は時計タンパク質KaiAの構造・機能ドメインを解明し、時計の発振に必須なC末端時計発振ドメインの原子構造をSPring-8の放射光を用いて1.8Åの分解能で解明した[5][5]T. Uzumaki et al. : Nature Struct. Mol. Biol. 11 (2004) 623-631.。本稿では、原子構造に基づいた時計の発振機構について紹介する。
2.KaiAの3ドメイン構造
現在までに、好熱性や糸状性などの生育環境や形態が異なる13種の藍色細菌でkaiABCが見つかっている。これらの藍色細菌のKaiAのアミノ酸配列のアライメント解析により、KaiAは、アミノ酸残基のほとんど保存されていないN末端ドメイン、比較的保存されている中央ドメイン、高度に保存されているC末端ドメイン、の3つの構造ドメインから構成されていることが明らかになった(図2)。13種の全てで保存されている残基は22残基のみで、全てC末端ドメインに局在していた。糸状性藍色細菌Anabaena sp. strain PCC 7120(以下Anabaena)のKaiAは、C末端ドメインのみから構成され、N末端ドメイン及び中央ドメインを欠失していた。別府温泉産の好熱性藍色細菌Thermosynechococcus elongatus BP-1のKaiAのプロテアーゼによる限定分解からも、KaiAが3つの構造ドメインから構成されていることが明らかになった [5][5]T. Uzumaki et al. : Nature Struct. Mol. Biol. 11 (2004) 623-631.。

図2 KaiAの3ドメイン構造と機能
N末端ドメインを水色、中央ドメインを橙色、C末端ドメインを緑色で示してある。
次に、SynechococcusのkaiA欠損変異株で、Synechococcus KaiAの各ドメインをそれぞれ発現させ、各構造ドメインのin vivoリズム解析を行った。その結果、N末端ドメインは振幅を増幅する機能を、中央ドメインは周期を24時間に調節する機能を、C末端ドメインは時計発振機能を担っていることが明らかになった。Anabaena KaiAとSynechococcus KaiAの各ドメインとで構成されるキメラタンパク質でも同様のことを確認した。さらにSynechococcus KaiA及びT. elongatus KaiAのドメイン欠失タンパク質を用いたin vitro生化学解析により、KaiAの生化学活性として知られている、二量体形成能[6][6]F. Hayashi et al. : Biochem. Biophys. Res. Commun. 316 (2004) 195-202.、KaiC結合能[7][7]H. Iwasaki et al. : EMBO J. 18 (1999) 1137-1145.、KaiCリン酸化促進能[8][8]H. Iwasaki et al. : Proc. Natl. Acad. Sci. USA. 99 (2002) 15788-15793.の全てがC末端ドメインに局在することを明らかにした[5][5]T. Uzumaki et al. : Nature Struct. Mol. Biol. 11 (2004) 623-631。そこで、時計発振機能に必須なC末端時計発振ドメインを結晶化し、X線結晶構造解析を行うことにした。
3.X線結晶構造解析
T. elongatus KaiAの時計発振ドメインを大腸菌で大量発現させ、高度に精製し、正八面体の結晶(P43212)を得ることができた(図3)。また位相決定のためにメチオニンの代わりにセレノメチオニンを取り込ませたタンパク質で結晶を作製した。SPring-8のビームラインBL26B1、BL26B2を用いてX線回折データを収集し、多波長異常分散法で解析し、1.8Åの分解能で原子構造を解明した。

図3 T. elongatus KaiA C末端ドメインの結晶
時計発振ドメインのサブユニットは6本のヘリックスからなり、右回りのスーパーヘリックス構造をとっていた(図4)。時計発振ドメインの全体構造は二量体で、二量体の空間充填モデルは2つの凹面を持つ凹レンズ型の形状をしていた(図5)。
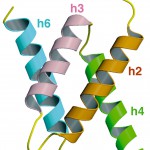
図4 サブユニットのリボンモデル
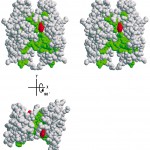
図5 二量体充填モデル(ステレオ図)と保存残基の位置
13種の藍色細菌で保存されているアミノ酸残基は緑色で示してある。ただし、時計発振機能に必須なHis270は赤色で示してある。
4.原子構造から分かったこと
これまで、どの残基がどの様に時計発振に関与しているかは全く分かっていなかった。一般に、進化を通じて保存されている残基は何らかの機能を担っていると考えられる。そこで、時計発振ドメインで保存されている22個のアミノ酸残基を原子構造上にマッピングしてみた。その結果、ほとんど全ての保存残基はKaiA分子の凹面中央部に並ぶか、分子の内側に側鎖を伸ばしていた(図5)。凹面最深部に位置するヒスチジン残基(His270)は、分子の外側に側鎖を伸ばしており(図5)、構造維持には関与しておらず、何らかの機能を担っていることが推定された。そこで、このHis270残基をアラニン残基に置換した一アミノ酸置換変異体を作製し、時計発振機能への影響を調べた。
T. elongatus KaiAをSynechococcus kaiA欠損変異株で発現させると、リズム発振は回復したが、そのリズムは非常に低振幅で、詳細な解析には適していなかった。そこで、Synechococcus kaiA欠損変異株でT. elongatus H270A変異に相当するSynechococcus KaiA H271A変異を持つ株を作製し、in vivoリズム解析を行った。対照の野性型KaiAを発現する株は正常なリズムを発振したが、この株は全くリズムを発振しなかった(図6)。したがってSynechococcusのHis271残基(T. elongatus His270に相当)は時計発振に必須な残基であることが明らかになった。次に、T. elongatus KaiA H270A変異タンパク質を用いて、in vitro生化学解析を行ったところ、KaiC結合能、KaiCリン酸化促進能は野生型KaiAの3割に減少していた。さらに円偏光二色計を用いてH270A変異タンパク質の耐熱性を解析した。その結果、H270A変異は、二次構造や耐熱性にはほとんど影響を与えていなかった。したがって、His270はKaiAの時計発振機能に必須でin vitro活性に重要な残基であると結論した。この様に、原子構造に基づいてin vivo、in vitroの機能解析を行うことにより、時計発振に必須な残基を世界で初めて同定することができた。
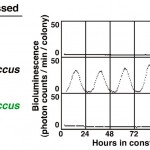
図6 H271A変異体のin vivoリズム解析
Synechococcus kaiA欠損変異株(宿主;上段)、野生型KaiA発現株(中段)、KaiA H271A変異タンパク質発現株(下段)のリズム。
これまで、概日リズムの周期や振幅に影響を与えるSynechococcus KaiAの一アミノ酸置換変異が多数報告されている[3, 9, 10][3]M. Ishiura et al. : Science 281 (1998) 1519-1523.
[9]Y. Taniguchi et al. : FEBS Lett. 496 (2001) 86-90.
[10]H. Nishimura et al. : Microbiology 148 (2002) 2903-2909.。これらの変異を持つSynechococcus変異体が発振するリズムは大きく分けて「長周期・低振幅リズム」と「軽微な周期延長リズム」とに分類できる(図7A)。しかし、これらの変異がどの様に時計発振に影響を与えているのかは全く分かっていなかった。そこで、KaiAの原子構造に基づいて、一アミノ酸置換変異とリズム変異との因果関係を解析した。その結果、分子の内部に位置し分子構造に大きな影響を与える変異(F224S、F225S、M241T、C273Y、E274K;図7B)は、「長周期・低振幅」の大きなリズム変異を引き起こした。一方、分子内部に位置していても類似残基への置換変異(I266V、F244V;図7C)であったり、分子表面に存在していたりで(E239G、E243A、D242G、D242V、A245D、R249H;図7C)、構造にはほとんど影響を与えない変異は、「軽微な周期延長」のリズム変異を引き起こした。
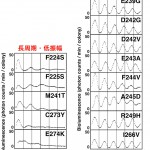
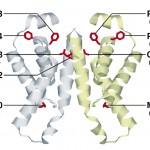

図7 一アミノ酸置換変異体のリズムと原子構造上の位置
A. 一アミノ酸置換変異体のリズム。
B.「長周期・低振幅リズム」を引き起こす残基の位置。変異を引き起こす残基の側鎖を赤色で示してある。
C.「軽微な周期延長リズム」を引き起こす残基の位置。変異を引き起こす残基で、分子内部に位置し、類似残基へ置換されていた残基の側鎖を黄色で、分子表面に位置している残基の側鎖を水色で示してある。BとCに表示してある各残基の下のかっこ内には相当するSynechococcusの残基を示してある。
5.おわりに
我々はC末端時計発振ドメインの原子構造に基づいたin vivoリズム解析とin vitro 生化学解析により機能上重要と推定される各アミノ酸の役割を解明し、時計発振機能を原子レベルで解明する先駆者となった。今後は、他の時計タンパク質や時計関連タンパク質、またそれらの複合体の原子構造を解明したい。
2004年の上半期に、我々に加え、アメリカのVakonakisら[11][11]I. Vakonakis et al. : Proc. Natl. Acad. Sci. USA. 101 (2004) 1479-1484.とYeら[12][12]S. Ye et al. : J. Biol. Chem. 279 (2004) 20511-20518.、カナダのGarcesら[13][13]R. Garces et al. : EMBO J. 23 (2004) 1688-1698.が相次いでKaiAの原子構造を報告した。各グループにより報告されたKaiAのC末端ドメインの構造は非常に良く似ており、本質的に同じであった。
本年、筆者らはX線結晶構造解析によりT. elongatusの時計タンパク質KaiBの原子構造を、SPring-8の放射光(ビームラインBL41XU)を用いて2.2Åの分解能で解明した[岩瀬ら、未発表データ]。上記カナダのグループも2004年にAnabaena KaiBの構造を2.2Åの分解能で報告している[13][13]R. Garces et al. : EMBO J. 23 (2004) 1688-1698.。2004年にアメリカのPattanayekらはKaiCの構造を2.8Åの分解能で報告した[14][14]R. Pattanayek et al. : Mol. Cell 15 (2004) 375-388.。2004年にVakonakisらは、KaiAのC末端時計発振ドメインとKaiCのC末端側30残基の短いペプチドとの複合体の構造をNMRで解明した[15][15]I. Vakonakis et al. : Proc. Natl. Acad. Sci. USA. 101 (2004) 10925-10930.。この半年の間に、時計タンパク質の原子構造が続々と明らかになってきている。今後は時計タンパク質KaiA-KaiB-KaiC複合体の原子構造の解明とそれに基づく構造-機能相関の解明が、時計発振の分子機構の解明の鍵となる。また構造解析だけでなく、各時計タンパク質の分子動力学シミュレーションや細胞内における時計タンパク質複合体の形成・解離の時空間変動の解析などの様々な解析が、時計発振の分子機構の解明に不可欠である。
本研究において、理化学研究所播磨研究所メンブレンダイナミクス研究チームの柴田洋之研究員、名古屋大学遺伝子実験施設の林史夫研究員、藤田真康氏、伊藤典代氏にご協力頂いた。X線回折実験ではSPring-8理研ビームラインのスタッフの皆さんにご協力頂いた。この場を借りて感謝の意を表したい。
参考文献
[1]E. Bünning : The Physiological clock, 3rd ed. Springer-verlag, New York (1973).
[2]M. W. Young and S. A. Kay : Nature Rev. Genet. 2 (2001) 702-715.
[3]M. Ishiura et al. : Science 281 (1998) 1519-1523.
[4]H. Iwasaki et al. : Cell 101 (2000) 223-233.
[5]T. Uzumaki et al. : Nature Struct. Mol. Biol. 11 (2004) 623-631.
[6]F. Hayashi et al. : Biochem. Biophys. Res. Commun. 316 (2004) 195-202.
[7]H. Iwasaki et al. : EMBO J. 18 (1999) 1137-1145.
[8]H. Iwasaki et al. : Proc. Natl. Acad. Sci. USA. 99 (2002) 15788-15793.
[9]Y. Taniguchi et al. : FEBS Lett. 496 (2001) 86-90.
[10]H. Nishimura et al. : Microbiology 148 (2002) 2903-2909.
[11]I. Vakonakis et al. : Proc. Natl. Acad. Sci. USA. 101 (2004) 1479-1484.
[12]S. Ye et al. : J. Biol. Chem. 279 (2004) 20511-20518.
[13]R. Garces et al. : EMBO J. 23 (2004) 1688-1698.
[14]R. Pattanayek et al. : Mol. Cell 15 (2004) 375-388.
[15]I. Vakonakis et al. : Proc. Natl. Acad. Sci. USA. 101 (2004) 10925-10930.
宇津巻 竜也 UZUMAKI Tatsuya
名古屋大学 遺伝子実験施設 植物ゲノム解析分野
〒464-8602 名古屋市千種区不老町
TEL:052-789-4527 FAX:052-789-4526
e-mail:uzumaki@gene.nagoya-u.ac.jp
中津 亨 NAKATSU Toru
京都大学大学院 薬学研究科 創薬科学専攻・構造生物薬学分野
〒606-8501 京都市左京区吉田下阿達町46-29
TEL:075-753-4606 FAX:075-753-9272
e-mail:nakatsu@pharm.kyoto-u.ac.jp
加藤 博章 KATO Hiroaki
京都大学大学院 薬学研究科 創薬科学専攻・構造生物薬学分野
〒606-8501 京都市左京区吉田下阿達町46-29
TEL:075-753-4606 FAX:075-753-9272
e-mail:katohiro@pharm.kyoto-u.ac.jp
石浦 正寛 ISHIURA Masahiro
名古屋大学 遺伝子実験施設 植物ゲノム解析分野
〒464-8602 名古屋市千種区不老町
TEL:052-789-4527 FAX:052-789-4526
e-mail:ishiura@gene.nagoya-u.ac.jp
BL26B2 2003年1月28日 1.5シフト
2003年2月20日 1.5シフト
BL26B1 2003年3月9日 3シフト
2003年3月22日 3シフト








