Volume 09, No.4 Pages 265 - 268
3. 最近の研究から/FROM LATEST RESEARCH
時分割X線小角散乱が明らかにする蛋白質の収縮と構造形成
Compaction and Structure Formation of Proteins Revealed by Time-Resolved Small Angle X-ray Scattering
[1]大阪大学 蛋白質研究所 Institute for Protein Research, Osaka University、[2]京都大学大学院 工学研究科 Graduate School of Engineering, Kyoto University、[3](独)理化学研究所 播磨研究所 Harima Institute, RIKEN
- Abstract
- The compaction process in protein folding has not been characterized well due to experimental difficulties. The submillisecond-resolved observation system for small-angle X-ray scattering was developed and applied for the process of apomyoglobin folding. It was demonstrated that the secondary and tertiary structures are largely organized cooperatively; however, the initial folding phase involves a significant collapse of their main chain structures. A common folding mechanism was proposed, in which hydrophobic environments realized by the initial collapse prompts the subsequent formation of helical structures.
はじめに
蛋白質は、コンパクトな構造に折り畳むことで機能を発揮するポリペプチドである。蛋白質が折り畳んだ構造は、アミノ酸の一次配列により決められる。このため、アミノ酸の一次配列から蛋白質の折り畳み構造を予測することを目指した研究が活発に続けられている。この予測は、もし可能になれば、DNAに含まれる遺伝情報の理解や人工蛋白質の設計などに必須の手段となるはずである。最近では、比較的小さな蛋白質の構造予測はある程度可能になり[1]、自然にはない新規な構造をもつ蛋白質を設計・作成した例も報告されている[2][2]B. Kuhlman, G. Dantas, G. C. Ireton, G. Varani, B. L. Stoddard et al. : Science, 302 (2003) 1364-1368.。しかし、アミノ酸残基数が百数十よりも多く、多少複雑な構造を持つ蛋白質では、予測はいまだに難しいのが現状である。
構造予測が難しい蛋白質は、酸性溶液や高濃度の尿素溶液などに溶かして変性させた後で生理条件に戻すと、いくつかの中間体を通って折り畳み構造を再生することが多い。一方で、構造予測が簡単な蛋白質の多くは、中間体を作らずに一気に折り畳み構造を作り上げる。そのため、蛋白質の折り畳み過程で観察される中間体はなぜ生じるのか、これらはどのような構造を持つのかという疑問が生じる。これらの疑問について調べることは、構造予測にも重要な知見を与えると予想される。
我々は、蛋白質の折り畳み運動をリアルタイムで観察し、過渡的な折り畳み中間体の構造や性質を理解することを目的とした研究を続けている。特に、放射光を使った時分割X線小角散乱(SAXS)測定により、蛋白質が主鎖を収縮させながら次第に折り畳み構造を作る過程を詳しく観察する手法を確立した[3][3]S. Akiyama, S. Takahashi, T. Kimura, K. Ishimori, I. Morishima, Y. Nishikawa, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 99 (2002) 1329-1334.。本稿では、我々の実験手法による最近の研究成果を紹介する[4][4]T. Uzawa, S. Akiyama, T. Kimura, S. Takahashi, K. Ishimori, I. Morishima, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 101 (2004) 1171-1176.。
実験の狙い
折り畳んだ蛋白質構造の特徴は、コンパクトであることと、αヘリックスやβシートなどの二次構造を多く持つことである。そのため、これらの特徴が折り畳み中間体でどの程度形成しているのかを調べることは、中間体の意義を考える上で本質的な情報を与えると予想される。そこで我々は、SAXSと円二色性(CD)分光法を使った折り畳み過程の観測を行うことにした。SAXSは溶液中の分子のコンパクトさや形状を計測できる手段である[5][5]藤澤哲郎:生物物理, 43 (2003) 29-32.。また、CD分光法は、蛋白質の二次構造含量を観測する手法である[6][6]S. Akiyama, S. Takahashi, K. Ishimori, I. Morishima : Nature Struct. Biol., 7 (2000) 514-520 .。さらに、寿命の短い折り畳み中間体を観測するために、約100マイクロ秒で二つの溶液を混合する高速ミキサーを開発し、上記二つの手段を組み合わせた[3][3]S. Akiyama, S. Takahashi, T. Kimura, K. Ishimori, I. Morishima, Y. Nishikawa, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 99 (2002) 1329-1334.。BL45XUに設置した我々のSAXS計測システムは、〜1.3mg/mlという低濃度の試料でサブミリ秒の時分割測定を可能にする[4][4]T. Uzawa, S. Akiyama, T. Kimura, S. Takahashi, K. Ishimori, I. Morishima, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 101 (2004) 1171-1176.。この感度と時間分解能は、BL45XUの高い輝度を最大限生かした結果であり、世界で我々だけがもつ性能である。
我々は、開発した装置を使ってアポミオグロビン(apoMb)の折り畳み過程を観察した[4][4]T. Uzawa, S. Akiyama, T. Kimura, S. Takahashi, K. Ishimori, I. Morishima, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 101 (2004) 1171-1176.。apoMbは153残基から成り、中性の溶液条件では、A〜E・G・Hとラベルされる7本のヘリックスから構成される折り畳み構造を持つ(図1)[7][7]D. Eliezer, P. E. Wright: J. Mol. Biol., 263 (1996) 531-538. 。ヘリックス含量が大きいため、apoMbはヘリックスだけで構成される蛋白質の代表だと考えられる。apoMbは、pH2ではヘリックス含量が少なく回転半径の大きい酸変性状態(U状態)を作る。そのため、U状態にある蛋白質をpH6にジャンプさせることで、折り畳み過程を観察できる[8][8]P. A. Jennings, P. E. Wright : Science, 262 (1993) 892-896.。本研究では、apoMb溶液をpH2から6へジャンプさせた後の二次構造とコンパクトさの変化を、CDとSAXSを用いて検出した。
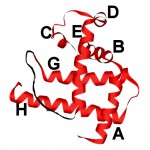
図1 apoMbの折り畳み構造
apoMbはpH6においてA〜E・G・Hとラベルされる7つのヘリックスを持つ折り畳み構造を形成する
測定結果
はじめに、時分割CD法でpHジャンプに伴うapoMbの折り畳み過程を観測した。得られた結果は、折り畳み開始後300マイクロ秒以内に30%のヘリックス含量を持った中間体Ⅰ1が形成し、次にヘリックス含量44%の中間体Ⅰ2が作られた後に、最終的な折り畳み状態N(ヘリックス含量55%)を形成するというモデルで解釈できた。すなわち、apoMbの折り畳みは次式のように進み、ヘリックス含量が段階的に変化した二つの中間体を含んでいる。
U → I1 → I2 → N
<300 µs 5 ms 50 ms>
次に、これらの中間体の回転半径と形状を観測するために、同じpHジャンプについて、時分割SAXS測定を行った。データをギニエ解析することにより得られた回転半径の時間変化をプロットしたものが、図2aである。驚いたことに、折り畳み開始後300マイクロ秒以内という短時間で、apoMbの回転半径が29.7Åから23.7Åへと大きく減少することがわかった。収縮した状態はⅠ1に対応する。また、Ⅰ2はⅠ1とほぼ同じ回転半径を持ち、CDで観察された時定数とほぼ同じ早さで、折り畳み状態N(回転半径18.2Å)に変化することも明らかとなった。
SAXSのデータをさらに解析することで、分子の形を推定できる。図2bに示したのは、散乱データをフーリエ変換して得られる距離分布関数である。距離分布関数は、分子の中の全ての原子間距離の分布を示し、主鎖の空間分布を推定する手がかりとなる。例えば、折り畳んだ状態の距離分布関数は、球状の構造に対応する対称的なベル型を示す。一方で、中間体Ⅰ1とⅠ2は、大きなピークと小さな肩を持つほぼ同じ形の距離分布関数を示した。これは、両状態が同じような形状を持ち、蛋白質の大部分が収縮した部分と、やや広がった小部分から成り立つことを示唆する。収縮ドメインの原子数を見積もると、分子全体の80%以上に達していた。
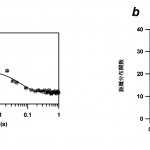
図2 apoMbの折り畳み過程の時分割SAXS測定結果
a)apoMbが折り畳み過程で示す回転半径の時間変化。縦軸に回転半径を、横軸に折り畳み開始後の時間をプロットした。b)折り畳み途中のapoMbの距離分布関数。青線は折り畳み開始後300µs、青点線は折り畳み開始後9ms、黒線は折り畳み状態を示す。折り畳み開始後300µsに凝集した部分(水色)は、全原子数の80%以上に対応した。
Ⅰ1状態のヘリックス含量は30%で、apoMbがpH4.2で作る平衡論的な中間体が持つA・G・Hヘリックスの含量と一致する[9][9]F. M. Hughson, P. E. Wright, R. L. Baldwin : Science, 249 (1990) 1544-1548.。しかし、これら三つのヘリックスだけでは、80%以上という収縮ドメインの原子数を説明できない。そこで我々は、まだヘリックスを形成していない主鎖領域もⅠ1状態の収縮ドメインに含まれると解釈した。さらに、Ⅰ2状態ではヘリックス含量が44%まで増えることから、Ⅰ1からⅠ2状態が作られるステップで、収縮ドメイン内部でヘリックス形成に伴う三次構造の再配向も起きると解釈した。
観察の意義
以上のapoMbの結果を、我々が以前に行ったシトクロムc(cyt c)の折り畳み過程についての結果と比較したい[3][3]S. Akiyama, S. Takahashi, T. Kimura, K. Ishimori, I. Morishima, Y. Nishikawa, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 99 (2002) 1329-1334.。両結果の比較のために、回転半径とヘリックス含量の相対変化をそれぞれXとY軸として図3にプロットした。apoMbは、U状態から出発して大きな収縮とヘリックスの形成を示した後(Ⅰ1)、ヘリックス含量のみを増やした中間体(Ⅰ2)を経由してN状態に至る。cyt cは、折り畳みの初期に収縮のみをみせた後(Ⅰ1)、ヘリックス含量とコンパクトさを成長させた中間体(Ⅰ2)を通って、N構造に変化する。これらの結果、第一の共通点として、二次構造を十分に作らないまま、折り畳みの初期に収縮が起きることが挙げられる。また、第二の共通点として、収縮が起きた後で二次構造の形成と蛋白質の収縮が同期して折り畳みが進むことが挙げられる。
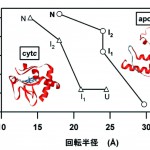
図3 apoMbとcyt cの折り畳み過程の比較
apoMb(○)とcyt c(△)が折り畳む過程で示す構造変化を、横軸に回転半径、縦軸にヘリックス含量として示した。
観察の第一の共通点から、我々は、折り畳み過程の最初に起こる運動は、疎水性相互作用による主鎖の収縮ではないか、と考えた。主鎖が収縮することにより、ポリペプチドの立体構造の自由度は大幅に制約される[10][10]K. A. Dill : Biochemistry 24 (1985) 1501-1509.。また、ポリペプチド周辺の疎水性環境はヘリックス形成能を変化させることも知られている[11][11]C. A. Rohl, A. Chakrabartty, R. L. Baldwin : Protein Sci., 5 (1996) 2623-2637.。これらの効果により、初期の収縮はその後の折り畳み運動を促進するのではないかと考えた。次に、第二の共通点から、ヘリックス形成と収縮が同期したことは、主鎖の正しい三次的な接触が形成された後にヘリックス構造が完成されることを示唆する。すなわち、主鎖が収縮した状態から折り畳み構造を探す運動の特徴は、一次配列上で離れた残基同士の正しい接触を探す運動であると解釈できる。我々は、以上の折り畳み機構を“Collapse and search mechanism”と名付けた[4][4]T. Uzawa, S. Akiyama, T. Kimura, S. Takahashi, K. Ishimori, I. Morishima, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 101 (2004) 1171-1176.。
提案した折り畳み機構の一般性を検討するために、我々は測定対象の蛋白質を増やしたいと考えている。特に、ヘリックスを多く持つ他の蛋白質でもapoMbと共通の機構が観察できるかどうか、βシートを含む蛋白質では変化が生じるか、などの興味に基づいた実験を進めている。また、折り畳み中間体を作らないとされる残基数が100以下の小蛋白質において、初期収縮が観察されるかどうかを調べる実験も計画している。これらの結果を比較することにより、構造予測にも役立つ一般的な蛋白質の性質を得ることが目標である。
SAXSは、変性も含めて蛋白質のコンパクトさを溶液中で観測できるほぼ唯一の手法である。しかし、実験の難しさから、それほど多くの蛋白質には適応されてこなかった。本稿で説明したように、放射光を利用することでSAXSの測定対象は大きく広がり、従来はほとんど不可能だった観測が可能になっている。我々は、今後も時分割SAXS観測を発展させ、手法の可能性を広げたいと考えている。
謝 辞
本研究を共同ですすめていただきました森島績教授、石森浩一郎博士、木村哲就さん(以上京大院工・分子工学)、秋山修志博士(理化学研究所・播磨)に感謝いたします。BL45XUの維持管理には前田雄一郎博士(理化学研究所・播磨)の支援をいただきました。本研究は科技団さきがけ研究と科研費により行われました。
参考文献
[1]P. Bradley, D. Chivian, J. Meiler, K. M. S. Misura, C. A. Rohl et al. : Proteins Struct. Funct. Genetics, 53 (2003) 457-468.
[2]B. Kuhlman, G. Dantas, G. C. Ireton, G. Varani, B. L. Stoddard et al. : Science, 302 (2003) 1364-1368.
[3]S. Akiyama, S. Takahashi, T. Kimura, K. Ishimori, I. Morishima, Y. Nishikawa, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 99 (2002) 1329-1334.
[4]T. Uzawa, S. Akiyama, T. Kimura, S. Takahashi, K. Ishimori, I. Morishima, T. Fujisawa : Proc. Natl. Acad. Sci. USA, 101 (2004) 1171-1176.
[5]藤澤哲郎:生物物理, 43 (2003) 29-32.
[6]S. Akiyama, S. Takahashi, K. Ishimori, I. Morishima : Nature Struct.
Biol., 7 (2000) 514-520 .
[7]D. Eliezer, P. E. Wright: J. Mol. Biol., 263 (1996) 531-538.
[8]P. A. Jennings, P. E. Wright : Science, 262 (1993) 892-896.
[9]F. M. Hughson, P. E. Wright, R. L. Baldwin : Science, 249 (1990) 1544-1548.
[10]K. A. Dill : Biochemistry 24 (1985) 1501-1509.
[11]C. A. Rohl, A. Chakrabartty, R. L. Baldwin : Protein Sci., 5 (1996) 2623-2637.

高橋 聡 TAKAHASHI Satoshi
大阪大学 蛋白質研究所
〒565-0871 吹田市山田丘2-1
TEL:06-6879-8615 FAX:06-6879-8616
e-mail : st@protein.osaka-u.ac.jp
略歴:1992年総研大博士(理学)。日米でのポストドクの後、1996年京大院工・分子工学助手。1999-2002科技団さきがけ研究員兼任。2003年より阪大蛋白研助教授。

鵜澤 尊規 UZAWA Takanori
京都大学大学院 工学研究科 分子工学専攻
〒615-8510 京都市西京区京都大学桂A4-134
TEL:075-383-2537 FAX:075-383-2541
e-mail : uzawa@tak.mbox.media.kyoto-u.ac.jp
略歴:2003年京大院工・分子工学修士修了。同博士課程在学中。

藤澤 哲郎 FUJISAWA Tetsuro
理化学研究所 播磨研究所 前田構造生物化学研究室
〒679-5148 佐用郡三日月町光都1-1-1
TEL:0791-58-2822 FAX:0791-58-1844
e-mail : fujisawa@sp8sun.spring8.or.jp
略歴:1989年大阪大博士(工学)。米でのポスドクの後、1990年より理化学研究所・研究員、1998年同先任研究員。








