Volume 05, No.6 Pages 389 - 393
3. 最近の研究から/FROM LATEST RESEARCH
筋小胞体カルシウムポンプの構造決定
Structure Determination of the Calcium Pump of Sarcoplasmic Reticulum
東京大学 分子細胞生物学研究所 Institute of Molecular and Cellular Biosciences, The University of Tokyo
- Abstract
- Structure of the calcium pump of muscle sarcoplasmic reticulum was determined at 2.6 Å resolution by X-ray crystallography. This is the first atomic structure of the cation pumps called P-type ATPases. Because the crystals were very thin, the use of SPring-8 was vital to this work. The structure of the Ca2+ -pump is overviewed very briefly.
はじめに
生体はイオンを非常に巧みに使っている。Na+ は細胞外に多くK+ は細胞内に多い。その結果として、代表的な細胞として筋細胞を例にとると、細胞の膜電位は内側が外側よりも約90mV低く保たれている。細胞が興奮するというのは、Na+ が外から濃度勾配に従ってチャネル蛋白質を通じて流入し、膜電位が0mV近く(或いはそれ以上)になってしまうことである。つまり、イオンの流入が神経細胞などの興奮の実体である。筋肉が収縮する時には、神経細胞の興奮によってカルシウムの放出チャネルが開き、カルシウムの貯め(筋小胞体と呼ばれる袋状のもの)から筋細胞中に放出される。そのカルシウムがアクチンフィラメント上のトロポニンに結合し、ミオシンがアクチンと結合できるようになって、すべり運動がおこる。さて、このままでは筋肉は収縮したまま、神経細胞も興奮したままである。もとの状態にイオン環境を戻してやらなければならない。そのためには、濃度勾配に従って流入してきたものを汲み出してやらなければならないわけで、ATPの持つ化学エネルギーを消費してイオンを濃度勾配に逆らって輸送する必要がある。この過程を担うものがイオンポンプと呼ばれる蛋白質である。
イオンポンプは生体膜の片側から反対側へイオンを運ぶのであるから、生体膜に埋まった膜蛋白質である。Na+ とK+ の濃度差を保つポンプはNa+ K+ -ATPaseと呼ばれ、カルシウムを運ぶポンプはCa2+ -ATPaseと呼ばれる。いずれも運搬するイオンによってスイッチがONになるATP分解酵素である。他にも胃のpHを維持しているH+ K+ -ATPaseや、銅などの重金属を排出するポンプがあり、総称してP型ATPase(あるいはP型イオンポンプ)と呼ばれる。ATPを分解して生成されるリン酸によって、酵素それ自体がリン酸化されるのでその名前がある。いずれも、機能しなくなったら細胞は生きていられなくなる、重要な機能を持った蛋白質群であり、Na+ K+ -ATPaseの発見(1957年)によって、1997年度のノーベル賞がデンマークのJ.C.スコウに授与されたほどのものである。
私達が構造決定に成功し[1][1]C. Toyoshima, M. Nakasako, H. Nomura, and H. Ogawa : Nature 405 (2000) 647-655.Natureの表紙にもなった蛋白質は、筋小胞体のカルシウムポンプであり、分子量11万、994個のアミノ酸残基から成る。P型ATPaseとして初めての構造決定というだけではなく、輸送するイオンがカルシウムであるところがこの仕事の意義を大きいものにしている。なぜなら、カルシウムは生体の機能を制御するためにもっともよく使われているイオンであり、国際カルシウム学会なるものがあるほどなのだ。なぜカルシウムなのかというのは面白い問題であるが、教科書的には①2価イオンであり、アミノ酸と程々強く結合する、②程々の大きさがあるため配位の仕方はかなりゆがんでいても良い(Mg2+ などはごく安定な8配位になる傾向が著しい)ため、蛋白質の側が選択性を持たすことが出来るからである。この事についてはあとで少しだけ触れる。
この仕事のもう一つの重要な点は結晶化であろう。Ca2+ -ATPaseは筋小胞体に大量に含まれており、ウサギ一匹から何十mgもとれる。精製も比較的簡単であり、そういう意味では誰が結晶化に成功してもおかしくない蛋白質といえる。出発点は2次元結晶が何層か重なっただけのごくごく薄い電子顕微鏡レベルの結晶であった。そこからうまくいった場合には2000層(30µm近く)まで、結晶を厚くすることが出来たわけである。それでもなお、機械的には弱い結晶であり、再現性も良くはないので、実験室でループにすくって急速凍結し(図1)、X線を照射して良い回折パターンが得られたものだけをSPring-8(BL44B2, BL41XU)に持ち込んで実験した(図2)。また、ごく薄い結晶であり、薄いものを選べばそこそこの電子顕微鏡像が得られ、一方向のみではあったが最初からかなり良い低角(6Å分解能まで)の位相が得られていた。この位相情報は重原子置換体の探索や溶媒平滑化の結果の検定に大変役に立った。

図1 実験室でナイロンループにすくい上げ、急速凍結したCa2+ -ATPaseの板状結晶。BL41XUで回折実験の直前に撮影。結晶の幅は300µm、厚さは20µm程度。

図2 Ca2+ -ATPase板状結晶からのX線回折パターン。BL44B2で得た初期のもの。波長は0.990Å、リガクRAXIS-Ⅳを使用。外側にあるぼやけたリングはガラス状の氷によるもの(〜1/3.7Å−1 )。
構造の概略
Natureに発表した論文[1][1]C. Toyoshima, M. Nakasako, H. Nomura, and H. Ogawa : Nature 405 (2000) 647-655.の要点をかいつまんで述べてみよう。
①カルシウムが2個結合した状態の構造であり、分解能は2.6Åである(図3)。
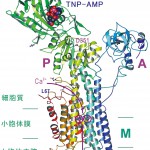
図3 Ca2+ -ATPaseの結晶構造の概略。この図ではM5ヘリックス(中央の黄緑色の長いへリックス)が紙面と平行で直立するように分子を置いているが、脂質二重膜の面はM5に対し(紙面に対しても)傾いている。M5へリックスの長さは60Åある。
②アミノ酸配列から予想されたとおり膜貫通領域は10本のαヘリックス(M1〜M10)から成る(図4)。細胞質領域は3つの明瞭に分離したドメインよりなり、その役割からA(actuator或いはanchor)、N(nucleotide binding)、P(phosphorylation)ドメインと命名した。NドメインにはATPの類似体のアデノシン部分が結合することを直接示した。また、Pドメインにはリン酸化部位(Asp351)が存在する。Aの役割については決定的なことはまだ言えない。
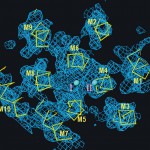
図4 Ca2+ -ATPaseの膜内部分を細胞質側からみたところ。M5をほぼ見通す向きから見ている。網目は溶媒平滑化を行った電子密度を、水色の2つの球は結合したカルシウムを示す。10本の膜貫通ヘリックスから成るが、M1〜M6とM7〜M10は明瞭に分離している。
③2個のカルシウムは横に並んで膜の中央より細胞質側に結合している。これ迄はイオン通路にそって縦に並んでいると考えられていた。
④2個のカルシウム結合は協同的であるが、それに対応して2個の結合サイトの構造は相当異なっている(図5)。
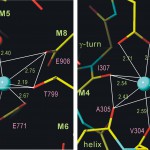
図5 Ca2+ -ATPaseのカルシウム結合部位の詳細。カルシウムの配位に寄与できる酸素原子はカルシウムからの距離が2.7Å程度まで。この原子間距離から価電子数(valence)を計算できる。配位の形はサイトⅠの方が歪みが大きく、価電子数も小さい。
⑤カルシウムを高い親和性を持って結合するために、膜貫通ヘリックスのうち2本(M4,M6)は結合部位付近でほどけている。
⑥カルシウム非存在下、デカバナジン酸存在下ではチューブ状結晶が得られるが、その電子顕微鏡による低分解能の三次元像[2][2]P. Zhang, C. Toyoshima, K. Yonekura, N. M. Green, and D. L. Stokes, : Nature 392 (1998) 835-839.と比較した結果、3つの細胞質側領域、特にAドメインは〜90°回転するという非常に大きなドメイン運動を示した(図6)。
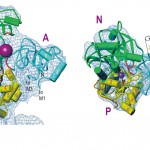
図6 Ca2+ -ATPaseのカルシウム非存在時の細胞質側ドメインの構造。チューブ状結晶から得た密度図(クーロンポテンシャルのマップ、水色の網目)にA,N,P3つのドメインが最も適合するように配置したもの。M4,M5はうまくマップと合わないが、M6とM7をつなぐループ(L67)は良く合うことに注意。赤紫色の大きな球はデカバナジン酸を示す。中抜きの2つの円筒はAドメインがPドメインに対し相対的に移動しないと仮定したときの2つのヘリックスの位置を示す。Nドメインは約20°Pドメイン側に傾斜している。bはaを上から見たところ。
膜貫通領域の構造
構造の全体にわたって詳しく述べている余裕は無いので、ここでは特にカルシウム結合部位の構造について述べよう。上に述べたように、Ca2+ -ATPaseの膜貫通ヘリックスはアミノ酸配列からの予測どおり10本であった。その配置は図4のようであり、M1〜M6とM7〜M10は別個のグループをつくっている。このことはバクテリア型のATPaseにはM7〜M10が欠けているものがあることに対応していると考えられる。
2つのカルシウム結合サイトはM5、M6、M8の側鎖だけで作られるサイトⅠと、ほとんどM4ヘリックスの上に出来ているサイトⅡとで構成される(図4)。2つのカルシウム結合部位の構造は相当に異なっている(図5)。サイトⅠはすべてアミノ酸残基の側鎖の酸素原子5個から成り、水も少なくとも1分子が配位している(図5)。配位する原子の位置は理想的な形からは遠く、原子間距離から計算した価電子数(valence)も水分子を入れても1.9とやや小さい(理想的には2.0である)[5][5]M. Nayal, and E. D. Cera : Proc. Natl. Acad. Sci. USA 91 (1994) 817-821.。サイトⅡの方では主鎖のカルボニル基の酵素原子3つが配位しており、Ca2+ 結合モチーフとして有名なEFハンドの変形と見なすことができ、Glu309が上からカルシウムに蓋をするような形になっている(図5)。配位の形も良く、安定な配位が実現されている。ここはPEGLというアミノ酸配列であり、プロリンがあるから、ヘリックスがほどけていることは十分考えられた。しかし、ほどけているとは言っても、turn構造が出来ており、ふらふらしているわけではない。いずれにしても、構造予測の際にはα−ヘリックスがほどけている可能性をも考えなければならないことが明らかになったわけで、その意義は大きいと思われる。また、M6もほどけていることがわかったが、2つのカルシウムに配位するAsp800の周りに限られている。
さて、サイトⅠに関与する残基とサイトⅡに関与する残基は違った性質を持つことが部位特異的変異の結果から得られている。すなわちサイトⅠに加えられた変異はカルシウムの結合を完全に無くしてしまうのに対し、サイトⅡでの変異は半分にするに過ぎない。サイトⅡに関与する残基2つに同時に変異を導入してもやはり半分であるから、サイトⅠへの結合は保たれるものと考えられる[3][3]Z. Zhang, D. Lewis, C. Strock, G. Inesi, M. Nakasako, H. Nomura and C. Toyoshima, : Biochemistry 39 (2000) 8758-8767.。つまりサイトⅠへのカルシウムの結合がサイトⅡへの結合を誘導することになる。これはよく知られたカルシウム結合の協同性を説明するものと考えられる。サイトⅡへの結合が、コンフォメーション変化を引き起こし、ATPの加水分解が始まる(それ以外のスイッチは無い)のだが、カルシウムの結合に伴ってドメインNの運動(熱運動)が誘起されるという報告もあり、ATPによるリン酸化がPドメインにあるAsp351で起こりえるようになる(ドメインNに結合したATPがAsp351に接近できるようになる)ということがその実態であるらしい。
細胞質側領域の構造変化
細胞質側領域が3つの非常に良く分離した領域(A,N,Pと名付けた)から成ることは既に述べた。Pドメインにはリン酸化されるアミノ酸残基であるAsp351がある。一方、ATPの類似物であり高い親和性を持つTNPAMPを結合させた結晶の構造を差フーリエ法で調べたところ、リン酸化部位とは大きく離れたNドメインに結合することが判明した。つまり、リン酸化が起こるためにはNドメインは大きく動き、Pドメインに近づかなければならないはずである。このことは電子顕微鏡による解析で得られたチューブ状結晶中のCa2+ -ATPaseの8Å分解能の密度図[2][2]P. Zhang, C. Toyoshima, K. Yonekura, N. M. Green, and D. L. Stokes, : Nature 392 (1998) 835-839.(厳密にはクーロンポテンシャルのマップ)と比較することで確かめられた。このチューブ状結晶はカルシウム非存在下、デカバナジン酸存在下で形成されるものである。蛋白質分解酵素による実験結果からはリン酸化状態(E2Pと呼ばれる状態)に近い状態にあると考えられる。この結果は図6に示したとおりであり、Nドメインは20°ほど傾きが変わっており、Pドメインに近くなっている。これでもまだATPがリン酸化残基に届くまでには足りないのであるが、バナジン酸の10量体であるデカバナジン酸(V10O286−、図6の赤い球)がNドメインとPドメインの間に挟まった形になって、もっと傾くのを妨害しているように見える。この部分には実際デカバナジン酸と対になる塩基性のアミノ酸残基がちょうど集まっている。驚くべきなのはAドメインであり、90°近くもほぼ水平に回転している。この領域の役割はほとんど判っておらず、唯一、TGESというP型ATPaseの目印の一つである配列があって、ATPase活性に重要であることが判っていたにすぎない。この配列は図6ではPドメインにある他の重要な配列(620TGD)のごく近傍に来ている。文献を調べてみて驚いたことには、鉄と過酸化水素で触媒される化学的切断の実験からこの配列の接近と離脱が示されていたのである[4][4]R. Goldshleger, S. J. D. Karlish : Proc. Natl. Acad. Sci. USA 94 (1997) 9596-9601.。
さらに興味深いことは、このように細胞質側の3つの領域をチューブ状結晶の密度図にあわせてみるとM4,M5というカルシウム結合サイトを形成している大事なαヘリックスはそのままではマップと合わないこと、M6,M7を結ぶループはPドメインの運動と連動して膜面からの高さが変わっていることである。実際、細胞質側領域だけではなく分子全体のモデリングを行ってみると細胞質側ドメインの運動をM5,M6ヘリックスの運動に変換する機構が備わっていることが判る。一方、M7〜M10は動かないようである。M5,M6がM8に対して動けば、サイトⅠに結合していたカルシウムははずれるであろう。そうすると協同性からサイトⅡに結合したカルシウムもはずれることになり、カルシウムの運搬が起こることになるのではないか。現在、カルシウムなしの状態の結晶化も順調に進んでいるから、近い将来に能動輸送の分子機構は構造から判ってしまうのではないかと期待している。
終わりに
SPring-8での実験はこのような研究のためには欠かせないものである。しかし、実験条件はとても最適化できているとはいえない。我々はこれまで使ったことのない強度のX線を相手にしているのであり、低温で実験しているとはいっても、照射損傷は明らかに起こっている。これまでは得たデータをその場で処理できないため実験条件に直ちに反映出来ないという重大な問題点があった。ようやく最近になってBL40B2付近では使い慣れたDenzo等によって、得たデータを直ちに評価できるようになった。大変喜ばしいこと(こうでなくては!)と思う。なお、SPring-8での回折実験では理研の足立伸一博士、神谷信夫博士、JASRIの河本正秀博士の協力を頂いた。深く感謝するものである。
参考文献
[1]C.Toyoshima,M.Nakasako,H.Nomura,and H.Ogawa : Nature 405(2000)647-655.
[2]P.Zhang,C.Toyoshima,K.Yonekura,N.M.Green,and D.L.Stokes, : Nature 392(1998)835-839.
[3]Z.Zhang,D.Lewis,C.Strock,G.Inesi,M.Nakasako,H.Nomura and C.Toyoshima, : Biochemistry 39(2000)8758-8767.
[4]R.Goldshleger,S.J.D.Karlish : Proc.Natl.Acad.Sci.USA 94(1997)9596-9601.
[5]M.Nayal,and E.D.Cera : Proc.Natl.Acad.Sci.USA 91(1994)817-821.
豊島 近 TOYOSHIMA Chikashi
東京大学 分子細胞生物学研究所 教授
〒113-0032 東京都文京区弥生1-1-1
TEL:03-5841-8492 FAX:03-5841-8491
e-mail:ct@iam.u-tokyo.ac.jp
中迫 雅由 NAKASAKO Masayoshi
東京大学 分子細胞生物学研究所 講師
〒113-0032 東京都文京区弥生1-1-1
TEL:03-5841-7895 FAX:03-5841-8493
e-mail:nakasako@iam.u-tokyo.ac.jp
野村 博美 NOMURA Hiromi
東京大学 分子細胞生物学研究所 技官
〒113-0032 東京都文京区弥生1-1-1
TEL:03-5841-8492 FAX:03-5841-8491
e-mail:hiron@iam.u-tokyo.ac.jp
小川 治夫 OGAWA Haruo
東京大学 分子細胞生物学研究所 助手
〒113-0032 東京都文京区弥生1-1-1
TEL:03-5841-7816 FAX:03-5841-8491
e-mail:haru@iam.u-tokyo.ac.jp








