Volume 16, No.2 Pages 76 - 80
1. 最近の研究から/FROM LATEST RESEARCH
小胞体内在性ジスルフィド結合還元酵素ERdj5によって促進される小胞体関連分解経路の構造的基盤
Structural basis of an ERAD pathway mediated by the ER-resident protein disulfide reductase ERdj5
九州大学 生体防御医学研究所 Medical Institute of Bioregulation, Kyushu University
- Abstract
- 小胞体は分泌タンパク質および膜タンパク質が合成される重要な細胞内小器官である。小胞体内で生じた構造異常タンパク質はサイトゾルに逆行輸送され、ユビキチン-プロテアソーム系により分解される。この機構は小胞体関連分解と呼ばれ、小胞体におけるタンパク質品質管理において必須の役割を担う。ERdj5は小胞体内在性の酸化還元酵素であり、誤ったシステイン間で形成されたジスルフィド結合を還元することで異常タンパク質の小胞体関連分解を促進する。本研究において我々は、SPring-8 BL44XUを用いた放射光実験により、ERdj5全長の高分解能結晶構造を解くことに成功した。さらに構造情報を基にした系統的な機能解析により、ERdj5が促進する構造異常タンパク質の小胞体関連分解経路の分子基盤を確立した。
はじめに
小胞体(Endoplasmic Reticulum、略してER)は分泌タンパク質および膜タンパク質の合成の場である。そこには、正しく立体構造形成(フォールド)したタンパク質のみを選択的に分泌させ、異常な構造をとった(ミスフォールドした)タンパク質は分解除去されるという品質管理システムが存在する。小胞体内で合成された新生ポリペプチド鎖は、幾つかの分子シャペロンや酸化還元酵素の助けを借りて正しくフォールドし、分泌経路に沿って所定の小器官へと運ばれる。しかし一方で、遺伝的変異によりフォールディングできないものや各種ストレスによって変性したタンパク質は、小胞体からサイトゾルへ送り返され、ユビキチン−プロテアソーム系により分解される。この一連の過程は小胞体関連分解(ER-associated degradation、略してERAD)と呼ばれる。これら小胞体品質管理システム(図1)の破綻はミスフォールド蛋白質の過剰な蓄積につながり、アルツハイマー病やパーキンソン病などの神経変性疾患を誘起することが報告されている。
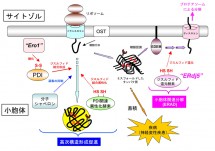
図1 小胞体におけるタンパク質品質管理機構
異常タンパク質の中には、誤った分子間ジスルフィド結合により多量体を形成し、そのままの状態では分解されにくいものも存在する。ERdj5は、小胞体で最初に発見されたジスルフィド結合還元酵素であり、誤ったジスルフィド結合を開裂することで異常凝集タンパク質の立体構造を解きほぐし、膜透過さらには分解されやすい形に変換し、小胞体関連分解を促進する[1][1] R. Ushioda, J. Hoseki, K. Araki, G. Jansen, D. Y. Thomas and K. Nagata: Science 321 (2008) 569-72.。ERdj5の結晶構造解析について、SPring-8 BL44XUで2008年より研究を行い(課題番号:2008A&B6803;2009A&B6905;2010A&B6505)、2011年2月にMolecular Cell誌にその成果を発表するに至った[2][2] M. Hagiwara, K. Maegawa, M. Suzuki, R. Ushioda, K. Araki, Y. Matsumoto, J. Hoseki, K. Nagata and K. Inaba: Mol. Cell. 41 (2011) 432-444.。本稿では、我々が解いたERdj5全長の結晶構造と、ERdj5が駆動する小胞体関連分解経路の作用機序を解説する。
1.ERADを促進するジスルフィド還元酵素ERdj5の発見
小胞体で成熟するタンパク質の多くは、その翻訳途上でAsn-X-Ser/Thr配列のアスパラギン残基に糖鎖(N結合型糖鎖)が付加される。そののちトリミングされた糖鎖はレクチン様分子シャペロンであるカルネキシン(CNX)やカルレティキュリン(CRT)に認識され、正しい立体構造形成が促される。CNX/CRTの作用によっても正しくフォールドできない糖タンパク質はEDEM1と呼ばれる因子によって認識され、ERADが促進されると報告されている[3,4][3] Y. Oda, N. Hosokawa, I. Wada and K. Nagata: Science 299 (2003) 1394-1397.
[4] M. Molinari, V. Calanca, C. Galli, P. Lucca and P. Paganetti: Science 299 (2003) 1397-1400.。
永田和宏教授(京都産業大学)らの研究グループは、EDEM1と相互作用する分子のスクリーニングを行い、その結果ERdj5を同定した[1][1] R. Ushioda, J. Hoseki, K. Araki, G. Jansen, D. Y. Thomas and K. Nagata: Science 321 (2008) 569-72.。ERdj5は、そのアミノ酸配列からHSP(Heat Shock Protein)-40ファミリータンパク質に共通するDnaJドメインとPDI(Protein Disulfide Isomerase)ファミリータンパク質に共通する4つのチオレドキシン(Trx)様ドメインをもつことが予想された。HSP40ファミリータンパク質はDnaJドメインを介してHSP70ファミリータンパク質に結合し、協同的に働く。したがってERdj5は、小胞体内在性のHSP70ファミリータンパク質であるBiP(Immunoglobulin heavy chain-binding protein)と協調しながらタンパク質の品質管理やストレス応答において重要な役割を担うと考えられる。
哺乳類細胞の小胞体には、ERdj5を含め、チオレドキシン様ドメインを持つPDIファミリータンパク質が約20種類知られている。多くのチオレドキシン様ドメインは活性中心としてCys-Xaa-Xaa-Cys(CXXC)モチーフをもち、ジスルフィド結合の形成、異性化および還元反応を司る。興味深いことに、ERdj5を過剰発現させた哺乳動物細胞では、ジスルフィド結合を有する幾つかの基質タンパク質の分解促進が観測された。この分解促進効果はジスルフィド結合依存的な多量体形成の抑制によると考えられ、システインを含まない基質のERADには促進効果はみられなかった。また試験管中での精製ERdj5を用いた実験においても、ERdj5が強いジスルフィド結合還元活性を示し、また酸化還元電位の測定から本酵素の活性部位が強い還元的性質を有することが示された。
以上のことから、ERdj5はミスフォールドタンパク質のジスルフィド結合を速やかに還元し、これにより構造を解きほぐすことで逆輸送チャネルを透過しやすい形に変換し、結果としてERADが促進されるという考えが提唱された[1][1] R. Ushioda, J. Hoseki, K. Araki, G. Jansen, D. Y. Thomas and K. Nagata: Science 321 (2008) 569-72.。
2.ERdj5の結晶構造
ERdj5が促進するERAD経路の作用機序を解明する目的で、ERdj5のX線結晶構造解析に取り組んだ。ERdj5は分子量が90 kDaにもおよぶPDIファミリー最大のマルチドメインタンパク質であり、またその低い発現効率および高い凝集性のため結晶化スクリーニングには大きな困難を伴った。しかしながら、徹底したサンプル調整条件および結晶化条件の検討により、構造解析可能な良質の結晶を作製することに成功した。ビームラインとして、これまで生体超分子複合体の結晶構造解析で数多くの成果をあげているSPring-8 BL44XUを用い、最終的にERdj5全長の結晶構造を2.4 Å分解能で解くことに成功した。その結果、当初予想した4つのTrx様ドメイン(Trx1〜Trx4)に加え、Trx1とTrx2の間にCXXCモチーフをもたないTrx様ドメインが2つ(Trxb1、Trxb2)存在することが明らかとなった。これら計6つのTrx様ドメインは同一平面上に存在し、Trx2とTrx3のリンカーループ領域を境に、Trx1、Trxb1、Trxb2、Trx2ドメインからなるN末端側クラスターとTrx3、Trx4ドメインからなるC末端側クラスターに分断できることが判明した(図2a)。BiPとの結合能を有するDnaJドメインのみがこの平面から外れ、Trx1とTrx2の境界の上に位置していた。
ERdj5はPDIファミリーに属するものの、その全体構造はPDIと大きく異なる。PDIはU字型に並ぶ4つのTrx様ドメインから成り、両端に位置するCXXCモチーフは互いに向き合っている。またU字の中央部には、基質との結合に関わるとされる大きな疎水性ポケットが存在する[5][5] G. Tian, S. Xiang, R. Noiva, W. J. Lennarz and H. Schidelin: Cell 124 (2006) 61-73.(図2b)。一方ERdj5の活性部位は互いに向き合うことはなく、特にC末端側クラスターに存在するTrx3とTrx4のCXXCモチーフは分子の外側を向く(図2a)。また分子の中央部に位置するクレフト領域はPDIとは対照的に疎水性に富んでおらず、この領域が基質との結合に関わるとは考えにくい。これら構造上の相違点から、ERdj5とPDIが全く異なる分子機構で独自の機能を発揮していることは容易に想像がつく。
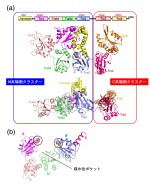
図2 ERdj5(a)とPDI(b)の結晶構造。レドックス活性部位(CXXCチーフ)は黒丸で表す。
3.ERdj5の各ドメインの機能的役割
ERdj5の構造情報をもとに、各ドメインの機能的役割を系統的に解析した。まずERdj5の主たる還元活性ドメインを同定するため、活性中心であるCXXCモチーフを不活性型のAXXAに置換した種々の変異体を作製し、パルスチェイス法によりそのERAD速度を観測したところ、Trx1、Trx2の活性部位をつぶした変異体は野生型と同様にERAD促進活性を示したのに対し、Trx3、Trx4の活性部位をつぶした変異体はERAD促進活性を大きく失った。このことは、C末端クラスターに含まれるTrx3、Trx4がERADを促進するのに必須な還元活性ドメインであることを示唆する。
次に精製ERdj5変異体を用い、試験管中での還元活性測定および酸化還元電位測定を行った。その結果、Trx1、Trx3およびTrx4ドメインがインスリンに対する強い還元活性を有することが判明した。Trx2ドメインに還元活性が観測されなかったことは、Trx2のCXXCモチーフがDnaJドメインに覆い隠されていることから説明可能である。一方各ドメインの酸化還元電位の測定の結果、ERADにおける主たる還元ドメインであるTrx3、Trx4が強い還元的性質を有することも示された。
次にEDEM1がERdj5のどの部位に結合するのか、系統的な共免疫沈降実験により調べた。ERdj5のN末端側クラスターのみあるいはC末端側クラスターのみをEDEM1と共にヒト細胞中で発現し、ERdj5抗体で免疫沈降を行ったところ、C末端側クラスターに特異的にEDEM1が検出された。この系にERAD基質も共発現したところ、ERdj5 C末端側クラスター・EDEM1・基質から成る三者複合体の形成が確認できた。このことは、ERAD基質をリクルートしたEDEM1がERdj5のC末端側クラスターに選択的に結合し、基質をERdj5のこのクラスターに提示することを強く示唆している。この知見は、ERAD基質が主としてERdj5のC末端側クラスターに含まれるTrx3、Trx4によって還元されるという先の見解とも合致する。
4.ERdj5が促進するERAD経路に沿った基質の動的挙動
以上得られた知見を統合し、カルネキシンから解離したERAD基質はまずEDEM1に認識・捕獲され、次にERdj5 C末端側クラスターへとリクルートされることで還元を受ける。その結果構造が解きほぐされた基質はERdj5のDnaJドメインに結合したBiPに捕獲され、ATPの加水分解に伴いBiPはERdj5から解離し、逆輸送チャネルへ基質を運ぶという一連のイベントが予想された。このことをさらに確認するため、ERAD基質をパルスラベルし、その後基質がどの因子と結合するか免疫沈降法により詳細に解析した。その結果予想したように、基質はまずカルネキシンと結合し、続いてカルネキシンからEDEM1、ERdj5への基質の受け渡しが観測された。さらにこのあと基質がERdj5からBiPへ移る過程も同実験により観測でき、ERdj5を介したERAD経路上の基質の動的挙動を解明するに至った。以上の実験により、我々が提唱するERdj5によって促進されるERAD経路モデル(図3)の妥当性が強く示された。
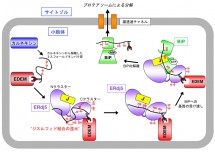
図3 ERdj5が促進する小胞体関連分解機構モデル
5.おわりに
以上の構造生物学的知見およびそれに基づく機能解析の結果、ERdj5に依存した一連のERAD経路の分子基盤を確立するに至った。しかしながら、これはあくまでも複数存在するはずのERAD経路の一つを明らかにしたに過ぎない。最近ERADに関わるとされるレクチン因子やレドックス因子が次々と報告され、様々な基質のERAD経路を網羅的に解析することが今後の最重要課題であろう。
さらに、ERdj5に還元力を供給する因子が何なのかという問題も含め、本来は酸化的である小胞体中のジスルフィド還元カスケードを同定することも興味深い残された課題である。小胞体におけるタンパク質品質管理およびレドックス恒常性維持のために張り巡らされた酸化還元経路は依然ほとんど未解明であり、今後の新規開拓分野であることは間違いない。本研究を遂行することで、将来的には異常タンパク質の蓄積や細胞内レドックス制御の破綻に起因する種々の疾病の成因解明および治療開発につながれば、望外の喜びである。
謝辞
本研究はSPring-8利用申請課題 2008A6803、2008B6803、2009A6905、2009B6905、2010A6505、2010B6505で行われたものです。BL44XUのビームライン管理者である中川敦史先生、鈴木守先生、山下栄樹先生に厚く御礼申し上げます。また本研究は京都産業大学・永田和宏教授の研究グループと共同で行ったものである。
参考文献
[1] R. Ushioda, J. Hoseki, K. Araki, G. Jansen, D. Y. Thomas and K. Nagata: Science 321 (2008) 569-572.
[2] M. Hagiwara, K. Maegawa, M. Suzuki, R. Ushioda, K. Araki, Y. Matsumoto, J. Hoseki, K. Nagata and K. Inaba: Mol. Cell. 41 (2011) 432-444.
[3] Y. Oda, N. Hosokawa, I. Wada and K. Nagata: Science 299 (2003) 1394-1397.
[4] M. Molinari, V. Calanca, C. Galli, P. Lucca and P. Paganetti: Science 299 (2003) 1397-1400.
[5] G. Tian, S. Xiang, R. Noiva, W. J. Lennarz and H. Schidelin: Cell 124 (2006) 61-73.
稲葉 謙次 INABA Kenji
九州大学 生体防御医学研究所 蛋白質化学分野
〒812-8582 福岡県福岡市東区馬出3-1-1
TEL:092-642-6433
e-mail:inaba-k@bioreg.kyushu-u.ac.jp








