Volume 27, No.3 Pages 193 - 196
1. 最近の研究から/FROM LATEST RESEARCH
長期利用課題報告1
ゲノム編集ツールCRISPR-Casヌクレアーゼの構造解析
Structural analysis of CRISPR-Cas nucleases
東京大学大学院 理学系研究科 Graduate School of Science, The University of Tokyo
- Abstract
- 原核生物のもつCRISPR-Cas機構に由来する様々なCasタンパク質は、ガイドRNAと相補的な核酸を特異的に切断する。この性質を利用したものとしてCas9の2本鎖DNA切断によるゲノム編集が挙げられるが、近年ではCas13によるコロナウイルスの高感度・高速検出技術が開発されるなど[1][1] H. Shinoda et al.: Commun. Biol. 4 (2021) 476.、その応用範囲を拡大している。本長期課題では応用的に重要視されているCRISPR-Cas酵素の結晶構造を決定し、そのRNA依存性核酸切断機構を明らかにすることに成功した。
1. CRISPR-Cas機構
CRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR associated)機構は原核生物のもつ獲得免疫機構であり、ファージやプラスミドなどに由来する外来核酸に対する防御を行う[2][2] R. Barrangou et al.: Science 315 (2007) 1709-1712.。CRISPR-Cas機構はCasタンパク質とcrRNA(CRISPR RNA)から構成される。Casタンパク質はcrRNAと複合体を形成し、crRNAのガイド配列と相補的な外来核酸を認識し、切断する。CRISPR-Cas機構は哺乳類細胞内でも機能することから、任意の配列の核酸を標的可能なゲノム編集ツールとして注目されている。CRISPR-Cas機構はそれを構成する遺伝子の種類などによりさまざまなタイプに分類されるが、その中でも特に単一のタンパク質が標的核酸の切断を担うことから、クラス2に分類されるII型、V型、VI型のCRISPR-Cas機構がゲノム編集ツールとして利用されている[3,4][3] K. S. Makarova et al.: Nat. Rev. Microbiol. 13 (2015) 722-736.
[4] H. Nishimasu and O. Nureki: Curr. Opin. Struct. Biol. 43 (2017) 68-78.。
ゲノム編集に広く利用されているのはII型のCRISPR-Cas機構で機能する、Cas9とよばれているCasタンパク質である。Cas9はcrRNAに加えてtracrRNAとよばれるRNAと複合体を形成し、crRNAのガイド配列と相補的な2本鎖DNAを切断する[5,6][5] G. Gasiunas, R. Barrangou, P. Horvath and V. Siksnys: Proc. Natl. Acad. Sci. U.S.A. 109 (2012) E2579-2586.
[6] M. Jinek et al.: Science 337 (2012) 816-821.(図1)。crRNAとtracrRNAを人工的に接続したsgRNAも同様の機能をもつことが知られている。Cas9によるDNAの認識にはガイド配列と標的DNAの相補性に加え、その近傍にPAM(protospacer adjacent motif)とよばれる特定の配列が必要である。また、Cas9は分子系統的にII-A型、II-B型、II-C型に分類され、さらにその中においても配列相動性が低く、由来する生物種によって複合体を形成するRNAの配列、認識するPAM配列、分子サイズなどが多様であることが知られている[7][7] K. Chylinski et al.: Nucleic Acids Res. 42 (10) (2014) 6091-105.。本研究グループはこれまでに様々な生物種に由来するCas9の結晶構造解析を行うことにより、その基礎的な分子メカニズムを解明してきた。まず、ゲノム編集ツールとして最もよく利用されているII-A型のStreptococcus pyogenes由来Cas9(SpCas9)に着目し、SpCas9-sgRNA-DNA複合体の結晶構造を世界にさきがけて決定した[8][8] H. Nishimasu et al.: Cell 156 (2014) 935-949.。次いで、II-A型のStaphylococcus aureus由来Cas9(SaCas9)、II-B型のFrancisella novicida由来Cas9(FnCas9)、II-C型のCampylobacter jejuni由来Cas9(CjCas9)のsgRNAおよびDNAとの複合体の結晶構造解析に成功した[9-11][9] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.
[10] H. Hirano et al.: Cell 164 (2016) 950-961.
[11] M. Yamada et al.: Mol. Cell 65 (2017) 1109-1121.。これら一連の構造解析により、Cas9によるRNA依存性DNA切断機構のみならず、多様なPAM認識機構およびRNA認識機構などを明らかにしてきた。
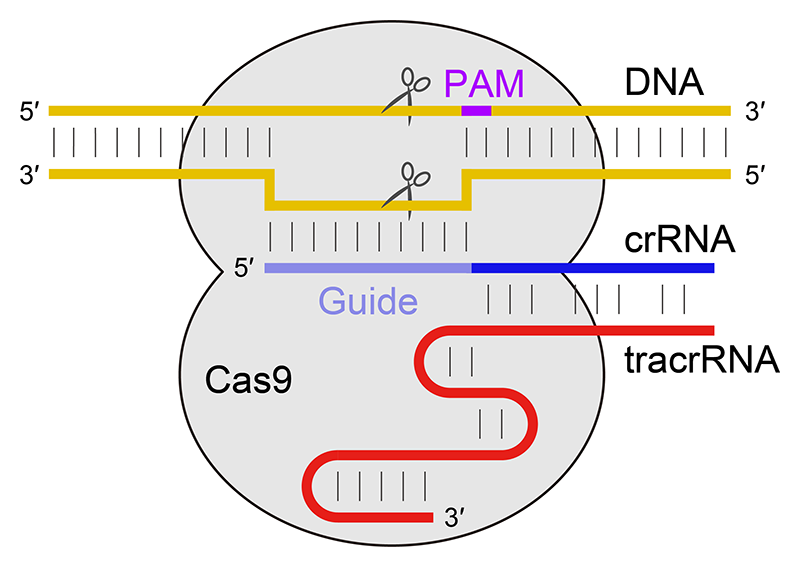
図1 Cas9による標的DNA切断機構
本長期課題では、新規ゲノム編集ツールとして期待されているCasタンパク質に焦点を当てて研究を推進した。CRISPR-Cas機構を利用したゲノム編集の問題点として、PAM配列の必要性による標的可能な配列の制限と、分子サイズが大きいことによる細胞内への導入効率の低さが挙げられる。例として、ゲノム編集に広く利用されているSpCas9は5′-NGG-3′(Nは任意の塩基)という配列のみを認識し、1368アミノ酸からなる比較的大きいタンパク質である。本研究ではそれらの課題を部分的に解決するものとして期待されているSpCas9改変体(SpCas9-NG)、Brevibacillus laterosporus由来Cas9(BlCas9)およびCorynebacterium diphtheriae由来Cas9(CdCas9)、加えて、Cas9と異なり配列依存的に1本鎖RNAと結合するVI型のCasタンパク質であるCas13bの結晶構造解析を行った。
2. SpCas9-NGの結晶構造解析
SpCas9の結晶構造から5′-NGG-3′の2塩基目および3塩基目のGはそれぞれArg1333およびArg1335により認識され、PAMを含む2本鎖DNAはPI(PAM-interacting)ドメインによって形成される正電荷を帯びた溝に結合することが明らかにされていた[12][12] M. Jinek et al.: Nature 513 (2014) 569-573.。そこで、我々はこのArg1335を変異させG塩基との塩基特異的な相互作用を無くすと同時に、2本鎖DNAの糖-リン酸骨格へ相互作用する変異をPIドメインに導入することにより、5′-NG-3′をPAMとして認識するSpCas9が得られると考えた。この仮説に基づき様々な変異体を設計し、DNA切断活性を測定したところ、Arg1335Val置換、Leu1111Arg置換、Asp1135Val置換、Gly1218Arg置換、Glu1219Phe置換、Ala1322Arg置換、Thr1337Arg置換の7つの変異をもつSpCas9は効率よく5′-TGN-3′ PAMをもつDNAを切断することが明らかとなった[13][13] H. Nishimasu et al.: Science 361 (2018) 1259-1262.。さらに、この7つの変異をもつSpCas9を用いて、65,536(4の8乗)通りの配列をもつDNAライブラリーをin vitroにおいて切断し次世代シークエンサーを用いて解析した結果、僅かな嗜好性があるものの5′-NG-3′ PAMを認識することが確かめられ、これをSpCas9-NGと命名した。SpCas9-NGによるPAMの認識機構を解明するために、SpCas9-NG、sgRNAおよび標的DNAからなる複合体を結晶化し、BL41XUにおいてX線回折データを収集し、SpCas9-sgRNA-標的DNA複合体の結晶構造をサーチモデルとした分子置換法により結晶構造を決定した。結晶構造から、予想通りArg1335Valの置換によりPAMの3番目のG塩基との塩基特異的な相互作用は失われ、Val1135およびPhe1219は糖骨格と疎水性相互作用を、Arg1111はリン酸骨格と静電相互作用をしていることが明らかになった(図2)。その一方でArg1218とArg1337は予想とは異なり、Arg1218は周辺のアミノ酸の主鎖と水素結合を形成していた一方で、Arg1337は結晶化に用いたDNAの塩基に特異的な水素結合を形成していた。このことから、Arg1218はPIドメインの構造安定化に寄与し、Arg1337はSpCas9-NGのPAM配列の嗜好性に関与していることが示唆された。
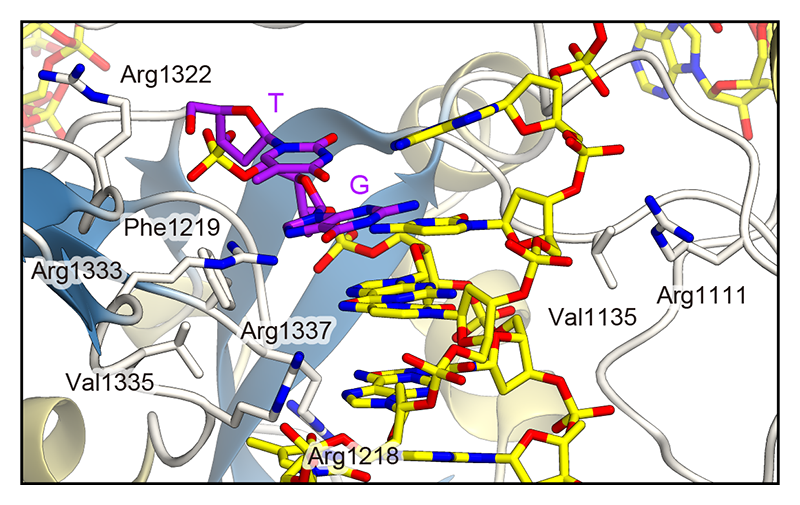
図2 SpCas9-NGによるPAMを含む2本鎖DNA認識機構
3. 小型Cas9の結晶構造解析
B.lacterosporus由来Cas9(BlCas9)およびC.diphtheriae由来Cas9はII-C型に属するCas9で小型(約1000アミノ酸残基)である[14,15][14] T. Karvelis et al.: Genome Biol. 16 (2015) 253.
[15] F. A. Ran et al.: Nature 520 (2015) 186-191.。BlCas9は5′-NNNNC-3′とC塩基1文字を認識するのに対し、CdCas9は5′-NNRHHHY-3′(R=A/G、H=A/C/T、Y=C/T)と緩い特異性で長いPAMを認識する。これらのCas9によるRNA依存性DNA切断機構を解明するために、BlCas9およびCdCas9のsgRNAおよび標的DNAを含む複合体をそれぞれ結晶化し、BL41XUにおいてX線回折データを収集し、SeMet置換体を用いたSAD法により結晶構造を決定した[16][16] S. Hirano et al.: Nature Commun 10 (2019) 1968.(図3)。BlCas9およびCdCas9は既存の小型のCas9と全体構造は類似していたものの、異なる配列をもつRNAを特異的に認識し複合体を形成していることが明らかになった。また、構造から各々の特異的なPAM認識機構が明らかとなった。BlCas9はAsp1023とLys1041がそれぞれC塩基とG塩基と水素結合を形成することにより強固にG:C塩基対を認識しており、これにより1塩基のみのPAM認識を可能にしていることが示唆された。一方で、CdCas9のPIドメインにはPhe1011、Lys1015、Pro1043、Leu1046などから形成される疎水性パッチが存在し、これがPAMを含む2本鎖DNAの主溝に接近していた。この疎水性パッチによるファンデルワールス相互作用によって、緩い特異性のPAM認識が可能になっていることが示唆された。さらに、SpCas9-NGと同様の戦略によりBlCas9の結晶構造を基にしてPIドメインに変異を入れ、異なる塩基配列をPAMとして認識する改変型BlCas9の開発に成功した。
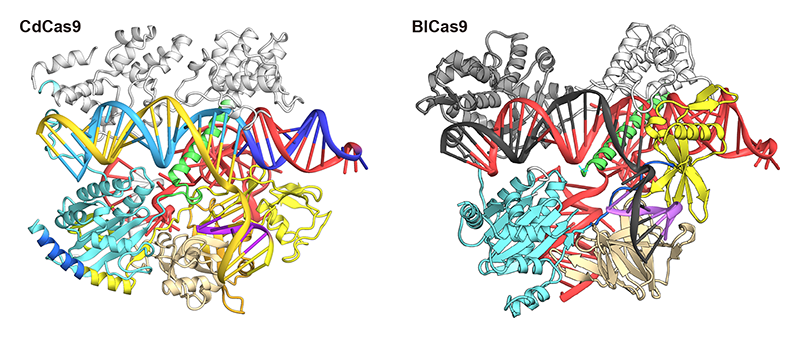
図3 CdCas9およびBlCas9の全体構造
4. Cas13btの結晶構造
近年、ゲノムデータベースの拡大によって様々な新規Casタンパク質が発見されており、その中でもCas13b(Cas13bt3)(775残基)は小型でcrRNA依存的に1本鎖RNAを切断する機能があることで注目されていた[17][17] S. Kannan et al.: Nat Biotechnol 40 (2) (2021) 194-197.。本研究では、Cas13bとcrRNAからなる複合体を結晶化し、BL41XUにおいてX線回折データを測定し、SeMet置換体を用いたSAD法により1.9 Å分解能で結晶構造を決定した(図4)。得られた構造から、Cas13bt3はREC(recognition)ローブとNUC(nuclease)ローブからなり、crRNAがRECローブにアンカーされていることが明らかとなった。RECローブはHelical-1ドメイン、Lidドメイン、Helical-2ドメインから構成され、NUCローブはHEPN1ドメイン、およびHEPN2ドメインから構成されていた。crRNAはガイド領域、2つのステム領域(Stem1、Stem2)、および2つのループ領域(internal loop, hairpin loop)から構成されており、Cas13bt3のRECローブによって認識されていた。特に、crRNAからフリップした3つの塩基、C(−8)、G(−28)、A(−32)はHelical-1ドメイン、Lidドメイン、Helical-2ドメインによって塩基特異的に認識されていた。Cas13bt3はすでに構造が報告されているPbuCas13bと比較して350塩基小さく、HEPN1ドメインを除いて配列相同性をもたないにも関わらず、全体構造はPbuCas13bと類似していた(RSMD:4.31 Å)。ドメインごとの比較から、Cas13bt3はすべてのドメインがPbuCas13bよりもそれぞれ30-100塩基ほど小さくなっていた。したがって、これらの違いがCas13bt3の小型化に貢献していることが明らかとなった。今回の結果は、CRISPR-Casシステムにおける共通性および多様性を明らかにするとともに、新たな革新的技術の開発基盤となることが期待される。本研究成果はMolecular Cell誌に掲載予定である。

図4 Cas13bt3の全体構造
参考文献
[1] H. Shinoda et al.: Commun. Biol. 4 (2021) 476.
[2] R. Barrangou et al.: Science 315 (2007) 1709-1712.
[3] K. S. Makarova et al.: Nat. Rev. Microbiol. 13 (2015) 722-736.
[4] H. Nishimasu and O. Nureki: Curr. Opin. Struct. Biol. 43 (2017) 68-78.
[5] G. Gasiunas, R. Barrangou, P. Horvath and V. Siksnys: Proc. Natl. Acad. Sci. U.S.A. 109 (2012) E2579-2586.
[6] M. Jinek et al.: Science 337 (2012) 816-821.
[7] K. Chylinski et al.: Nucleic Acids Res. 42 (10) (2014) 6091-105.
[8] H. Nishimasu et al.: Cell 156 (2014) 935-949.
[9] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.
[10] H. Hirano et al.: Cell 164 (2016) 950-961.
[11] M. Yamada et al.: Mol. Cell 65 (2017) 1109-1121.
[12] M. Jinek et al.: Nature 513 (2014) 569-573.
[13] H. Nishimasu et al.: Science 361 (2018) 1259-1262.
[14] T. Karvelis et al.: Genome Biol. 16 (2015) 253.
[15] F. A. Ran et al.: Nature 520 (2015) 186-191.
[16] S. Hirano et al.: Nature Commun 10 (2019) 1968.
[17] S. Kannan et al.: Nat Biotechnol 40 (2) (2021) 194-197.
東京大学大学院 理学系研究科
〒113-0033 東京都文京区本郷7-3-1
TEL : 03-5841-4391
e-mail : nureki@bs.s.u-tokyo.ac.jp








