Volume 23, No.3 Pages 210 - 214
1. 最近の研究から/FROM LATEST RESEARCH
長期利用課題報告2
ゲノム編集ツールCas9エンドヌクレアーゼのX線結晶構造
Crystallographic Analysis of CRISPR-Cas Genome Editor Nucleases
東京大学大学院 理学系研究科 Graduate School of Science, The University of Tokyo
- Abstract
- 原核生物のもつCRISPR-Cas獲得免疫機構に関与するRNA依存性DNAヌクレアーゼCas9はガイドRNAと相補的な2本鎖DNAを選択的に切断する性質をもつ。近年、Cas9を利用したゲノム編集技術は基礎研究から臨床応用にいたる幅広い分野において急速に普及した。本長期利用課題では異なる細菌に由来する多様なCRISPR-Cas酵素の結晶構造を決定し、そのRNA依存性DNA切断機構を原子レベルで明らかにすることに成功した。さらに、構造情報を基にした新規のゲノム編集ツールの開発にも成功した。
1. CRISPR-Cas系
原核生物は外来核酸に対する防御機構としてCRISPR-Cas(clustered regularly interspaced short palindromic repeat-CRISPR-associated)とよばれる獲得免疫機構をもつ[1][1] R. Barrangou et al.: Science 315 (2007) 1709-1712.。CRISPR-Cas系ではCas(CRISPR-associated)タンパク質とcrRNA(CRISPR RNA)が複合体を形成し、crRNAのガイド配列と相補的な外来核酸を認識・切断する。CRISPR-Cas系は2つのクラスに分類される[2,3][2] K. S. Makarova et al.: Nat Rev Microbiol 13 (2015) 722-736.
[3] H. Nishimasu and O. Nureki: Curr Opin Struct Biol 43 (2017) 68-78.。クラス1のCRISPR-Cas系には複数のCasタンパク質からなるCas-crRNA複合体が関与する。一方、クラス2のCRISPR-Cas系では単一のCasタンパク質が外来核酸を切断する。クラス2のCRISPR-Cas系はII型、V型、VI型に分類される。II型CRISPR-Cas系においてはRNA依存性ヌクレアーゼCas9がcrRNAおよびtracrRNA(trans-activating crRNA)と複合体を形成し、crRNAのガイド配列と相補的な2本鎖DNAを切断する[4,5][4] G. Gasiunas, R. Barrangou, P. Horvath and V. Siksnys: Proc Natl Acad Sci USA 109 (2012) E2579-2586.
[5] M. Jinek et al.: Science 337 (2012) 816-821.(図1)。Cas9は2つのヌクレアーゼドメイン(RuvCとHNH)をもち、HNHドメインはガイドRNAと相補的なDNA鎖(相補鎖)を切断する一方、RuvCドメインはもう一方のDNA鎖(非相補鎖)を切断する。Cas9によるDNA認識にはガイドRNAとの相補性に加え、PAM(protospacer adjacent motif)とよばれる特定の塩基配列が必要である。crRNAとtracrRNAを人工的に連結したsgRNA(single-guide RNA)も同様の機能をもち[5][5] M. Jinek et al.: Science 337 (2012) 816-821.、20塩基のガイド配列は自由に変更できるため、Cas9-sgRNA複合体を用いることにより、ゲノムDNAの狙った位置を特異的に切断することが可能である。したがって、2013年以降、Cas9は革新的なゲノム編集ツールとして広く普及した[6][6] L. Cong et al.: Science 339 (2013) 819-823.。
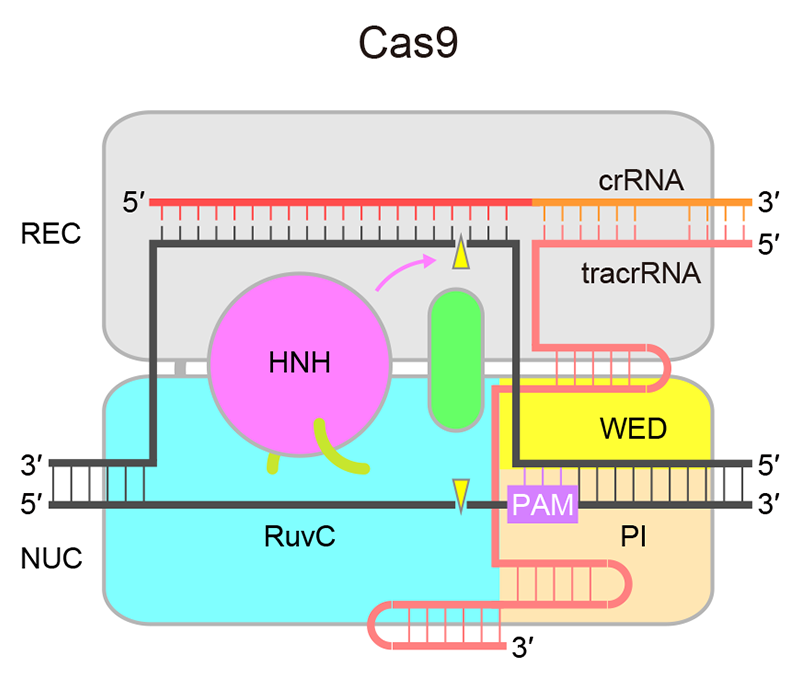
図1 Cas9によるDNA切断機構
2014年、本研究グループはゲノム編集に広く利用されているStreptococcus pyogenes由来Cas9(SpCas9)に着目し、SpCas9-sgRNA-DNA複合体の結晶構造を世界にさきがけて決定し、Cas9によるRNA依存性DNA切断の分子機構を明らかにした[7][7] H. Nishimasu et al.: Cell 156 (2014) 935-949.。さらに、他の研究グループにより報告されたSpCas9単体[8][8] M. Jinek et al.: Science 343 (2014) 1247997.、SpCas9-sgRNA複合体[9][9] F. Jiang, K. Zhou, L. Ma, S. Gressel and J. A. Doudna: Science 348 (2015) 1477-1481.、SpCas9-sgRNA-標的DNA複合体[10,11][10] C. Anders, O. Niewoehner, A. Duerst and M. Jinek: Nature 513 (2014) 569-573.
[11] F. Jiang et al.: Science 351 (2016) 867-871.の結晶構造から、Cas9によるDNA切断機構が明らかになってきた。しかし、異なる細菌に由来するCas9のアミノ酸配列の相同性は低く、ガイドRNAやPAMの塩基配列は大きく異なるため、Cas9の作動機構には不明な点が残されていた。SpCas9はNGGという配列をPAMとして認識する一方、Staphylococcus aureus由来Cas9(SaCas9)、Francisella novicida由来Cas9(FnCas9)、Campylobacter jejuni由来Cas9(CjCas9)はそれぞれNNGRRT(RはAまたはG)、NGG、NNNVRYAC(VはA、GまたはC、YはTまたはC)という配列をPAMとして認識する[12,13][12] F. A. Ran et al.: Nature 520 (2015) 186-191.
[13] E. Kim et al.: Nature Commun 8 (2017) 14500.。さらに、2015年にはV型CRISPR-Cas酵素であるCpf1(Cas12a)が発見された[14][14] B. Zetsche et al.: Cell 163 (2015) 759-771.。Cpf1はCas9と同様にRNA依存的に2本鎖DNAを切断するが、(1)tracrRNAを必要としない、(2)TTTVという配列をPAMとして認識する、(3)PAMから離れた位置でDNAを切断する、(4)HNHドメインをもたない、などの特徴をもつため、Acidaminococcus sp.由来Cpf1(AsCpf1)やLachnospiraceae bacterium由来Cpf1(LbCpf1)はCas9と相補的なゲノム編集ツールとして利用されている。
本長期利用課題では異なる細菌に由来するCRISPR-Cas酵素の結晶構造を決定することにより、その多様なRNA認識機構、PAM認識機構、DNA切断機構を明らかにすることに成功した。
2. SaCas9の結晶構造
SaCas9(1053残基)はSpCas9(1368残基)よりもサイズが小さいため、ウイルスベクターへの導入効率の高いゲノム編集ツールとして利用されている[12][12] F. A. Ran et al.: Nature 520 (2015) 186-191.。SaCas9の作動機構の理解を目指し、SaCas9-sgRNA-DNA複合体を結晶化し、BL41XUにおいてX線回折データを収集し、SeMet置換体を用いたSAD法により結晶構造を決定した[15][15] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.(図2)。結晶構造から、SaCas9はSpCas9と同様に、RECローブとNUCローブからなる構造をもつことがわかった。NUCローブはRuvCドメイン、HNHドメイン、WEDドメイン、PIドメインから構成されていた。WEDドメインは新規フォールドをもつことが明らかとなった。sgRNAのガイド配列は標的DNAとRNA:DNAヘテロ2本鎖を形成し、2つのローブの間に結合していた。SaCas9とSpCas9のRECドメインおよびWEDドメインは構造が異なり、それぞれのsgRNAを特異的に認識していた。PAM(TTGAAT)は2本鎖を形成しWEDドメインとPIドメインの間に結合していた。PAMの1文字目、2文字目のT塩基(T1とT2)はSaCas9と相互作用していなかった。一方、PAMの3文字目がGであることと一致して、G3はArg1015と2本の水素結合を形成していた。A4はAsn985と水素結合し、A5はAsn985、Asn986、Arg991と水分子を介した水素結合により認識されていた。したがって、SaCas9はAとGに共通のN7と相互作用することにより、PAM(NNGRRT)の4文字目および5文字目のR(AまたはG)を認識していることが明らかになった。さらに、PAMの6文字目のTに対する嗜好性と一致して、T6はArg991と水素結合していた。PAM認識残基を変異させると、DNA切断活性が低下したことから、これらの相互作用の重要性が確認された。配列相同性は低いにもかかわらず、SaCas9とSpCas9のPIドメインは類似の構造をもっていた。しかし、PAM認識残基は異なっており、SpCas9ではArg1333とArg1335がPAM(NGG)を認識していた。これらの構造比較から、SaCas9とSpCas9が異なる配列をPAMとして認識する分子機構が明らかになった。
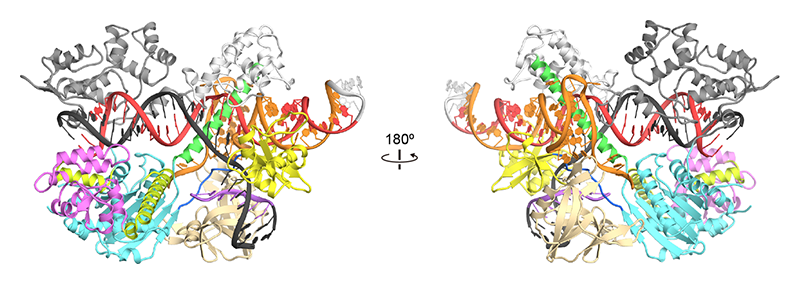
図2 SaCas9-sgRNA-DNA複合体の結晶構造
3. FnCas9の結晶構造
FnCas9は1629残基からなり、Cas9の中でも最もサイズが大きく、SpCas9やSaCas9との配列相同性が低い。FnCas9の作動機構の理解を目指し、FnCas9-sgRNA-DNA複合体を結晶化し、BL41XUにおいてX線回折データを収集し、SeMet置換体を用いたSAD法により結晶構造を決定した[16][16] H. Hirano et al.: Cell 164 (2016) 950-961.(図3)。FnCas9はRuvCドメイン、REC1ドメイン、REC2ドメイン、REC3ドメイン、HNHドメイン、WEDドメイン、PIドメインの7つのドメインから構成されていた。FnCas9のRuvCドメインおよびHNHドメインはSpCas9やSaCas9と同様の構造をとっていた。一方、RECドメインおよびWEDドメインは新規フォールドをもっていた。SpCas9、SaCas9、FnCas9の構造比較から、これらの3種のCas9の間の配列相同性は低いにもかかわらず、DNA切断機構は保存されていることが明らかになった。一方、3種のCas9の間でsgRNAの構造は大きく異なっており、それぞれ特徴的な構造をもつRECドメインとWEDドメインにより特異的に認識されていた。結晶構造から、FnCas9がNGGという配列をPAMとして認識する分子基盤も明らかになった。T1はFnCas9と相互作用していなかった一方、G2とG3はArg1585とArg1556とそれぞれ水素結合していた。さらに、得られた構造情報を基に変異を導入し、NGGではなくYGという配列をPAMとして認識するFnCas9改変体の作製に成功した。
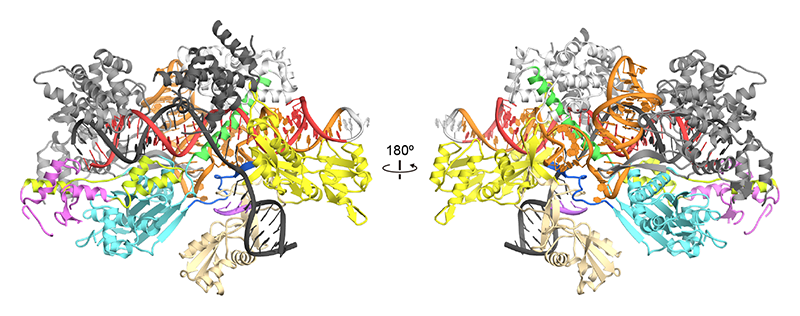
図3 FnCas9-sgRNA-DNA複合体の結晶構造
4. CjCas9の結晶構造
CjCas9は984残基からなる最小のCas9であるため、小型のゲノム編集ツールとして注目されている[13][13] E. Kim et al.: Nature Commun 8 (2017) 14500.。CjCas9の作動機構の理解を目指し、CjCas9-sgRNA-DNA複合体を結晶化し、BL41XUにてX線回折データを収集し、SeMet置換体を用いたSAD法により結晶構造を決定した[17][17] M. Yamada et al.: Mol Cell 65 (2017) 1109-1121.(図4)。結晶構造から、CjCas9は他のCas9と同様に、2つのローブからなることが確かめられた。RECローブはREC1ドメインとREC2ドメインから構成される一方、NUCローブはRuvCドメイン、WEDドメイン、および、PIドメインから構成されていた。CjCas9はSpCas9よりも小さなREC1ドメインとPIドメインをもち、さらに、SaCas9よりも小さなWEDドメインをもっていた。これらの構造比較からCjCas9の小型化の分子基盤が明らかになった。結晶構造から、CjCas9のsgRNAは他のCas9のsgRNAと異なり、予想外の3重らせん構造をもつことが明らかになった。さらに、他のCas9は非相補鎖の塩基をPAMとして認識するのに対し、CjCas9は相補鎖と非相補鎖の両方の塩基と水素結合を形成することによりPAM(NNNVRYAC)を認識していることが明らかになった。
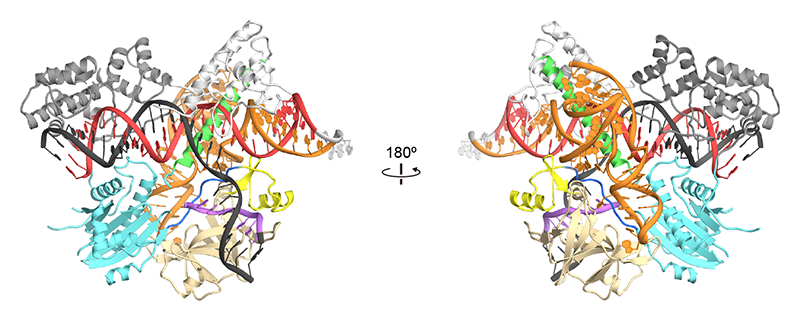
図4 CjCas9-sgRNA-DNA複合体の結晶構造
5. Cpf1の結晶構造
Cpf1の作動機構の理解を目指し、AsCpf1-crRNA-DNA複合体を結晶化し、BL41XUにおいてX線回折データを測定し、SeMet置換体を用いたSAD法により結晶構造を決定した[18][18] T. Yamano et al.: Cell 165 (2016) 949-962.(図5)。結晶構造から、Cpf1は2つのローブ(RECとNUC)からなることが明らかになった。RECローブはREC1ドメインとREC2ドメインから構成され、NUCローブはRuvCドメイン、WEDドメイン、PIドメイン、Nucドメインから構成されていた。RuvCドメインを除く5つのドメインは新規フォールドをもっていた。crRNAのガイド配列は標的DNAと20塩基のRNA:DNAヘテロ2本鎖を形成し、RECローブとNUCローブの間に結合していた。一方、crRNAの他の領域は予想外のシュードノット構造を形成し、WEDドメインとRuvCドメインによって認識されていた。PAM(TTTA)は歪んだ2重らせん構造をとり、WEDドメイン、REC1ドメイン、PIドメインと相互作用していた。PAMの連続したT塩基はPIドメインのLys607と水素結合していた。結晶構造と変異体解析の結果、NucドメインはDNA切断に関与することが示唆された。Cas9とCpf1の構造比較から2つのCRISPR-Cas酵素の間の機能的な収斂が明らかになった。さらに、LbCpf1-crRNA-DNA複合体[19][19] T. Yamano et al.: Mol Cell 67 (2017) 633-645.およびAsCpf1改変体-crRNA-DNA複合体[20][20] H. Nishimasu et al.: Mol Cell 67 (2017) 139-147.の結晶構造を決定し、Cpf1のPAM認識機構の詳細を明らかにした。
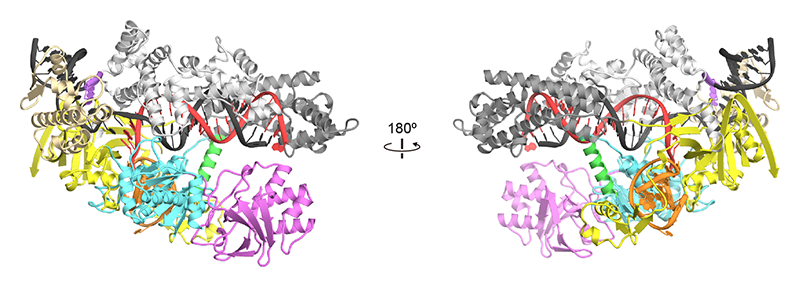
図5 AsCpf1-crRNA-DNA複合体の結晶構造
謝辞
X線回折実験は、SPring-8のBL41XU(課題番号:2015A0119)において行った。本研究は、JST戦略的創造研究推進事業(さきがけ)、JSPS、文部科学省および国立研究開発法人日本医療研究開発機構、内閣府戦略的イノベーション創造プログラム(SIP)の支援を受けて行った。
参考文献
[1] R. Barrangou et al.: Science 315 (2007) 1709-1712.
[2] K. S. Makarova et al.: Nat Rev Microbiol 13 (2015) 722-736.
[3] H. Nishimasu and O. Nureki: Curr Opin Struct Biol 43 (2017) 68-78.
[4] G. Gasiunas, R. Barrangou, P. Horvath and V. Siksnys: Proc Natl Acad Sci USA 109 (2012) E2579-2586.
[5] M. Jinek et al.: Science 337 (2012) 816-821.
[6] L. Cong et al.: Science 339 (2013) 819-823.
[7] H. Nishimasu et al.: Cell 156 (2014) 935-949.
[8] M. Jinek et al.: Science 343 (2014) 1247997.
[9] F. Jiang, K. Zhou, L. Ma, S. Gressel and J. A. Doudna: Science 348 (2015) 1477-1481.
[10] C. Anders, O. Niewoehner, A. Duerst and M. Jinek: Nature 513 (2014) 569-573.
[11] F. Jiang et al.: Science 351 (2016) 867-871.
[12] F. A. Ran et al.: Nature 520 (2015) 186-191.
[13] E. Kim et al.: Nature Commun 8 (2017) 14500.
[14] B. Zetsche et al.: Cell 163 (2015) 759-771.
[15] H. Nishimasu et al.: Cell 162 (2015) 1113-1126.
[16] H. Hirano et al.: Cell 164 (2016) 950-961.
[17] M. Yamada et al.: Mol Cell 65 (2017) 1109-1121.
[18] T. Yamano et al.: Cell 165 (2016) 949-962.
[19] T. Yamano et al.: Mol Cell 67 (2017) 633-645.
[20] H. Nishimasu et al.: Mol Cell 67 (2017) 139-147.
東京大学大学院 理学系研究科
〒113-0033 東京都文京区本郷7-3-1
TEL : 03-5841-4391
e-mail : nisimasu@bs.s.u-tokyo.ac.jp
東京大学大学院 理学系研究科
〒113-0033 東京都文京区本郷7-3-1
TEL : 03-5841-4391
e-mail : nureki@bs.s.u-tokyo.ac.jp








