Volume 18, No.1 Pages 8 - 13
1. 最近の研究から/FROM LATEST RESEARCH
セントロメアの形成に必須なCENP-T-W-S-Xの構造解析
Structural Analysis on CENP-T-W-S-X, Which is Required for Centromere Formation
国立遺伝学研究所 分子遺伝研究部門 Department of Molecular Genetics, National Institute of Genetics
- Abstract
- 細胞分裂の過程において、紡錘体微小管は染色体上の特殊領域に結合して染色体を娘細胞へと均等に分配する。紡錘体微小管が結合する染色体上の特殊構造はキネトコアと呼ばれ、キネトコアが形成される染色体領域はセントロメアと定義されている。通常、染色体上にセントロメア領域は一カ所だけであるが、その領域はDNA配列の特異性によって規定されていない。しかし、いったん決まったセントロメア領域は、次世代細胞においてもセントロメアとなるので、DNA配列以外のエピジェネティックな機構により、セントロメア領域は規定されていると考えられる。エピジェネティックマーカーとしては、セントロメアに特異的なヒストンであるCENP-Aが注目されている。筆者らは、CENP-Aに加えてセントロメアに特異的なクロマチン構造の構築には、CENP-T-W-S-Xと名付けた複合体が重要であると提唱している。最近、CENP-T-W-S-Xの構造解析に成功し、その構造からセントロメア形成に重要である特異的なクロマチン構造の実体が見えてきた。
はじめに
生物の生命維持のためには、全ゲノム情報を担う染色体が、細胞周期の過程を通じて正確に複製され、娘細胞へと均等に分配されなければならない。染色体の複製や分配に異常がおきると、娘細胞における染色体数に変化が生じて、がん化の原因になることが知られている。したがって、正確な均等分配を保障する染色体分配の分子機構を解明することは、基礎生物学および医科学の両面から大切な研究である。細胞周期のS期で複製された染色体は、M期に両極から延びた紡錘体微小管によって捉えられ、娘細胞へと分配されるが、この染色体分配の際に、紡錘体微小管と結合する染色体上の特殊構造がキネトコア(動原体)と呼ばれている(図1)。キネトコアが形成される染色体領域が、セントロメアと定義される。セントロメアは、通常染色体上に一カ所形成されるが、セントロメア領域に特異的なDNA配列は存在しない。しかし、セントロメア領域の位置は、世代を超えて受け継がれるので、DNA配列に依存しない分子機構(このことをエピジェネティックな分子機構と呼ぶ)によってセントロメア領域の位置情報は規定されていると考えられている。このセントロメア領域の位置情報を規定する目印(これをエピジェネティックマーカーと呼ぶ)は大変興味深い。DNA配列が目印でないとすると、DNAと結合するクロマチンタンパク質の中に目印情報が潜んでいる可能が高い。これまでの多くの研究によって、セントロメア領域を規定するエピジェネティックマーカーの一つは、セントロメア領域に特異的に存在するヒストンH3であるCENP-Aであると考えられている [1, 2] [1] B. E. Black and D. W. Cleveland: Cell 144 (2011) 471-479.
[2] M. Perpelescu and T. Fukagawa: Chromosoma 120 (2011) 425-446.。CENP-Aは種を超えて保存され、どのようなDNA配列を持つセントロメア領域にもCENP-Aは存在しているので、CENP-Aがセントロメアの形成に重要な働きを担っていることは示唆される。しかし、CENP-Aだけでは、キネトコアは形成できないので [2] [2] M. Perpelescu and T. Fukagawa: Chromosoma 120 (2011) 425-446.、CENP-Aと一緒にセントロメア領域に特異的なクロマチン構造の構築や機能的なキネトコアの形成に関与するタンパク質の役割を解明することが重要であると筆者らは考えていた。そこで、セントロメアDNAに結合するタンパク質の探索を行った結果、CENP-T-Wというタンパク質がセントロメアに存在しているDNA(特異的な配列はない)と結合することを数年前に見いだした [3] [3] T. Hori, M. Amano, A. Suzuki, C. B. Backer, J. P. Welburn et al.: Cell 135 (2008) 1039-1052.。そこで、セントロメア領域に特異的なクロマチン構造の実体の解明がセントロメア形成の分子機構の理解につながると考え、CENP-T-Wの構造解析を手がかりに研究を進めることにした。
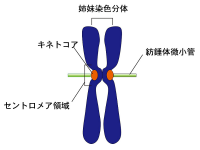
図1 分裂期染色体
分裂期の染色体は、両極から伸びた紡錘体微小管によって捉えられ、娘細胞へ分配される。紡錘体微小管の結合する染色体の特殊構造がキネトコアであり、キネトコアが形成される染色体領域をセントロメアと呼ぶ。
1.セントロメアDNAと結合するCENP-T-W複合体
我々の研究室では、セントロメアに特異的なクロマチン構造を構成するタンパク質を同定する目的で、セントロメアタンパク質CENP-HおよびCENP-Iと結合するタンパク質の探索を行い、複数のセントロメアタンパク質を同定した [4] [4] M. Okada, I. M. Cheeseman, T. Hori, K. Okawa, I. X. McLeod et al.: Nat. Cell Biol. 8 (2006) 446-457.。同時期に、日米の他のグループは、セントロメア特異的なヒストンであるCENP-Aとゆるやかに結合するタンパク質の同定を試みて、複数のタンパク質の同定に成功している [5, 6] [5] D. R. Foltz, D. R. Jansen, L. E. Black, B. E. Bailey, A. O. Yates, J. R. III et al.: Nat. Cell Biol. 8 (2006) 458-469.
[6] H. Izuta, M. Ikeno, N. Suzuki, T. Tomonaga, N. Nozaki et al.: Genes Cells 11 (2006) 673-684.。これらの結果と我々の研究室での解析を併せて、CENP-C, -H, -I, -K, -L, -M, -N, -O, -P, -Q, -R, -S, -T, -U, -W, -Xの16種類がセントロメアのクロマチン領域に存在するタンパク質である。これにCENP-Aを加えたタンパク質が、細胞周期を通じて、セントロメアのクロマチン領域に存在している。これらタンパク質は、細胞周期を通じて構成的にセントロメア領域に存在していることから、我々はこれらをCCAN(Constitutive Centromere Associated Network)タンパク質と呼んでいる [7, 8] [7] I. M. Cheeseman and A. Desai: Nat. Rev. Mol. Cell. Biol. 9 (2008) 33-46.
[8] T. Hori and T. Fukagawa: Chromosome Res. 20 (2012) 547-561.。
我々は、これらCCANタンパク質の中で、CENP-TとCENP-Wに特に注目して研究を行い、これらが複合体を形成して、セントロメアのDNAと直接に結合していることを見いだした [3] [3] T. Hori, M. Amano, A. Suzuki, C. B. Backer, J. P. Welburn et al.: Cell 135 (2008) 1039-1052.。当初、CENP-T-WがセントロメアDNAと結合することから、CENP-Aと結合していることを予想していたが、意外なことに、CENP-T-W複合体とDNAとの結合は、CENP-Aとは非依存的におこることが明らかとなった [3] [3] T. Hori, M. Amano, A. Suzuki, C. B. Backer, J. P. Welburn et al.: Cell 135 (2008) 1039-1052.。しかしながら、CENP-T-Wのセントロメアへの局在性には、CENP-Aヌクレオソームの存在が必要であることから、セントロメアに特異的なクロマチン構造では、CENP-T-W-DNA複合体とCENP-Aを含むヌクレオソームの両方が重要であると考察した [3] [3] T. Hori, M. Amano, A. Suzuki, C. B. Backer, J. P. Welburn et al.: Cell 135 (2008) 1039-1052.。次の疑問点は、どのようにCENP-T-W複合体は、セントロメア領域を認識してセントロメアDNAと結合しているのであろうかということであった。さらに、CENP-T-Wがどのように、セントロメア形成に関与しているのかについては不明な点が多かった。そこで、我々は、CENP-T-W複合体とセントロメアのクロマチン構築との関連を明らかにする目的で、構造生物学、生化学、細胞生物学を駆使して研究を行い興味深い結果を得ることができた [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。
2.CENP-T-Wの構造解析
我々の研究室では、タンパク質の生体内での機能解析には、ニワトリのDT40細胞を使用している。ニワトリのDT40細胞内では、遺伝子改変が比較的効率よく行うことができるためである。そこで、ニワトリのCENP-TおよびCENP-Wの構造解析を試みた。CENP-Tは、600アミノ酸程度の長さを持つタンパク質であるが、我々は、以前にC末端領域約110アミノ酸の領域がCENP-Wと結合してDNAと相互作用することを見いだしていた。そこで、ニワトリCENP-TのDNA結合に関わるC末端領域約110アミノ酸の領域と完全長のニワトリCENP-Wを大腸菌内で発現させて精製した後、複合体の再構成を行った。その後、再構成したCENP-T-W複合体の結晶化を試み、結晶のX線構造解析を行った。X線構造解析は、SPring-8のBL38B1およびBL44XUのビームライン(課題番号: 2010B1060, 2011A6633, 2011A1213)を用いた。その結果、2.2 Å分解能で構造決定に成功した(図2, PDB: 3B0C, 3B0D)。構造解析の結果、CENP-T-Wはヘテロ2量体を形成し、構造は、ヌクレオソーム中のヒストン [10] [10] K. Luger, A. W. Mäder, R. K. Richmond, D. F. Sargent and T. J. Richmond: Nature 389 (1997) 251-260.の2量体部分と類似していた。ヌクレオソームは、ヒストンH2A、H2B、H3、H4を2分子ずつ含む8量体のまわりをDNAが1.65回巻いた構造をとることが明らかにされているが [10] [10] K. Luger, A. W. Mäder, R. K. Richmond, D. F. Sargent and T. J. Richmond: Nature 389 (1997) 251-260.、構造解析の過程でヒストン分子とDNAの結合部位が明らかにされている。その情報を参考にして、CENP-TおよびCENP-WのDNA結合に関わる領域を予想した。実際に、その領域に変異を持つCENP-T-W複合体を精製してDNA結合活性を解析した結果、結合活性が低下することを確認できた。
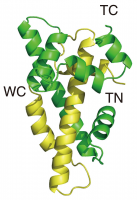
図2 CENP-T-W複合体の構造
CENP-T-W 2量体の構造(リボン図)。CENP-T、CENP-Wは、それぞれ、緑、黄色で表している。
さらに、このDNA結合活性の低下が、生体内のCENP-T-W複合体の機能と関連しているかを調べる目的で、野生型のCENP-Wの代わりに、DNA結合が低下した変異型CENP-Wを発現するようなDT40細胞を作成したところ、変異タンパク質はセントロメアへ局在できないため機能しないことが明らかになった [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。構造解析、生化学および細胞生物学実験の結果を総合的に考察して、ヒストンに構造が類似したセントロメアタンパク質複合体CENP-T-WとDNAとの結合は、セントロメア機能に必須であると結論した。
3.CENP-T-W複合体と構造が類似したCENP-S-X複合体
我々の研究室では、複数のセントロメアタンパク質を解析する過程において、CENP-S-X複合体を同定していた [11] [11] M. Amano, A. Suzuki, T. Hori, C. Backer, K. Okawa et al.: J. Cell Biol. 186 (2009) 173-182.。興味深いことに、CENP-S-X複合体は、CENP-T-Wと共沈降することが、CENP-Sの免疫沈降実験から示されていた [11] [11] M. Amano, A. Suzuki, T. Hori, C. Backer, K. Okawa et al.: J. Cell Biol. 186 (2009) 173-182.。そこで、セントロメアのクロマチン形成や機能形成を理解するためには、CENP-T-W複合体とCENP-S-X複合体との関係をより詳細に解析する必要性があると考えて、CENP-T-W複合体の解析と同様に、ニワトリCENP-S-X複合体を大腸菌内で発現、精製後、構造生物学的解析を行った。X線構造解析は、SPring-8のBL38B1のビームライン(課題番号: 2010B1059, 2011A1211)を用いた。精製したCENP-S-X複合体をゲル濾過に供したところ、CENP-S-Xの予想される分子量より大きい場所にピークが得られたので、CENP-S-X複合体は多量体を形成することが示唆された。結晶化した後に、X線結晶構造解析を行った結果、2.15 Å分解能で構造決定に成功し、結晶中では、CENP-S-X複合体は4量体構造を形成することが明らかとなった(図3, PDB: 3B0C)。CENP-S-X複合体の構造もまた、ヒストンに類似しており、ヒストンとの構造類似性から、DNA結合領域を予想できた。CENP-Wと異なり、DNAへの結合性を低下させた変異型CENP-S-X複合体は、セントロメアへの局在そのものは可能であったものの、他のセントロメアタンパク質の集合に不全がおきており、CENP-S-Xの機能が正常に機能していなかった [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。以上の結果から、CENP-S-X複合体の機能にもDNA結合活性が貢献していると考察できた。
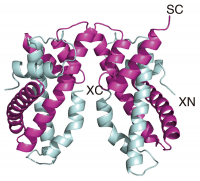
図3 CENP-S-X複合体の構造
CENP-S-X 4量体の構造(リボン図)。CENP-S、CENP-Xは、それぞれ、マゼンタ、シアンで表している。
4.ヘテロ4量体構造を形成するCENP-T-W複合体とCENP-S-X複合体
CENP-T-W複合体とCENP-S-X複合体は、ともにヒストンに構造が類似していた(図2, 3)。CENP-T-W複合体は2量体を形成し、CENP-S-X複合体は4量体を形成するといった違いはあるものの、この構造の類似性は、機能的に類似していることを予想させる。また、以前の研究で、CENP-T-W複合体とCENP-S-X複合体が結合していることが示唆されていたので [11] [11] M. Amano, A. Suzuki, T. Hori, C. Backer, K. Okawa et al.: J. Cell Biol. 186 (2009) 173-182.、セントロメア内において、これらの両複合体が協調して機能している可能性を考えた。そこで、CENP-T-W複合体とCENP-S-X複合体の結合様式を理解する目的で、両複合体を混合してゲル濾過解析を行った。上述のように、結晶中ではCENP-T-W複合体は2量体をCENP-S-Xは4量体構造を形成しているので、この両複合体が結合すれば、6量体かあるいは多量体を形成して、より分子量の大きい場所にゲル濾過のピークが検出できると考えた。ところが、1本のピークが、CENP-S-X複合体単独のピーク(4量体と予想されるピーク)より、分子量がやや小さい場所に検出できた。実際に、このピーク内には、CENP-T、-W、-S、-Xが、ほぼ同じ量ずつ含まれることは、SDS-PAGEの解析で明らになった。これらの結果を総合的に考察すると、CENP-S-X単独とCENP-T-W単独ではそれぞれ4量体と2量体を形成するが、両者を混合させると、CENP-S-Xの4量体の一部がCENP-T-Wの2量体と交換してCENP-T-W-S-Xのヘテロ4量体を形成すると考えられた [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。
そこで、この生化学的解析の結果による考察を明らかにするために、CENP-T-W-S-X複合体を結晶化しての構造解析を試みた。X線構造解析は、SPring-8のBL38B1のビームライン(課題番号: 2011B1146)を用いた。その結果、2.4 Å分解能でCENP-T-W-S-X複合体の構造を決定することができた(図4, PDB: 3VH5, 3VH6)。予想通りに、CENP-T-W-S-Xはヘテロ4量体を形成しており、このヘテロ4量体に必要な自由エネルギーは、CENP-S-Xの4量体形成よりも、低いことが計算された。このことは、CENP-T-W-S-X 4量体が、CENP-S-Xの4量体に比べて安定であること示している [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。さらに、結晶構造の解析から4量体形成に必須なCENP-SおよびCENP-Tのアミノ酸部位を予想することができた。実際に、これらのアミノ酸部位に変異を加えたCENP-Sあるいは、CENP-T変異タンパク質を精製して生化学的な解析を行った結果、変異型CENP-Sあるいは変異型CENP-Tは、それぞれCENP-XあるいはCENP-Wと2量体を形成することはできるものの、CENP-T-W-S-Xのへテロ4量体を形成できないことが判明し、予想したアミノ酸領域が4量体形成に関わることを証明できた [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。
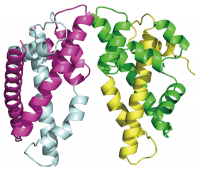
図4 CENP-T-W-S-X複合体の構造
CENP-T-W-S-Xへテロ4量体の構造(リボン図)。CENP-T、CENP-W、CENP-S、CENP-Xは、それぞれ、緑、黄色、マゼンタ、シアンで表している。
5.機能的なキネトコアの形成に必須なCENP-T-W-S-Xのへテロ4量体構造
in vitroで再構成されたCENP-T-W-S-Xが、溶液あるいは結晶中では、ヘテロ4量体を形成することは明らかになったが、この4量体形成が細胞内でセントロメア機能とどのように関わっているかを明らかにすることは重要である。我々は、CENP-SあるいはCENP-Tのノックアウト細胞を保有しているので、4量体が形成できない変異型CENP-Sあるいは変異型CENP-Tをそれぞれのノックアウト細胞へ導入してそれぞれの機能を補えるかどうかを解析した(図5)。4量体形成ができないCENP-S変異体は、核内に拡散した局在を示し、セントロメアへ局在できなかった(図5A)。この結果は、CENP-Sのセントロメアへの局在には、CENP-T, -W, -Xとの4量体形成が必須であること示している。また、CENP-Tの変異体をCENP-TのノックアウトDT40細胞へ導入した結果、変異型CENP-Tは、セントロメアには局在できるものの、シグナルの強さは、野生型のシグナルに比べて20%程度に減少していた。さらに、細胞分裂期に異常を持つ細胞が多く観察され、セントロメア形成に不全がおきている可能性が考えられた。そこで、セントロメアに存在する微小管結合タンパク質であるNdc80の局在を調べると、そのセントロメア局在も大きく損なわれていた(図5B)。これらの結果から、CENP-T-W-S-Xのヘテロ4量体構造の形成は、細胞内においてもセントロメア領域の正常なクロマチン構造の構築に必須であることが、明らかとなった [9] [9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.。
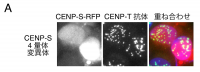
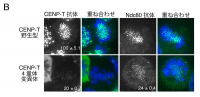
図5 CENP-T-W-S-Xの4量体形成変異体は、セントロメア機能を失う
(A)変異を導入して4量体を形成できないCENP-SにRFPを融合したCENP-S-RFP(赤)は、セントロメアに局在しない。セントロメアの場所はCENP-T 抗体(緑)でマークした。(B)変異を導入して4量体を形成できないCENP-Tは、セントロメア局在が大きく失われる。この細胞では、微小管結合タンパク質Ndc80の局在も失われる。
6.ヌクレオソームに似た特性を持つDNA-CENP-T-W-S-X複合体
ヒストン8量体の周りにDNAが、1.65回巻きついて、ヌクレオソーム構造をとることは、よく知られている [10] [10] K. Luger, A. W. Mäder, R. K. Richmond, D. F. Sargent and T. J. Richmond: Nature 389 (1997) 251-260.。ヒストンと構造が類似したCENP-T-W-S-Xヘテロ4量体も、DNA結合部位が類似していることから、DNAがCENP-T-W-S-X複合体の周辺を巻いたように結合する可能性も予想された。この予想に答えるために、CENP-T-W-S-X複合体がDNAへ超らせんを導入する活性(DNAスーパーコイリング導入活性)を有しているかどうかを解析した。これは確立した手法があり、環状型のDNA(プラスミドDNAなど)を酵素トポイソメラーゼⅠによりリラックス型に変換させたのち、DNAへ対象とするタンパク質を加えて、DNAの高次構造の変化をアガロース電気泳動で解析する方法である。ヒストンを用いてこの実験を行うと、ヌクレオソームの形成に準じて、環状型DNAのトポアイソマーを電気泳動においてはしご状に検出することができる。実際にCENP-T-W-S-X複合体を用いてDNAスーパーコイリング導入活性を調べると、ヒストンより活性は弱いものの、トポアイソマーが検出でき、スーパーコイリング導入活性を有することが明らかになった(図6)。CENP-SあるいはCENP-Xの変異型タンパク質では、スーパーコイリング導入活性が減少したことからも、CENP-T-W-S-Xの周辺をDNAが巻くように結合している可能性が高いと結論した。ただし、ヒストンに比べてその活性が弱いことから、ヒストンのようにDNAが約1.65回完全に巻き付くような結合ではないと予想している。
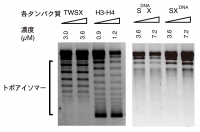
図6 CENP-T-W-S-XによるDNA超らせん化導入活性検定
CENP-T-W-S-Xを対象としたDNAスーパーコイリング導入活性のアッセイ結果(左の2レーン)。ヒストンH3-H4と同様にトポアイソマーが観察できる(3, 4 レーン)。一方、CENP-Sあるいは、CENP-Xに変異を導入すると活性は見られない(右のゲル)。
おわりに
ここで紹介した研究結果は、いくつかの新規セントロメアタンパク質の構造解析にとどまらず、セントロメアにおける新規のクロマチン構造の存在を示唆したものである(図7) [12] [12] T. Fukagawa: Epigenetics 7 (2012) 672-675.。これは、構造生物学や、セントロメア生物学の分野だけでなく、クロマチン生物学全体に大きなインパクトを与えるものと考えている。ヒストンには、多くのタイプが存在して、様々なタイプの組み合わせによって独自のクロマチン機能を果たすことが知られている。例えば、ヒストンH2AZとH3.3がハイブリッドヌクレオソームを形成して転写の活発な領域に存在することが近年報告されている [13] [13] C. Jin, C. Zang, G. Wei, K. Cui, W. Peng et al.: Nat. Genet. 41 (2009) 941-945.。このように、多様なヒストンタイプの組み合わせは、ヒストン修飾とは次元を異にするクロマチンマークである。セントロメア領域にも、ヒストンH3のあるタイプであるCENP-Aが独自にヌクレオソームを形成して、セントロメアのアイデンティティを獲得している。近年、CENP-Aを含むヌクレオソームのX線結晶構造も明らかにされている [14] [14] H. Tachiwana, W. Kagawa, T. Shiga, A. Osakabe, Y. Miya et al.: Nature 476 (2009) 232-235.。CENP-T-W-S-Xに関しては、この組み合わせにおいてセントロメアで機能を発揮することは、明らかであるが、CENP-S-Xは、セントロメア領域に加えてDNA損傷部位に存在することが知られている [15, 16] [15] T. R. Singh, D. Saro, A. M. Ali, X. F. Zhen, C. H. Du et al.: Mol. Cell 37 (2010) 879-886.
[16] Z. Yan, M. Delannoy, C. Ling, D. Daee, F. Osman et al.: Mol. Cell 37 (2010) 865-878.。その際、CENP-S-Xは、FANCMというタンパク質と複合体を形成している [17] [17] Y. Tao, C. Jin, X. Li, S. Qi, L. Chu et al.: Nat. Commun. 3 (2012) 782.。もちろん、DNA損傷部位には、CENP-T-Wが存在しないことから、その部位ではCENP-S-XーFANCM複合体でDNAと結合している可能性が考えられる。しかしながら、CENP-S-Xタンパク質がCENP-T-Wと結合すると、セントロメアに局在し独自のクロマチン構造を形成するような機能の使い分けが行われているようである(図7)。既存のヒストンタイプでなく、ヒストンと類似したタンパク質の組み合わせで独自の機能を発揮するという我々のモデルは、これまでのモデルを広げた新しいクロマチンコードと言える [12, 18] [12] T. Fukagawa: Epigenetics 7 (2012) 672-675.
[18] D. R. Foltz and P. T. Stukenberg: Cell 148 (2012) 394-396.
。このように、ヒストンと類似タンパク質の組み合わせによるコードは、今後も発見される可能性は高いと我々は考えている。
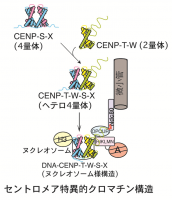
図7 セントロメアに特異的なクロマチン構造
CENP-S-Xの4量体とCENP-T-Wの2量体が結合して、CENP-T-W-S-Xのヘテロ4量体を形成する。CENP-T-W-S-X複合体は、ヒストンに似た構造をとっており、通常のヒストンの周りをDNAが巻くように、CENP-T-W-S-Xの周りも、DNAが巻いたような独自の構造をつくる(DNA-CENP-T-W-S-X構造:ヌクレオソーム様構造)。セントロメアには、CENP-Aを含むヌクレオソーム、ヒストンH3からなるヌクレオソーム、そして、CENP-T-W-S-Xを含むヌクレオソーム構造が強調して働いていると考えている。
謝辞
本研究におけるX線結晶構造解析は、SPring-8のBL38B1およびBL44XUのビームラインを使用している(課題番号は本文参照)。ここに感謝する。
参考文献
[1] B. E. Black and D. W. Cleveland: Cell 144 (2011) 471-479.
[2] M. Perpelescu and T. Fukagawa: Chromosoma 120 (2011) 425-446.
[3] T. Hori, M. Amano, A. Suzuki, C. B. Backer, J. P. Welburn et al.: Cell 135 (2008) 1039-1052.
[4] M. Okada, I. M. Cheeseman, T. Hori, K. Okawa, I. X. McLeod et al.: Nat. Cell Biol. 8 (2006) 446-457.
[5] D. R. Foltz, D. R. Jansen, L. E. Black, B. E. Bailey, A. O. Yates, J. R. III et al.: Nat. Cell Biol. 8 (2006) 458-469.
[6] H. Izuta, M. Ikeno, N. Suzuki, T. Tomonaga, N. Nozaki et al.: Genes Cells 11 (2006) 673-684.
[7] I. M. Cheeseman and A. Desai: Nat. Rev. Mol. Cell. Biol. 9 (2008) 33-46.
[8] T. Hori and T. Fukagawa: Chromosome Res. 20 (2012) 547-561.
[9] T. Nishino, K. Takeuchi, K. E. Gascoigne, A. Suzuki, T. Hori et al.: Cell 148 (2012) 487-501.
[10] K. Luger, A. W. Mäder, R. K. Richmond, D. F. Sargent and T. J. Richmond: Nature 389 (1997) 251-260.
[11] M. Amano, A. Suzuki, T. Hori, C. Backer, K. Okawa et al.: J. Cell Biol. 186 (2009) 173-182.
[12] T. Fukagawa: Epigenetics 7 (2012) 672-675.
[13] C. Jin, C. Zang, G. Wei, K. Cui, W. Peng et al.: Nat. Genet. 41 (2009) 941-945.
[14] H. Tachiwana, W. Kagawa, T. Shiga, A. Osakabe, Y. Miya et al.: Nature 476 (2009) 232-235.
[15] T. R. Singh, D. Saro, A. M. Ali, X. F. Zhen, C. H. Du et al.: Mol. Cell 37 (2010) 879-886.
[16] Z. Yan, M. Delannoy, C. Ling, D. Daee, F. Osman et al.: Mol. Cell 37 (2010) 865-878.
[17] Y. Tao, C. Jin, X. Li, S. Qi, L. Chu et al.: Nat. Commun. 3 (2012) 782.
[18] D. R. Foltz and P. T. Stukenberg: Cell 148 (2012) 394-396.
国立遺伝学研究所 分子遺伝研究部門
〒411-8540 静岡県三島市谷田1111
TEL:055-981-6744
e-mail:tnishino@nig.ac.jp
国立遺伝学研究所 分子遺伝研究部門
〒411-8540 静岡県三島市谷田1111
TEL:055-981-6792
e-mail:tfukagaw@nig.ac.jp








