Volume 09, No.4 Pages 269 -274
3. 最近の研究から/FROM LATEST RESEARCH
光合成の酸素発生に関わるタンパク質の立体構造を解明
−光合成植物の進化に新たな知見−
Elucidation of the Crystal Structure of the PsbP Protein Involved in the Oxygen Evolution in Photosynthesis
京都大学大学院 生命科学研究科/(独)理化学研究所 播磨研究所 Graduate School of Biostudies, Kyoto University / Harima Institute, RIKEN
- Abstract
- Photosynthesis is a multi-step reaction that utilizes light energy to convert carbon dioxide into sugar and generate oxygen as by-product. The first step of photosynthesis is the oxygen-evolving reaction performed by the protein-pigment complex called “photosystem II”, and PsbP is one of the protein subunits constituting photosystem II. Since PsbP exists only in higher plants and green algae, its existence has been the subject of inquiry in the process of plant evolution. In order to elucidate the origin and function of PsbP from its 3D structure, a high-resolution analysis based on multiple wavelength anomalous dispersion method using the X-ray of SPring-8 was conducted. The result showed that the structure of PsbP was not similar to any known structures in photosystem II from cyanobacteria, primitive organisms doing oxygenic photosynthesis. This research shows one aspect of the evolution of oxygenic photosynthetic organisms.
1.はじめに
「光合成」は一般的には「植物が太陽光を利用して二酸化炭素を吸収し、糖に変換すると同時に酸素を発生する反応」として理解されている。厳密にはこれを「酸素発生型光合成」と呼ぶが、もう少し詳しくみると、その最初のステップは太陽エネルギーを利用して水分子を分解し、酸素と水素イオン、そして二酸化炭素の還元に必要な電子を取り出す反応から始まる。この反応を行うのが、植物の場合、葉緑体という細胞内小器官に存在する光化学系Ⅱと呼ばれるタンパク質複合体である。
酸素発生型光合成を行う生物には高等植物だけでなく、コケや真核藻類、さらには原核生物であるシアノバクテリアも含まれる。このうちシアノバクテリアは、太古の時代に真核生物のなかに取り込まれて葉緑体の起源となった生物に近いと考えられている光合成を行う細菌である。従って、光化学系Ⅱ複合体を構成するタンパク質もシアノバクテリアから高等植物にいたるまで基本的にはよく保存されている。ところが、実際に水分解−酸素発生反応が起る反応中心の周辺にあるタンパク質に関しては、高等植物・緑藻はシアノバクテリアと異なるタンパク質を持っている。即ち、シアノバクテリアではPsbV、PsbUと呼ばれるタンパク質が高等植物・緑藻ではPsbP、PsbQへ機能的に置き換わっているのである[1][1]J. De Las Rivas, M. Balsera and J. Barber : Trends in Plant Sci. 9 (2004) 18.(図1)。これらのタンパク質は、水分解−酸素発生反応に必須な補欠因子であるカルシウムイオンと塩素イオンの光化学系Ⅱへの結合に関わり、その反応を最適化するのに必要[2][2]A. Seidler : Biochim. Biophys. Acta 1277 (1996) 35.なのだが、光合成生物の進化の過程において、おそらくはその生育環境にあわせて変化したのだと推測される。しかしながらその詳細は明らかにされておらず、植物科学における謎の一つであった。
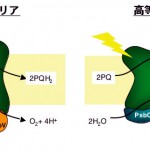
図1 シアノバクテリアと高等植物の光化学系II複合体の簡単な模式図による比較
光化学系Ⅱ複合体は光エネルギーを利用して水分子を酸素、水素原子、そして電子に分解する。取り出された電子は、プラストキノン(PQ)と呼ばれる分子を還元し(PQH2)、次のステップへと運ばれる。水分解−酸素発生反応を直接触媒するのは4分子のマンガン原子(Mn)である。その周辺には酸素発生系とよばれる膜表在型のタンパク質が存在するが、その構成タンパク質がシアノバクテリア(PsbO, PsbV, PsbU, PsbQ?)と高等植物(PsbO, PsbP, PsbQ)では異なっている。PsbQに関しては、シアノバクテリアの光化学系ⅡにPsbQと似たタンパク質が存在するとの報告がある[15][15]Y. Kashino et al. : Biochemistry 41 (2002) 8004.が、どこに結合しているかは明らかではない。(図は文献[1][1]J. De Las Rivas, M. Balsera and J. Barber : Trends in Plant Sci. 9 (2004) 18. を参考に作成した。)
こうした謎に迫る一つの手段として、タンパク質の立体構造を比較する事が考えられる。近年の構造生物学の進展に伴い、光化学系Ⅱに関してもその全体の立体構造が明らかにされつつあるが、特に好熱性シアノバクテリア由来の光化学系Ⅱに関しては、SPring-8を利用した日本の研究者グループを含め、複数のグループが高分解能構造解析に向けてしのぎを削っている[3-5][3]A. Zouni et al. : Nature 409 (2001) 739.
[4]N. Kamiya and J.-R. Shen : Proc. Natl. Acad. Sci. USA. 100 (2003) 98.
[5]K. N. Ferreira, T. M. Iverson, K. Maghlaoui, J. Barber and S. Iwata : Science 303 (2004) 1831.。その結果、シアノバクテリアの光化学系Ⅱ反応中心の周辺にあるタンパク質、PsbO、PsbU、PsbVに関してもその構造と複合体内における存在様式に関する情報が得られている。一方で、高等植物由来の光化学系Ⅱに関しては、電子顕微鏡を用いたおおまかな情報が報告されている[6][6]K. -H. Rhee, E. P. Morris, J. Barber and W. Külbrandt : Nature 396 (1998) 283.のみで、従ってこれまでPsbP、PsbQに関する構造情報は全く存在していなかった。
最近、イタリアのグループによって、ホウレンソウ由来のPsbQの結晶構造が1.9Åの分解能で報告された[7][7]V. Calderone et al. : EMBO Rep. 4 (2004) 900.。ついで筆者らのグループが、タバコ由来のPsbPの結晶構造を1.6Åの分解能で報告した[8][8]K. Ifuku, T. Nakatsu, H. Kato and F Sato : EMBO Rep. 5 (2004) 362.。これによって、シアノバクテリアと高等植物の光化学系Ⅱを構成するサブユニットのうち、一部の低分子サブユニットを除いた代表的なサブユニットの立体構造情報が明らかになったことになる。本稿では筆者が構造解析に携わった高等植物由来のPsbPの結晶構造に関して、最新の知見を踏まえながら紹介したいと思う。
2.結晶化と構造解析
PsbPは親水性のタンパク質であり、かつ植物中に大量に存在する事から、X線結晶構造解析に必要な高純度のタンパク質を精製することは比較的容易である。ところがこれまで複数のグループがPsbPの構造決定に挑戦してきたにもかかわらず、いずれも結晶化に成功していなかった。その一つの要因として、これまで光合成研究には伝統的にホウレンソウやエンドウといった、葉緑体を生化学的に単離しやすい植物材料が好んで使われてきたために、結晶化にもそうした植物に由来するPsbPのみが試されていた事が考えられた。そこで筆者らは、色々な植物からPsbP遺伝子を集め、各々のPsbPを大腸菌で作らせて精製し、その中から良い結晶が得られるものを探した。その結果、タバコに由来するPsbPの一つがX線結晶構造解析に適していることが判明した[9][9]K. Ifuku, T. Nakatsu, H. Kato and F. Sato : Acta Crystallogr. D59 (2003) 1462. (図2)。

図2 タバコ由来PsbPタンパク質の結晶
得られた結晶はX線結晶構造解析に十分なX線回折能を持っていたが、結晶ごとに結晶を構成する単位格子が微妙に異なるやっかいな性質を示した。そこで結晶中に含まれるタンパク質分子の組成を調べた結果、一部が分解されて短くなったタンパク質分子が含まれていることが判明した。そこでその分解を防ぐ試薬を探し出して結晶化の際に加えたところ、今度は逆に結晶化が全く見られなくなってしまった。結局、分解を受け易い部分をあらかじめ削ったタバコPsbPを大腸菌で発現させて精製し、結晶化させる事でこの問題を克服した。
次に問題となったのが、新規なタンパク質構造を決めるのに欠かせない位相決定の段階であった。位相決定には重原子同形置換法と多波長異常分散法が用いられるが、PsbPの結晶は重原子溶液中で容易に単位格子構造が変化するために重原子同形置換法は断念せざるを得なかった。つぎにSPring-8の放射光(ビームラインBL45PX)を利用した多波長異常分散法による決定を試みたが、この場合もなかなか位相の決定には至らず非常に苦労した。試行錯誤の結果、重原子との反応が結晶に一部ひび割れが入る程に進んでいて、かつX線回折能を完全には失っていない結晶から得たデータを手がかりに、重原子置換結晶の構造を2.0Åの分解能で決定できた。後から考えれば、PsbP結晶の単位格子構造が重原子溶液中で変化する過程において、結晶中の全ての単位格子構造が完全に変わりきらない間は結晶内部が双晶の状態になっており、たとえ重原子が結合していても位相決定には結びつかなかったのであろう。その後、SPring-8のビームラインBL44B2で収集したNative結晶のデータを用いて分子置換法を行い、最終的には1.6Åの分解能でPsbPの立体構造決定に至った。こうした一連の実験経過を振り返ると、一個の重原子置換結晶で構造決定ができる多波長異常分散法と、迅速かつ高精度なデータ収集による有意義な試行錯誤が可能であったSPring-8を利用できたことが成功の鍵であった。
3.立体構造から明らかになったこと
PsbPの構造は、アミノ末端側の短いβストランドで構成される構造(ドメインⅠ:ピンク色の部分)と、β-シート構造の両側をαへリックスで挟んだ中央の構造(ドメインⅡ:青色の部分)の2つのドメイン構造で構成されていた(図3)。このうちドメインⅠの配列は、筆者らの過去の研究においてカルシウムイオンと塩素イオンの保持に重要であると判明している[10, 11][10]K. Ifuku and F. Sato : Biochim. Biophys. Acta 1546 (2001) 196.
[11]K. Ifuku and F. Sato : Plant Cell Physiol. 43 (2002) 1244.が、イオン保持に必須な残基であるN末端15残基が結晶構造に欠けているため、イオン保持機構に関する情報は得られなかった。このPsbPのN末端15残基は、おそらく光化学系Ⅱと結合して初めてイオン保持に必要な構造をとると考えられる。一方でドメインⅡに関しては、分子表面の静電ポテンシャルの解析からその内部に植物種間で保存されたアミノ酸残基からなる塩基性のパッチが存在しており、その反対側の表面は逆に酸性に荷電していた。こうした特徴はシアノバクテリアのPsbVにおいても認められた(図4)。シアノバクテリアの光化学系Ⅱの全体構造から、PsbVは塩基性の表面で膜タンパク質側と相互作用し、酸性の表面でPsbUと相互作用していると考えられる。PsbPとPsbVの、各々の光化学系Ⅱにおける役割は似ている部分があるため、おそらくはPsbPも塩基性パッチで膜タンパク質と、酸性表面でPsbQと相互作用している事が予想される。この予想の妥当性に関しては現在、生化学的な証明を進めている。
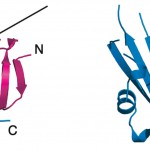
図3 タバコ由来PsbPタンパク質の結晶構造のステレオ図
PsbPの構造はN末端側のドメイン(ピンク色)とC末端側のドメイン(青色)に分けられ、両者の間には矢印で示した位置にプロテアーゼで切断され易いサイトが存在する。
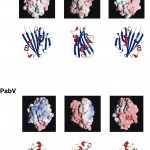
図4 PsbPとPsbVタンパク質の表面電荷と立体構造の比較
PsbPとPsbVの分子表面は、正に荷電した領域(青色)と負に荷電した領域(赤色)が明確に存在しており、その点では共通点がある。しかしながら両者の立体構造は全く異なる。
PsbPとPsbVの類似性が認められる一方で、PsbPと似た構造を示すタンパク質は、PsbVを含めたこれまでに報告されているシアノバクテリアの光化学系Ⅱの結晶構造中には存在せず、それらのタンパク質の中からPsbPが生じたとは考えにくかった。最近、シアノバクテリアにもPsbPと弱い配列相同性を示すタンパク質が存在することが明らかとなり、シアノバクテリアの“PsbP”として扱われている場合がある。一方で、シアノバクテリアの“PsbP”により近いタンパク質が、高等植物にもPsbPとは別に存在していることから、筆者らはこれらPsbPホモログを「PsbP-like protein」として高等植物や緑藻のPsbPとは区別して呼んでいる。これらPsbP-like proteinの役割に関しては、高等植物においてもシアノバクテリアにおいてもよく分かっていない。ごく最近にシアノバクテリアのPsbP-like proteinの遺伝子破壊株の解析結果から、おそらくこのタンパク質も光化学系Ⅱの反応に関与している可能性が、日本植物生理学会年会において報告された[12][12]H. Ohkawa et al. : Plant Cell Physiol. S45 (2004) S80.。そこでは遺伝子破壊株の表現型から、シアノバクテリアのPsbP-like proteinは高等植物のPsbPとはかなり性質や役割が異なることが示唆されていた。今後、そうしたPsbP-like proteinがどのように光化学系Ⅱ反応に関わっているのか、その具体的な光化学系Ⅱにおける結合部位や、化学量論的な結合量が明らかにされる必要がある。
こうした研究の一方で、新規なタンパク質構造が決定された際には、そのタンパク質と似た構造を示すタンパク質を検索することで、そのタンパク質の機能や類縁関係に関する情報が得られる場合が多い。そこで筆者らもPsbPと似た構造をDALIデーターベース上で検索した。驚いた事にPsbPの立体構造は、特にドメインⅡの部分においてRan-GTPaseとの結合タンパク質であるMog1pと似ている事が判明した(図5)。Ran-GTPaseは、結合しているGTPの加水分解を通して、細胞核へのタンパク質の輸送や細胞分裂の制御等にも関わる重要な酵素であり、真核生物に普遍的に存在している。そしてMog1pは、そのRan-GTPaseがGTPと結合した活性化された状態に保つのに重要な役割を果たしている[13][13]M. Oki and T. Nishimoto : Proc. Natl. Acad. Sci. USA. 95 (1998) 15388.。最近、高等植物の光化学系Ⅱにおいても、その反応で中心的な役割を果たすD1タンパク質の分解−再合成過程にGTPが重要な役割を持つ事が報告された。しかも光化学系ⅡにおいてPsbPと直接相互作用していると考えられているPsbOタンパク質にそのGTPが結合している事が報告された[14][14]C. Spetea et al. : Proc. Natl. Acad. Sci. USA. 101 (2004) 1409.。シアノバクテリアでは、そうしたGTPが関わる光化学系Ⅱの代謝制御は報告されていない。これらの事実はPsbPがMog1pと類似した役割を光化学系Ⅱで持っている可能性を示唆しており、高等植物がPsbPを獲得した理由を解明する手がかりになるかもしれない。
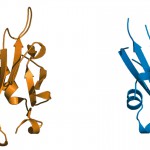
図5 Mog1p(左)とPsbP(右)の立体構造上の類似性
4.おわりに
PsbP分子自体の発見は1980年代になされ、in vitroの実験系における機能解析も多くがその頃になされた。それ以後、その分子研究には大きな進展がなかったが、今回のPsbPの構造解析の結果を踏まえて研究が再加速すると考えている。もちろん、立体構造から推測される事柄は全て実験的に検証される必要がある。特にPsbPとMog1pとの構造上の類似性の持つ生物学的な意味に関しては、現状では全く推論の域を出ていない。しかしながら立体構造が与える示唆は非常に魅力的であり、分子生物学、生化学的な裏付けを得られるかどうか現在検討している段階である。すでに筆者のグループではPsbPの発現をRNA干渉法で抑えた植物個体を確立しており、その解析から何らかの情報が得られると期待している。さらにPsbPの立体構造は、シアノバクテリアなどに存在するPsbP-like protein (PsbPの祖先?)の機能を考える上でも重要であり、それらの機能解析を通じてPsbPの分子進化の全容が明らかにできると考えている。
近年、光化学系Ⅱ複合体研究に関しては構造解析以外にも様々な進展がある。ひとつにはこれまでに知られていなかった低分子サブユニットが多数同定されてきている事であり、これは膜タンパク質の可溶化、精製手法や微量蛋白質の同定技術の進歩によるところが大きい。シアノバクテリアの光化学系ⅡにPsbQに似た蛋白質が確認されたのはその一例である[15][15]Y. Kashino et al. : Biochemistry 41 (2002) 8004.。また表在タンパク質全体の分子進化に関しては、日本の研究グループによって、様々な真核藻類の表在タンパク質の組成と性質を網羅的に解析する研究が行われている。実際に真核藻類の一つである紅藻からは、高等植物と緑藻のPsbQの祖先であることが示唆されるタンパク質が発見された[16][16]H. Ohta et al. : Eur. J. Biochem. 270 (2003) 4156.。今後国内外を問わず、この分野の研究が盛んになる事が予想され、筆者ら今回の研究成果はその一つのきっかけになるのではないかと考えている。こうした一連の研究よって、原核生物から真核生物に至る、酸素発生型光合成生物の進化の流れの大きな一側面が解明されると期待している。
最後に、本研究は筆者が現在所属している京都大学大学院生命科学研究科の佐藤文彦教授と、理化学研究所播磨研究所メンブレンダイナミクス研究チームの加藤博章チームリーダーと中津亨研究員(現在、京都大学大学院薬学研究科教授と助教授を各々兼任)との共同研究による成果である。また実験の全ての過程において、現京都大学大学院薬学研究科博士研究員の清水哲哉博士にお世話になった。さらにX線回折実験ではSPring-8の理研ビームラインのスタッフの皆さんの協力を頂いた。この場を借りて全ての方々に感謝致します。
参考文献
[1]J. De Las Rivas, M. Balsera and J. Barber : Trends in Plant Sci. 9 (2004) 18.
[2]A. Seidler : Biochim. Biophys. Acta 1277 (1996) 35.
[3]A. Zouni et al. : Nature 409 (2001) 739.
[4]N. Kamiya and J.-R. Shen : Proc. Natl. Acad. Sci. USA. 100 (2003) 98.
[5]K. N. Ferreira, T. M. Iverson, K. Maghlaoui, J. Barber and S. Iwata : Science 303 (2004) 1831.
[6]K. -H. Rhee, E. P. Morris, J. Barber and W. Külbrandt : Nature 396 (1998) 283.
[7]V. Calderone et al. : EMBO Rep. 4 (2004) 900.
[8]K. Ifuku, T. Nakatsu, H. Kato and F Sato : EMBO Rep. 5 (2004) 362.
[9]K. Ifuku, T. Nakatsu, H. Kato and F. Sato : Acta Crystallogr. D59 (2003) 1462.
[10]K. Ifuku and F. Sato : Biochim. Biophys. Acta 1546 (2001) 196.
[11]K. Ifuku and F. Sato : Plant Cell Physiol. 43 (2002) 1244.
[12]H. Ohkawa et al. : Plant Cell Physiol. S45 (2004) S80.
[13]M. Oki and T. Nishimoto : Proc. Natl. Acad. Sci. USA. 95 (1998) 15388.
[14]C. Spetea et al. : Proc. Natl. Acad. Sci. USA. 101 (2004) 1409.
[15]Y. Kashino et al. : Biochemistry 41 (2002) 8004.
[16]H. Ohta et al. : Eur. J. Biochem. 270 (2003) 4156.
伊福 健太郎 IFUKU Kentaro
京都大学大学院 生命科学研究科/
理化学研究所 播磨研究所 メンブレンダイナミクス研究グループ
〒606-8502 京都市左京区北白川追分町
TEL:075-753-6381 FAX:075-753-6398
e-mail:ifuku@kais.kyoto-u.ac.jp
略歴:
1996年 京都大学 農学部 農芸化学科卒業
2001年 京都大学大学院 農学研究科 博士課程修了
2001年 博士(農学)取得
2001年 文部科学省未来開拓推進事業 リサーチアソシエイト(京都大学)
2001年〜現在 理化学研究所 播磨研究所 非常勤連係研究員を兼任
2002年〜現在 京都大学大学院 生命科学研究科 助手








