Volume 08, No.6 Pages 413 - 415
3. 最近の研究から/FROM LATEST RESEARCH
tRNA修飾酵素による構造変化したtRNAの認識機構
Alter native Tertiary Structure of tRNA for Recognition by a Post – Transcriptional Modification Enzyme
東京大学大学院 理学系研究科 Graduate School of Science, The University of Tokyo
- Abstract
- Transfer RNA (tRNA) canonically has the clover-leaf secondary structure with the acceptor, D, anticodon, and T arms, which are folded into the L-shaped tertiary structure. To strengthen the L form, post-transcriptional modifications occur on nucleotides buried within the core, but the modification enzymes are paradoxically inaccessible to them in the L form. In this study, we determined the crystal structure of tRNA bound with archaeosine tRNA-guanine transglycosylase, which modifies G15 of the D arm in the core, by using the X-ray diffraction data set collected at BL41XU, SPring-8. The bound tRNA assumes an alternative conformation (“λform”) drastically different from the L form. All of the D arm secondary base pairs and the canonical tertiary interactions are disrupted. Furthermore, a helical structure is reorganized, while the rest of the D arm is single-stranded and protruded. Consequently, the enzyme precisely locates the exposed G15 in the active site, by counting the nucleotide number from G1 to G15 in the λform.
tRNA、rRNA、snRNA等の機能を持つRNAには多種多様の修飾塩基(ACGU以外のヌクレオチド残基)が見出されているが、生命現象におけるその意義が近年ようやく明らかにされつつある。例えば真核生物のrRNAに非常に多く見出されるシュードウリジンなど、多くのRNA修飾の役割は不明であったが、近年の研究からこれらの修飾塩基の一部はRNAの高次構造の形成と安定化に重要であることが明らかになってきた。
RNAの構造を安定化している修飾サイトは、通常、RNAのドメイン間の相互作用など、高次構造的な相互作用(tertiary interaction)に関わっており、その結果、これらの修飾サイトはRNAの高次構造に埋もれて存在している。このようなRNAの高次構造的な相互作用は、比較的小さく単純な構造を持つtRNAにも多く存在している。特に、バリアブル・ループ、 Dアームの一部で構成される、 tRNAのL字型構造の中心部分(コア;図1)には、高次構造的な相互作用が形成されており、多くの修飾塩基がこの「コア」に埋もれている。従って、これらの高次構造を補強する修飾を行うtRNA修飾酵素は、最終的な折り畳みとは異なる構造を持つ、標的サイトが露出した状態のtRNAを認識して修飾を行っていると推測される。
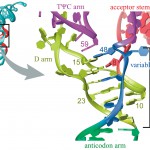
図1 tRNAコアの構造
アーケオシン(7-formamidino-7-deazaguanosine)は、古細菌のtRNAのDループ上15位に存在する修飾塩基である。Dループ上の15位はバリアブル・ループ上の48位と塩基対を形成し、さらにTループ上の59位とスタックしている(図1)。そのため、アーケオシンはこれら3つのループ間の相互作用を補強する![]() のような役割をしていると考えられている。
のような役割をしていると考えられている。
アーケオシンtRNAグアニントランスグリコシラーゼ(ArcTGT)は、このアーケオシンのtRNAへの導入に関わる修飾酵素である。すなわち、ArcTGTはtRNA 15位グアニル酸残基のグアニンとアーケオシン前駆体preQ0塩基を入れ替える反応を触媒する。ArcTGTの標的サイトはtRNAのコア構造に埋もれており(図1)、通常のL字型状態のtRNAでは、ArcTGTは標的サイトにアクセスすることが出来ない。そのためArcTGTは何らかの構造変化を起こしたtRNAに結合し、preQ0塩基を導入していると推測される。tRNAの変異体解析の結果から、ArcTGTによるtRNA認識には正規のL字型の構造は必要ではなく、高次構造の一部が形成できないようなtRNA変異体でも効率よく認識されることが分かっている。また、ArcTGTはtRNAの配列に関係なく、位置特異的にDループ上の15位を認識して修飾を導入すると推測されているが、これらの具体的なメカニズムは不明であった。
我々は、「tRNA修飾酵素による構造変化したtRNAの認識機構」および「蛋白質による位置特異的な核酸認識の機構」を立体構造に基づいて明らかにすることを目的とし、ArcTGT・tRNA複合体のX線結晶構造解析を行い、結晶構造を3.3Å分解能で決定した(図2)。
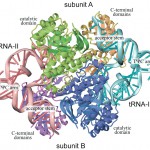
図2 ArcTGT・tRNA複合体の全体構造
複合体の構造解析の結果、ArcTGTは大きく構造変化を起こしたtRNAに結合することが明らかになった(図3)。この構造変化したtRNAは、単に変性してしまうのではなく、修飾の標的部分が露出するようにtRNAの二次構造、三次構造が組み変わった「オルタナティブ」な高次構造(「λ型」構造と呼ぶ)をとっていた。このλ型tRNAでは、 Dアームの構造が完全に破壊されU8位からU22位がtRNA本体から飛び出し、そのうちU8位からU17位がArcTGTに認識されている。一方、正規のL型tRNAではDアームを中心にコア構造が構成されているが、λ型tRNAにおいては、元Dステムの一部とバリアブル・ループにより新たなステム構造と内部ループ構造よりなる「DVヘリックス」が形成され、λ型tRNAのアクセプター・アーム側とアンチコドン・アーム側を接続している。すなわちDVヘリックスは、L型tRNAにおけるコア構造と同様の役割を果たしていると考えられる。
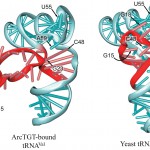
図3 ArcTGTに結合したtRNAの構造変化
さらに、ArcTGTによる配列非特異的かつ位置特異的なtRNA 15位の認識は、tRNAバックボーンの糖と燐酸を1残基ずつ認識することで達成されていた。ArcTGTのC末端ドメインはtRNAのアクセプター・ステムのバックボーンを正確に認識し、飛び出したDアームの付け根の部分U8位を酵素に対して正確に位置づけていた。さらにArcTGTは、U8位からA14位の(一本鎖になったRNAの)バックボーンを1残基ずつ認識し、ポリヌクレオチド鎖の長さを測ることで、正確にG15位を触媒部位に位置づけていた。ArcTGTのC末端ドメインは真核生物・古細菌のRNA修飾酵素に広く見出されるPUAドメインを含んでいる。PUAドメインの機能は未知であったが、今回の構造解析の結果から、位置特異的なtRNA認識において、アクセプター・ステムを酵素に対して正確な位置に結合させるという重要な役割を担っていることが明らかになった。ArcTGTのPUAドメインはrRNA、snRNAのシュードウリジン化に関わるsnoRNPの触媒コンポーネントであるCbf5pのPUAドメインと特に相同性が高く、Cbf5pのPUAドメインも同様にRNAステムを認識することで位置特異的なRNA認識に関わっている可能性が示唆される。
石谷 隆一郎 ISHITANI Ryuichiro
東京大学大学院 理学系研究科 生物化学専攻
〒113-0033 東京都文京区本郷7-3-1
TEL:03-5841-4394 FAX:03-5841-8057
e-mail:ishitani@biochem.s.u-tokyo.ac.jp
横山 茂之 YOKOYAMA Shigeyuki
東京大学大学院 理学系研究科 生物化学専攻
理化学研究所 ゲノム科学総合研究センター 同研究所播磨研究所
〒113-0033 東京都文京区本郷7-3-1
TEL:03-5841-4392 FAX:03-5841-8057
e-mail:yokoyama@biochem.s.u-tokyo.ac.jp








