Volume 08, No.5 Pages 354 - 358
2. 最近の研究から/FROM LATEST RESEARCH
新種タンパク質を作る鍵となる酵素のX線結晶構造解析
Crystal structure of a key enzyme for producing novel proteins
東京大学大学院 理学系研究科 Graduate School of Science, The University of Tokyo
- Abstract
- The archaeal/eukaryotic tyrosyl-tRNA synthetase (TyrRS)–tRNATyr pairs do not cross-react with their bacterial counterparts. This ‘orthogonal’ condition is essential for using the archaeal pair to expand the bacterial genetic code. We solved the 1.95 Å-resolution structure of an archaeal TyrRS–tRNATyr–L-tyrosine complex by using the X-ray diffraction data sets collected at the beamline BL41XU, SPring-8. This structure reveals that this archaeal TyrRS strictly recognizes the C1:G72 base pair, whereas the bacterial TyrRS recognizes the G1:C72 in a different manner using different residues. These diverse tRNA recognition modes form the basis for the orthogonality.
1.はじめに
天然の20種類のアミノ酸でタンパク質を構成する場合、アミノ酸の官能基は限られているために、タンパク質の構造、機能、および化学的性質の拡張には限界があると考えられる。現在、今までにない有用な性質をもつ新種タンパク質をデザインし、生産するうえで、人為的に天然には存在しないアミノ酸(非天然型アミノ酸)をタンパク質に導入する方法が注目されている。われわれは、非天然型アミノ酸を含む新種のタンパク質をアロプロテインと名づけ[1]、アロプロテインの生合成について研究を行ってきた。部位特異的に非天然型アミノ酸が導入されたアロプロテインを効率よく生産するためには、天然の遺伝暗号系を拡張することが不可欠である。アミノアシルtRNA合成酵素(aaRS)は遺伝暗号をかたちづくる酵素であり、それゆえアロプロテインを作る鍵となる酵素と言える。
遺伝暗号の拡張とは、既存の生体系の遺伝暗号をつくるコンポーネントであるaaRSおよび転移RNA(tRNA)とは相互作用しない、新しいaaRSとtRNAを人為的に組み込むことである。概要を図1に示す。非天然型アミノ酸を認識するaaRS(aaRS*)と、アミノ酸をコードしないコドンと対合するtRNA(tRNA*)のペアが、宿主系の他のどのaaRS・tRNAの組とも相互作用しない、「直交した」関係になければならない。すなわち、aaRS*はtRNA*以外をアミノアシル化せず、tRNA*はaaRS*以外にアミノアシル化されないことが不可欠である。そのようなaaRS*・tRNA*ペアを実現するために、他生物種のペアを宿主の系に導入する方法が取られている。現在までに、aaRSのひとつであるチロシルtRNA合成酵素(TyrRS)と、対応するtRNA(tRNATyr)を利用して、遺伝暗号の拡張が行われている。具体的には、古細菌のTyrRS・tRNATyrペアは大腸菌の系と直交し、逆に真正細菌のTyrRSとtRNATyrのペアは哺乳動物細胞の系と直交することを利用して、遺伝暗号の拡張が行われている[2-4]。
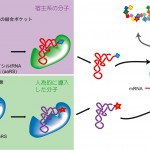
図1 遺伝暗号の拡張とアロプロテインの生産
本研究では、アロプロテインの生産に用いられている古細菌由来チロシルtRNA合成酵素(TyrRS)と、その基質であるtRNA、アミノ酸(チロシン)との三重複合体の立体構造を、SPring-8の放射光を用いたX線結晶構造解析によって、1.95Å分解能という原子レベルの分解能で明らかにした[5]。
2.X線結晶構造解析の実際
X線結晶構造解析には、複合体の良質な単結晶の調製が必須である。この過程が最も困難であった。結晶が得られたらX線回折実験を行うわけであるが、実験室系のX線では強度が弱すぎて検出が困難であった。そのためにSPring-8のビームラインBL41XUに何度か通い、試行錯誤を繰り返した。10ヶ月ほどでポリクリスタルの状態であった結晶を単結晶化することに成功し、1.95Å分解能という良質のデータをBL41XUで測定することができた(図2)。構造解析のためには、回折強度を測定するだけでなく、それぞれの回折の位相を決定しなければならない。本研究では、位相問題の解決のためにセレノメチオニンを含む複合体の結晶を調製し、回折データを収集、処理してSe原子の異常分散効果をもとに位相を決定している。回折強度に対する異常分散効果の寄与を大きくするためにはX線波長をSeのK吸収端に合わせ、回折データを測定しなければならないため、波長変更が可能でかつ強度の強い放射光が必須である。測定を行ったBL41XUでは、吸収端の測定や波長変更の作業が自動化されており、容易に測定することができた(図2)。幸いにして、わずか2回のデータ測定で構造解析に必要な良質のデータを得ることができ、複合体の結晶構造が決定できた。
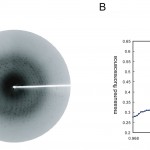
図2 BL41XUで得られた複合体結晶の回折像(A)とセレノメチオニン結晶中のSe原子のX線吸収微細構造(XAFS)(B)
3.複合体の立体構造から明らかになったこと
TyrRSとtRNA、アミノ酸との複合体の立体構造から、TyrRSがどのように特定のtRNAとアミノ酸を厳密に認識しているかが明らかとなった。
tRNAはL字型をしているが、TyrRSはそのL字の両端である、アクセプター部位とアンチコドンを認識している(図3)。それまでの生化学的実験では、アクセプター部位の最初の塩基対、C1 : G72と73番のアデニン(A73)、そしてアンチコドンを認識することは示されていた[6]。本研究はTyrRS側がどのようにそれらの塩基を特異的に認識するかを初めて明らかにした。TyrRS・tRNATyrペアの直交性は古細菌/真核生物と真正細菌の間で、tRNAの1 : 72塩基対が、C : Gペアであるのか、その逆のG : Cペアであるのかという違いに基づいている。古細菌複合体の立体構造と真正細菌複合体の立体構造[7]を比較すると、TyrRS上で認識に寄与している部位は同じであるにもかかわらず、残基の違いによって、塩基の認識に重要な水素結合のつくりかたが大きく異なることが明らかとなった(図4)。古細菌TyrRSでは図の左側のドメインに水素結合供与基が局在するのに対し、真正細菌TyrRSでは水素結合受容基が局在していた。そのことが1 : 72塩基対認識に対して全く反対の特異性を与えているものと考えられる。アンチコドンについても両者では全く異なった残基によって認識されていた。古細菌TyrRSではアンチコドン1文字目のグアニンをアスパラギン酸残基 (Asp286)が水素結合によって強く認識していた(図5)。
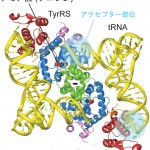
図3 古細菌TyrRS−tRNATyr−チロシン複合体の全体構造
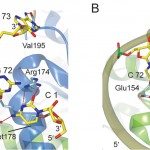
図4 アクセプター部位の比較(A)古細菌(B)真正細菌(文献5より引用)
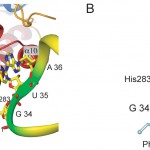
図5 アンチコドンの認識、BはAの簡略化した図。(文献5より引用)
アミノ酸であるチロシンを結合するポケットの構造と、チロシンの認識機構も詳細に明らかとなった (図6)。チロシンの認識については、真正細菌TyrRSと古細菌TyrRSの間でおおむね保存されていたが、細かい残基の配向は異なっていた。真正細菌TyrRSの3-ヨードチロシンという非天然型アミノ酸を認識する変異体は、チロシン結合ポケットに2ヶ所の変異があるが、それらを古細菌TyrRSに移植しても3-ヨードチロシンは認識されなかったことから、このポケットの細かな違いが、いかに非天然型アミノ酸認識に大きく寄与しているかがうかがえる。
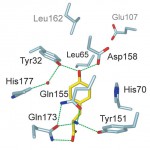
図6 チロシン認識。チロシンは黄色で示す。文献5より引用
4.解明された立体構造の応用性
本研究で得られた構造情報を用いれば、様々なアミノ酸をTyrRSの人工的なデザインによって自由に細菌に取り入れさせることができると期待される。例えば、構造情報に基づいたTyrRSの基質結合部位のコンピュータデザインなどによって、工業的に有用な酵素や、がん特効薬などの医薬品となるタンパク質を自由に開発できるであろう。
現在までに、われわれのグループでは、遺伝暗号の拡張に重要な、tRNAとアミノ酸の特異性の改変を実際に構造に基づいて行っている。先に述べたアンチコドンを認識するアスパラギン酸残基をアルギニン残基に置換することによって、アンチコドンの認識が変化することを示した。この変異体はtRNA*にあたるG34C tRNATyr変異体を野生型より66倍強く認識することが明らかとなった[5]。チロシン認識についても、真正細菌の変異の移植では不可能であった3-ヨードチロシンを結合するような変異体を作り出すことに成功している。今後は、遺伝暗号の拡張とアロプロテインの生産という研究の有用性を広く示すために、創薬などに役立つ有用なアロプロテインを生産できるようなTyrRSの変異体をデザインすることが課題であり、引き続き構造解析と生化学的解析という2つのアプローチから研究を行っている。
参考文献
[1]H. Koide et al. : Biosynthesis of a protein containing a nonprotein amino acid by Escherichia coli: L-2-aminohexanoic acid at position 21 in human epidermal growth factor. Proc. Natl. Acad. Sci. U. S. A. 85 (1988) 6237-41.
[2]L. Wang et al. : Expanding the genetic code of Escherichia coli. Science 292 (2001) 498-500.
[3]D. Kiga et al. : An engineered Escherichia coli tyrosyl-tRNA synthetase for site-specific incorporation of an unnatural amino acid into proteins in eukaryotic translation and its application in a wheat germ cell-free system. Proc. Natl. Acad. Sci. U. S. A. 99 (2002) 9715-20.
[4]K. Sakamoto et al. : Site-specific incorporation of an unnatural amino acid into proteins in mammalian cells. Nucleic Acids Res. 30 (2002) 4692-4699.
[5]T. Kobayashi et al. : Structural basis for orthogonal tRNA specificities of tyrosyl-tRNA synthetases for genetic code expansion. Nat. Struct. Biol. 10 (2003) 425-32.
[6]P. Fechter et al. : Major tyrosine identity determinants in Methanococcus jannaschii and Saccharomyces cerevisiae tRNATyr are conserved but expressed differently. Eur. J. Biochem. 268 (2001) 761-7.
[7]A.Yaremchuk et al. : Class I tyrosyl-tRNA synthetase has a class II mode of cognate tRNA recognition. EMBO J. 21 (2002) 3829-40.
小林 隆嗣 KOBAYASHI Takatsugu
東京大学大学院 理学系研究科
生物化学専攻
〒113-0033
東京都文京区本郷7-3-1
TEL:03-5841-4394
FAX:03-5841-8057
e-mail:koba@biochem.s.u-tokyo.ac.jp
略歴
2000年 東京大学 理学部卒業
2002年 東京大学 理学系研究科 修士課程修了
現在、同博士課程1年。非天然型アミノ酸のタンパク質への導入を研究。
横山 茂之 YOKOYAMA Shigeyuki
東京大学大学院 理学系研究科 生物化学専攻 理化学研究所 ゲノム科学総合研究センター 同研究所播磨研究所
〒113-0033
東京都文京区本郷7-3-1
TEL:03-5841-4392
FAX:03-5841-8057
e-mail:yokoyama@biochem.s.u-tokyo.ac.jp
略歴
1991年 東京大学 理学系研究科教授
1993年 理化学研究所(理研)細胞情報伝達研究室主任研究員兼務
1996年 (2001年まで)科学技術振興事業団ERATO横山情報分子プロジェクト総括責任者兼務
1998年以降、理研ゲノム科学総合研究センター タンパク質構造・機能研究グループ プロジェクトディレクター、理研播磨研究所 ストラクチュローム研究グループリーダー兼務。








