Volume 18, No.3 Pages 208 - 213
1. 最近の研究から/FROM LATEST RESEARCH
S-SAD法による毒素原性大腸菌(ETEC)由来 線毛構成蛋白質CofAの立体構造決定
Structure of the CFA/III major pilin subunit CofA from human enterotoxigenic Escherichia coli determined at 0.90 Å resolution by sulfur-SAD phasing
[1]大阪大学大学院 薬学研究科 Department of Pharmaceutical Sciences, Osaka University、[2]大阪大学 微生物病研究所 Research Institute for Microbial Diseases, Osaka University
- Abstract
- 毒素原性大腸菌(ETEC)はその病原性を発揮するために、菌体表面上に線毛などを形成し、腸管上皮に定着する必要がある。CofAは、ETECの腸管への定着に関わるCFA/IIIと呼ばれる定着因子の主要線毛構成蛋白質である。本研究において、我々はSPring-8のビームラインBL38B1を使用した放射光実験を行い、蛋白質に内在する硫黄原子の異常分散効果を利用したS-SAD法を適用することで、CofAの結晶構造を0.9 Åの高分解能で解明した。さらに、結晶構造解析で得られたCofAの立体構造情報を基に、分子動力学シミュレーション法を用いてCFA/III線毛モデルの構築を行い、その特徴的な表面構造を明らかにした[1][1] Fukakusa, S., Kawahara, K., Nakamura, S., Iwashita, T., Baba, S. et al.: Structure of the CFA/III major Pilin subunit CofA from human enterotoxigenic Escherichia coli determined at 0.90 rsolution by sulfur-SAD phasing. Acta Cryst. D68 (2012) 1418-1429.。
1.はじめに
発展途上国における旅行者や乳幼児の間で深刻な問題となっている毒素原性大腸菌(Enterotoxigenic Escherichia coli: ETEC)は、飲食物を介してヒトの腸管内に到達、増殖し、産生する腸管毒素エンテロトキシンによってコレラ様の下痢を引き起こす[2][2] Qadri, F., Svennerholm, A. M., Faruque, A. S., Sack, R. B.: Enterotoxigenic Escherichia coli in Developing Countries: Epidemiology, Microbiology, Clinical Features, Treatment, and Prevention. Clin Microbiol Rev 18 (2005) 465-483.。ETECの病原性発現には腸上皮細胞への定着が必須であり、腸管定着因子として、現在までにCFA/I、CFA/II、CFA/IIIなどの因子群が発見され[3][3] Gaastra, W. & Svennerholm, A. M.: Colonization factors of human enterotoxigenic Escherichia coli (ETEC). Trends Microbiol 4 (1996) 444-452.、それぞれの作用機序の解明と定着阻害法の開発が盛んに進められている。
ETECの腸管定着因子の多くは、菌体表面上に発現される蛋白質の性質に依存した様々な定着機構を有しており、それらは線毛を形成するものと非線毛性のものに大別される[3][3] Gaastra, W. & Svennerholm, A. M.: Colonization factors of human enterotoxigenic Escherichia coli (ETEC). Trends Microbiol 4 (1996) 444-452.。我々が研究対象としているCFA/IIIは、線毛の形態や構成蛋白質のアミノ酸配列情報からIV型に分類される線毛を形成する[4][4] Dhakal, B. K., Bower, J. M., & Mulvey, M. A. : Pili, Fimbriae. Encyclopedia of Microbiology, edited by M. Schaechter (2009) 470-489. Oxford: Elsevier.。このIV型線毛は、その主要線毛構成蛋白質の分子量の違いなどからさらにIVa型線毛とIVb型線毛に分類され、CFA/III線毛はIVb型線毛に属している[5][5] Taniguchi, T., Fujino, Y., Yamamoto, K., Miwatani, T., Honda, T.: Sequencing of the gene encoding the major pilin of pilus colonization factor antigen III (CFA/III) of human enterotoxigenic Escherichia coli and evidence that CFA/III is related to type IV pili. Infect. Immun. 63 (1995) 724-728.。これまでの研究からCFA/IIIを構成する14個の遺伝子群からなるcofオペロンの内、線毛の主要な構成蛋白質を発現する遺伝子としてcofAが同定されており、CFA/III線毛の形成の際には、その主要線毛構成蛋白質CofAのN末端側に存在するシグナル配列がプレピリンペプチダーゼ(CofP)によって切断される必要があることが明らかになっている。
IVb型線毛に分類される線毛には、CFA/III線毛の他に、コレラ菌のTCP線毛や、腸管病原性大腸菌に発現するBFP線毛が知られており、それぞれ線毛の構造や形成機構、そして定着機構に関して詳細な研究例があるが[6][6] Craig, L., Pique, M. E., Tainer, J. A.: Type IV pilus structure and bacterial pathogenicity. Nature Rev. Microbiol. 2 (2004) 363-378.、CFA/III線毛に関しては腸管側の受容体の同定も含めて未だ機能解析が進んでいない。そのため、本研究では、X線結晶構造解析を用いてCFA/III線毛の主要な構成蛋白質であるCofAの立体構造を明らかにし、得られる構造を基にして、線毛のフィラメントモデルを構築することで、ETECの腸管付着に関わるCFA/III線毛の性質を明らかにすることを試みた。
2.硫黄原子を用いたS-SAD法によるCofAの結晶構造決定
一般的に、IV型線毛の構成蛋白質は、N末端側に配列相同性の高い疎水性領域を保持しており難溶性である。そこで、溶解度を改善する目的で、他のIVb型線毛構成蛋白質の配列情報を参考に、N末端側の疎水性部分(約28残基)を除いたCofAの大腸菌を用いた大量発現系を構築し、精製及び結晶化を行った。蒸気拡散法により得られたCofAの単結晶は、SPring-8のビームラインBL38B1で回折実験を行ったところ、測定波長0.9 Åにおいて最高分解能0.9 Åの極めて良質な回折像を示した。回折実験に関わる統計値を表1に示している。
| データセット | Native | S-SAD |
|---|---|---|
| 結晶学的データとデータ測定に関わる統計値 | ||
| ビームライン | BL38B1 (SPring-8) | BL38B1 (SPring-8) |
| 検出器 | ADSC Quantum 315 | ADSC Quantum 315 |
| 振動角(°) | 1 | 1 |
| X線の波長 (Å) | 0.9 | 1.5 |
| 分解能(最外殻)(Å) | 30.86-0.90 (0.95–0.90) | 47.64-1.38 (1.42–1.38) |
| 独立な反射数 | 91809 | 51931 |
| 多重度(最外殻) | 3.6 (3.5) | 6.8 (4.7) |
| 反射の完全性(最外殻) | 96.4 (94.9) | 99.5 (99.6) |
| 結晶空間群 | P21 | P21 |
| 格子定数 | ||
| a, b, c (Å) | a= 33.82, b= 47.71, c= 42.45 | a= 33.82, b= 47.64, c= 42.53 |
| β (°) | 107.52 | 107.57 |
| Rmerge (最外殻)(%) | 0.050 (0.170) | 0.041 (0.226) |
| <I/σ(I)>(最外殻) | 18.6 (7.1) | 32.1 (6.3) |
| 位相決定に関わる統計値 | ||
| 硫黄原子のサイト数 | 5 | |
| Figure of merit(FOM) | 0.833 | |
| 精密化に関わる統計値 | ||
| 分解能(最外殻) (Å) | 30.86-0.90 (0.91-0.90) | |
| 反射数 | 91733 | |
| Rwork(最外殻)(%) | 0.120 (0.146) | |
| Rfree(最外殻)(%) | 0.130 (0.158) | |
| 温度因子(Å2) | 4.5 | |
| 原子数(非対称単位) | ||
| 蛋白質 | 3,189 | |
| 水 | 237 | |
| R.m.s.d. | ||
| 結合距離 (Å) | 0.01 | |
| 結合角(°) | 1.54 | |
| ラマチャンドランプロット(%) | ||
| 推奨範囲内 | 97.7 | |
| 許容範囲内 | 2.3 | |
構造決定にあたり、まず立体構造が現在報告されているIVb型線毛構成蛋白質の中で最も配列相同性の高い(相同性約28%)TCP線毛の主要構成蛋白質であるTcpA(PDBID: 1oqw)の立体構造を用いた分子置換法を試みたが、有意な解を得ることはできなかった。そのため、初期位相の決定は、蛋白質内に存在する硫黄原子を用いた単波長異常分散(S-SAD)法の適用を試みた。
今回、結晶化に用いたCofAは、全体で182残基からなる分子中に硫黄原子が5個存在し、予想される異常分散強度(<∆F±>/
[9] Watanabe, N.: From phasing to structure refinement in-house: Cr/Cu dual wavelength system and an loopless free crystal mounting method. Acta Cryst. D62 (2006) 891-896.。この点に関して、HASYLAB(ドイツ)など海外の放射光施設では3.0 Å以上の波長を利用できる構造生物学用のビームラインの開発も進められているが、長波長での測定ほど異常分散効果は大きくなるものの、空気や溶媒の吸収効果による影響が大きくなり、測定誤差が大きくなる問題も抱えている。さらに、測定の多重度を上げ、シグナルノイズ比を改善するために放射光を照射し続けることにより生じる結晶の損傷も無視できない問題として挙げられる。これらの様々な要因がS-SAD法の一般的な普及を難しくしているといえる。
これまでに、SPring-8のBL38B1では、X線ビームの安定性やフラックスの向上、コリメータやビームストップなどの最適化によるバックグランド信号の低減に加えて、長波長での測定において更に高分解能の回折データ収集が可能となる従来に比べて面積の大きい検出器(Quantum 315 CCD検出器、ADSC社製)の使用など、精密な測定が必要とされるS-SAD法に適した実験環境の整備が行われてきた。今回、この測定環境において、CofAの単結晶の回折データを0.9 Åから1.7 Åまで波長を変えて収集したところ、波長が1.5 Åの場合に測定された回折データが有意な異常分散差強度を与えることを確認することができた。すなわち、精密に調整された放射光のビームラインを利用し、適切な波長を選ぶことで、S-SAD用のデータセットの取得が充分可能であることが示された(図1)。得られたデータセットについてプログラムSHELXDによる重原子位置の探索を行ったところ、CofA中の硫黄原子5個すべてが発見された。しかしながら、続けてプログラムSHELXEによる位相決定及び数種のプログラムを用いた電子密度の改良を行ったところ、解釈可能な電子密度マップを得ることができなかった。これは結晶の溶媒含有率が29%と低く、溶媒平滑化などによる位相改良が難しいことが原因と考えられた。そこで位相誤差を減少させるため、プログラムSHELXEによる電子密度の改良時に0.9 Åの高分解能データによる分解能の拡張を行ったところ解釈可能な電子密度マップを得ることに成功した(図2)。
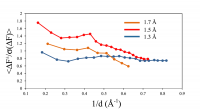
図1 SPring-8 BL38B1で実施した各測定波長で得られた回折データの異常分散差強度の分解能の逆数に対するプロット

図2 プログラムSHELXEによる位相改良後の電子密度マップ(2Fo-Fc)
(a)S-SAD法による初期位相決定直後、
(b)高分解能の回折データにより分解能を拡張した後に得られる電子密度マップ。それぞれのマップのTrp117付近の領域を拡大して示す(1.5 σ)。
得られたCofAの結晶構造は5本のαへリックス、8本のβストランド、そして2本の310へリックスから構成され、Cys132 - Cys196間にジスルフィド結合を形成していた。また、他のIV型線毛構成蛋白質と同様に、全体としてαβフォールドを形成していた[5][5] Taniguchi, T., Fujino, Y., Yamamoto, K., Miwatani, T., Honda, T.: Sequencing of the gene encoding the major pilin of pilus colonization factor antigen III (CFA/III) of human enterotoxigenic Escherichia coli and evidence that CFA/III is related to type IV pili. Infect. Immun. 63 (1995) 724-728.。CofAの構造は、主に構造コア、αβループ領域、D領域と呼ばれる3領域に分けることができ、中心部の疎水性相互作用により安定化されている構造コアを取り囲むように、αβループ領域、D領域が存在し、構造表面に露出していた。この為、これらの表面に露出している領域は、線毛の他のサブユニットとの相互作用に深く関わる領域であることが示唆された。
3.CFA/III線毛モデルの構築
CofAの類似構造を探索した結果、アミノ酸配列の相同性は低いもののCofAの立体構造はコレラ菌由来のTcpAに類似しており、構造コア部分で重ね合わせた場合のCα原子間の根二乗平均距離は約2.16 Åであった(図3)。IVb型線毛において、TcpAはもっともよく研究されている蛋白質の一つであり、X線結晶構造解析、クライオ電子顕微鏡、水素/重水素交換質量分析の結果から構築されたTcpAのフィラメントモデルが提案されている[10][10] Li, J., Lim, M. S., Li, S., Brock, M., Pique, M. E. et al.: Vibrio cholerae toxin-coregulated pilus structure analyzed by hydrogen/deuterium exchange mass spectrometry. Structure 16 (2008) 137-148.。報告されたフィラメントモデルにおいて、N末端の疎水性領域間、そしてαβループ領域とD領域の間に形成される2種類のサブユニット間相互作用が線毛の安定性に重要であり、Met1, Glu5, Arg26, Leu76, Glu83の5残基が安定性の維持に必須であることが変異実験の結果と併せて明らかにされている。CofAにおいてもN末端の疎水性領域にMet1, Glu5, Arg26が存在し、TcpAのLeu76およびGlu83と立体構造上で同じ位置に、対応する残基 (図中でTcpAのLeu76がCofAのLeu86に相当し、TcpAのGlu83がCofAのGlu93に相当する)が存在する(図3)。
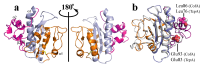
図3.0.9 Åの分解能で決定されたCofAの結晶構造
CofAの全体構造(a)とCofAとTcpAの立体構造の重ね合わせ結果(b)、CofAの全体構造において、構造コア部分を青色、αβループ領域はマゼンタ色、D領域はオレンジ色、そして分子内に存在するジスルフィド結合は黄色で表示している。
そこで、これらの知見を参考に、TCP線毛のモデルとしてすでに報告されているTcpAフィラメントモデル(PDBID:1orq)の各サブユニットの構造に、結晶構造から得られたCofAの構造を重ね合わせることでCofAフィラメントモデルの構築を行った。また、結晶化の際の溶解度向上のために除いた、N末端部分の28残基からなる疎水性領域は、IVa型線毛に関して以前に全長の立体構造が報告されているPAK Pilinの結晶構造(PDBID:1oqw)のN末端のαへリックスを基に構築した。この様に作製したモデルの妥当性を検証するためにサブユニット間の相互作用領域における原子間距離の評価をおこなったところ多数の原子が衝突することが明らかになり、これは TcpAとCofAの溶媒露出領域(αβループ領域とD領域)の構造の違いに起因することが明らかであった。そこで、プログラムMODELLERによるシミュレーテッドアニーリング法によるエネルギー最少化と分子動力学シミュレーションプログラムGROMACSを用いたエネルギー最少化を併用し、モデル構造の最適化を行った。最終的に構築されたCofAのフィラメントモデルのサブユニット間相互作用について評価を行ったところ原子の明らかな衝突は見られなかった。
得られたCofAのフィラメントモデルは1巻き(360˚)当たり6個のサブユニットが左巻き螺旋状に並んだものが3本束なった形のいわゆる左巻き3-startへリックス構造を形成しており、一周当たり18個(6個×3本)のCofAサブユニットから構成されていた(図4)。またその直径は約80 Åであり、以前に電子顕微鏡像から得られたCFA/III線毛の直径の値(約70 Å)とほぼ同等であった[11][11] Taniguchi, T., Akeda, Y., Haba, A., Yasuda, Y., Yamamoto, K. et al.: Gene cluster for assembly of pilus colonization factor antigen III of enterotoxigenic Escherichia coli. Infect Immun. 69 (2001) 5864-5873. 。TCP線毛のフィラメント形成に重要であった前述の残基について観察したところ、N末端部においてはMet1のN末端窒素とGlu5の側鎖間に相互作用が見られ、また、αβループ領域のGlu93はα1へリックスに存在するArg26と相互作用していた。同様に、αβループ領域のLeu86はD領域のAla189, Ile192と疎水性相互作用していることも明らかとなり、線毛形成の際におけるこれらのアミノ酸残基の重要性がモデル構造から示唆された(図4)。
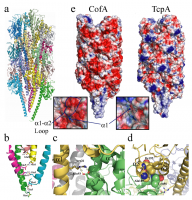
図4.CofAの結晶構造を基に構築したCFA/III線毛モデル
CFA/III線毛モデルの側面(a)、N末端部分におけるMet1とGlu5の相互作用(b)、αβループ領域のGlu93とα1へリックスのArg26の相互作用(c)、αβループ領域のLeu86とD領域のAla189、Ile192の疎水性相互作用(d)、CFA/III線毛モデルとTCP線毛モデルの表面構造(e)、表面構造において正電荷を青色、負電荷領域を赤色で示す。CFA/III線毛とTCP線毛のαβループ領域の拡大図を併せて示す。
4.CFA/III線毛モデルの表面構造
今回我々が構築したCofAの結晶構造情報に基づくCFA/III線毛のモデルと以前に報告されたTCP線毛のモデルの表面構造を比較することでCFA/III線毛の二つの特徴が明らかとなった。双方の線毛表面の静電ポテンシャル分布をプログラムAPBS を用いて計算し、比較したところ、TcpAフィラメントモデルにおいては線毛表面において、正、負に帯電する二つの領域が存在するのに対し、CofAフィラメントモデルの表面は全体的に負に帯電する領域に覆われていた(図4)。このことから、しばしば線毛凝集が確認されるTCP線毛に比べて、CFA/III線毛は線毛同士が静電的反発によって凝集しにくくなっていると推察できる。また、TcpAフィラメントモデルにおいてはサブユニット間の隙間において、フィラメント内部に存在するN末端側のαへリックスの一部が露出しており、この部分がTcpAのD領域と相互作用することによりTCP線毛同士の相互作用に重要な役割を果たしていると考えられているが、CofAフィラメントモデルにおいてはこの隙間がα1-α2へリックス間に存在するTcpAの立体構造には存在しない23残基からなる長いループ構造で埋められており(図4)、N末端側のαへリックスが他のサブユニットと相互作用することを不可能にしている。すなわち、同じIVb型線毛に属しながらもCFA/III線毛とTCP線毛とは、その線毛の機能が大きく異なる可能性がフィラメントモデルの比較により明らかとなった。
この様な、CFA/III線毛の表面構造の特徴は、これまでに報告された腸管病原性大腸菌に発現するBFP線毛やサルモネラ菌が発現する線毛(主要構成蛋白質はPilS)などの他のIVb型線毛とも異なっており、際立った特徴といえる。そのため、現在は、本研究で構築した線毛モデルが提示する特徴的な表面構造をターゲットとした定着阻害物質の探索や、当該構造を抗原として認識する抗体の開発を通して、CFA/III線毛の腸管定着機構の更なる理解と効果的なワクチンの開発へ向けた研究を行っている。
5.おわりに
近年、放射光における実験環境の整備が飛躍的に進み、従来まで測定が困難であった蛋白質中に含まれる硫黄原子の微弱な異常分散効果を精度よく測定することが可能となり始めた。この様な精密測定へ向けた実験環境の高度化に加えて、適した測定波長を選定することにより、我々は放射光施設SPring-8のBL38B1における実験において、分子中の硫黄原子の異常分散効果が極めて微弱であると予想されたETEC由来の線毛構成蛋白質CofAの立体構造を、S-SAD法を用いて初めて決定することが出来た。S-SAD法は、重原子の結晶への導入や、蛋白質のセレノメチオニン誘導体を調製する必要がなく、蛋白質中に内在する硫黄原子の異常分散効果のみで立体構造決定が可能である強みがある。そのため、今後、単に新規構造を決定する手法としてだけではなく、蛋白質結晶構造解析のハイスループット化へ向けて、益々注目を集める手法になると予想される。
謝辞
本研究は、著者らと大阪大学薬学研究科の岩下丘樹氏、大阪大学大学院工学研究科の小林祐次特任教授、高輝度光科学研究センターの馬場清喜博士、大阪大学微生物病研究所の西村光広博士、本田武司名誉教授、飯田哲也特任教授、ならびに谷口暢特任准教授との共同研究の成果である。また本研究は文部科学省「新興・再興感染症研究拠点形成プログラム」から一部支援を受け、大型放射光施設SPring-8での測定(課題番号:2011B1259)で行われた。
参考文献
[1] Fukakusa, S., Kawahara, K., Nakamura, S., Iwashita, T., Baba, S. et al.: Structure of the CFA/III major Pilin subunit CofA from human enterotoxigenic Escherichia coli determined at 0.90 rsolution by sulfur-SAD phasing. Acta Cryst. D68 (2012) 1418-1429.
[2] Qadri, F., Svennerholm, A. M., Faruque, A. S., Sack, R. B.: Enterotoxigenic Escherichia coli in Developing Countries: Epidemiology, Microbiology, Clinical Features, Treatment, and Prevention. Clin Microbiol Rev 18 (2005) 465-483.
[3] Gaastra, W. & Svennerholm, A. M.: Colonization factors of human enterotoxigenic Escherichia coli (ETEC). Trends Microbiol 4 (1996) 444-452.
[4] Dhakal, B. K., Bower, J. M., & Mulvey, M. A. : Pili, Fimbriae. Encyclopedia of Microbiology, edited by M. Schaechter (2009) 470-489. Oxford: Elsevier.
[5] Taniguchi, T., Fujino, Y., Yamamoto, K., Miwatani, T., Honda, T.: Sequencing of the gene encoding the major pilin of pilus colonization factor antigen III (CFA/III) of human enterotoxigenic Escherichia coli and evidence that CFA/III is related to type IV pili. Infect. Immun. 63 (1995) 724-728.
[6] Craig, L., Pique, M. E., Tainer, J. A.: Type IV pilus structure and bacterial pathogenicity. Nature Rev. Microbiol. 2 (2004) 363-378.
[7] Wang, J., Dauter, M., Dauter, Z.: What can be done with a good crystal and an accurate beamline?. Acta Cryst. D62 (2006) 1475-1483.
[8] Liu, Q., Dahmane, T., Zhang, Z., Assur, Z., Brasch, J. et al.: Structures from Anomalous Diffraction of Native Biological Macromolecules. Science 336 (2012) 1033-1037.
[9] Watanabe, N.: From phasing to structure refinement in-house: Cr/Cu dual wavelength system and an loopless free crystal mounting method. Acta Cryst. D62 (2006) 891-896.
[10] Li, J., Lim, M. S., Li, S., Brock, M., Pique, M. E. et al.: Vibrio cholerae toxin-coregulated pilus structure analyzed by hydrogen/deuterium exchange mass spectrometry. Structure 16 (2008) 137-148.
[11] Taniguchi, T., Akeda, Y., Haba, A., Yasuda, Y., Yamamoto, K. et al.: Gene cluster for assembly of pilus colonization factor antigen III of enterotoxigenic Escherichia coli. Infect Immun. 69 (2001) 5864-5873.
大阪大学大学院 薬学研究科 高分子化学分野
〒565-0871 大阪府吹田市山田丘1-6
TEL: 06-6879-8222
e-mail: kkkazuki@phs.osaka-u.ac.jp
大阪大学大学院 薬学研究科 高分子化学分野
〒565-0871 大阪府吹田市山田丘1-6
TEL: 06-6879-8222
e-mail: s-fukakusa@phs.osaka-u.ac.jp
大阪大学大学院 薬学研究科 高分子化学分野
〒565-0871 大阪府吹田市山田丘1-6
TEL: 06-6879-8222
e-mail: ohkubo@phs.osaka-u.ac.jp
大阪大学 微生物病研究所 附属遺伝情報実験センター
ゲノム情報解析分野
〒565-0871 大阪府吹田市山田丘3-1
TEL: 06-6879-8365
e-mail: nshota@gen-info.osaka-u.ac.jp








