Volume6 No.2
SPring-8 Section A: Scientific Research Report
IMP-6メタロ-β-ラクタマーゼの結晶構造解析
Crystallographic Analysis of IMP-6 Metallo-β-Lactamase
奈良県立医科大学
Nara Medical University
- Abstract
-
IMP-6メタロ-β-ラクタマーゼ(IMP: imipenemase、イミペネム分解酵素)は、IMP-1からS196Gのアミノ酸置換により生じた酵素である。IMP-1ではイミペネム、メロペネムに対する活性が同程度であるのに対し、IMP-6はメロペネムに対する活性が、イミペネムに対する活性の約7倍に増加する。本研究では、IMP-6の基質特異性が変化した原因を探るために、X線結晶構造解析を試みた。その結果、分解能は不十分ながら、基質との疎水性相互作用の変化が原因であることが示唆された。
Keywords: β-ラクタマーゼ、基質特異性、結晶構造解析
背景と研究目的:
β-ラクタム系抗菌薬は、その安全性や有用性から臨床の現場で最も頻繁に使用されている抗菌薬である。ところが、使用の拡大が選択圧となり、細菌の抗菌薬耐性の機序の高速な進化をもたらしている。その最も重要なメカニズムが β-ラクタマーゼの産生である。β-ラクタマーゼは β-ラクタム系抗菌薬の β-ラクタム環を加水分解し、不活化する酵素である。β-ラクタマーゼはA~Dの4種類に分類される[1]。クラスA, C, Dは活性中心にセリン残基を持つ酵素であるが、クラス Bは活性中心に1、あるいは2個の亜鉛イオンを有する金属酵素であり、メタロ-β-ラクタマーゼ(MBLs)と称される。MBLsはペニシリン、第1~第4世代セフェム、カルバペネムに対する加水分解活性を持ち、一般の β-ラクタマーゼ阻害剤に対する耐性を有し、遺伝子の平行移動を起こしやすいことから、特に臨床的な興味を持たれている[2]。MBLsは構造と、亜鉛イオンに対するリガンドのアミノ酸配列により、サブクラス B1、B2、B3 に分けられている[3]。
IMP-1はサブクラスB1に属し、1994年に日本において Serratia marcescens と Pseudomonas aeruginosa による産生が初めて認められた酵素である[4]。以降、IMP型MBLsは2018年2月までに71種の亜種のDNA配列が登録されている。IMP-1はカルバペネム系抗菌薬の内、臨床でよく使用されるイミペネム(kcat / Km = 1.2 µM-1s-1)とメロペネム(kcat / Km = 1.5 µM-1s-1)に対してほぼ同等の加水分解活性を示す[4]。一方、2001年に日本で発見されたIMP-6はIMP-1の Ser196 が Gly に置換された酵素であり、イミペネムに対する kcat / Km が 0.61 µM-1s-1 であるのに対し、メロペネムに対する kcat / Km が 4.2 µM-1s-1 と高く、メロペネムに対する加水分解活性が、イミペネムに対する活性の約7倍に増加している[5]。S196G のアミノ酸置換により、IMP-6の構造がIMP-1からどのように変化し、基質特異性が変化するのかを探るために、IMP-6を結晶化し、X線結晶構造解析を試みた。
実験:
発現ベクターpET28aにIMP-6遺伝子をクローニングし、大腸菌BL21(DE3)を形質転換した株を用いてIMP-6を高発現させた。培養した形質転換体を超音波破砕機を用いて破砕、遠心分離した上清を陽イオン交換カラムクロマトグラフィー(Macroprep High S Support (BIO-RAD)、及びCM-Toyopearl 650S (Tosoh))に添加し、電気泳動的に単一にまで精製した。精製したIMP-6のN末端アミノ酸配列を決定したところ、報告されているIMP-6のアミノ酸配列と一致した[5]。精製タンパク質を用いて市販の結晶化溶液キットを用いたスクリーニングを行った。最終的にハンギングドロップ法を用い、5 mM HEPES pH 7.3 中でタンパク質濃度 20 mg/mL の溶液と、結晶化母液として 0.13 M ammonium sulfate, 0.1 M MES pH 6.0, 24 % polyethylene glycol monomethyl ether 5000 を等量混合して 15℃ で静置することにより、0.1×0.1×0.1 mm の立方体状結晶を得た。
得られた結晶を 20 %PEG200 を含む結晶化母液に浸漬し、BL44XUにおいて、100 K の窒素気流下、露光時間 0.5秒、振動角 1º 、波長 0.9000 Å、カメラ長 400 mm で回折データを収集した。検出器はMAR300HE(Rayonix)を使用した。その結果、分解能 3.00 Å までの回折データを収集した。得られた回折データは、プログラムHKL2000で処理した。
IMP-6の構造は、IMP-1(PDB code: 1DD6)の構造を用い、CCP4 suiteのプログラムMolrepを用いて分子置換法により求めた。得られた初期解をCCP4 suiteのプログラムREFMAC 5.5により、分解能 50.00 – 3.00 Å の範囲で精密化した。
結果および考察:
複数個の結晶について回折データの測定を行ったが、いずれも分解能 3.00 Å を越える良好なデータは得られなかった。最もよいデータについての統計値を表1に示した。結晶の空間群はP212121、格子定数は a=49.09±0.01 Å、b=78.55±0.01 Å、c=261.51±0.01 Å であり、非対称単位中には4分子のIMP-6が存在した。
表1.回折強度データ統計値
| Resolution (Å) | 50.00+3.00 (3.05-3.00) |
| Observed reflections | 152777 (7305) |
| Unique reflections | 21905 (1790) |
| Averaged I /σ(I) | 6.7 (2.2) |
| Completeness(%) | 99.9 (100.0) |
| Redundancy | 7.0 (7.0) |
| Rmerge# (%) | 21.5 (75.4) |
#Rmerge = ΣhklΣi|I(hkl)i-‹I(hkl)›|/ΣhklI(hkl).
分子置換法により求めた電子密度図は、シグナル配列が除かれた、228アミノ酸からなる成熟型IMP-6の内、Leu4-Glu219の主鎖すべてと8割程度の側鎖について帰属が可能であった。しかしながら分解能が不十分であったため、活性中心に存在するはずの2原子の Zn2+ イオンと水分子は観測できなかった。また、Rwork / Rfree は、0.249/ 0.369 までしか精密化できなかった。
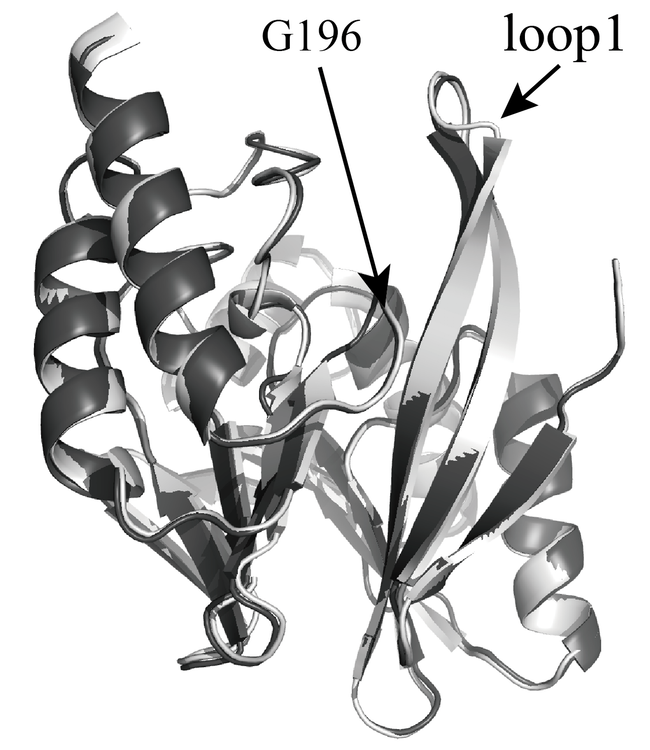
図1にIMP-1(1DD6)とIMP-6の主鎖の構造を重ね合わせた図を示した。両者の構造は非常に似通っており、主鎖全体のRMSDは 0.430 Å であった。アミノ酸配列において、IMP-1とIMP-6が異なるのは Gly196(IMP-6)のみである。この位置は、活性中心を構成する2原子の亜鉛イオンのうち、Zn2 に配位しているHis197のN末側にある。このアミノ酸残基の Cα における変位は僅かに 0.27 Å であった。ところが、アミノ酸置換が生じた部位と反対側のN末端ドメインにおいて、β-sheet2 のC末端部分に続くloop1領域(His37 – Pro50)では最大 1.39 Å(Glu41)、またPhe69でも 1.00 Å の変位が観測された。前者は疎水性相互作用により基質と相互作用する領域、後者は基質と相互作用する疎水性ポケットを形成するアミノ酸残基の1つとして知られている[6, 7]。従って、IMP-6におけるメロペネムに対する活性の上昇は、Ser196→Gly のアミノ酸置換による、基質との疎水性相互作用の変化によるものであることが示唆された。また、アミノ酸置換の位置と、構造が大きく変化した位置が異なることから、IMP型メタロ-β-ラクタマーゼは潜在的な構造の柔軟性があり、僅かなアミノ酸置換が導入されることによって構造が変化し、活性に影響を与えることが予想された。
図1. IMP-1 (dark gray) と IMP-6 (pale gray) の構造比較
今後の課題:
IMP-6の構造を原子レベルで解析するためには、より分解能が高く、良好な回折データを収集する必要がある。しかしながら、実験期間中には良好な結晶を得ることができなかった上、結晶化の再現ができなくなった。従って、一旦構造解析を中断し、結晶化条件の再検索から実験をやり直すこととした。
参考文献:
[1] R. P. Amber, Philos. Trans. R. Soc. Lond. B Biol. Sci., 289, 321-331 (1980).
[2] N. Laraki et al., Antimicrobial Agents Chemother., 43, 902-906 (1999).
[3] M. Galleni et al., Antimicrobial Agents Chemother., 45, 660-663 (2001).
[4] E. Osano et al., Antimicrobial Agents Chemother., 38, 71-78 (1994).
[5] H. Yano et al., Antimicrobial Agents Chemother., 45, 1343-1348 (2001).
[6] N. O. Concha et al., Biochemistry, 39, 4288-4298 (2000).
[7] Y. Yamaguchi et al., Biol. Pharm. Bull., 38, 96-101 (2015).
ⒸJASRI
(Received: March 23, 2018; Early edition: June 22, 2018; Accepted: July 3, 2018; Published: August 16, 2018)