Volume6 No.1
SPring-8 Section A: Scientific Research Report
小胞体分子シャペロンER-60によるペプチド結合様式
Peptide Binding Mechanism of Endoplasmic Reticulum Chaperone ER-60
a京都大学,b丸和栄養食品株式会社
aKyoto University, bMaruwa Foods and Biosciences, Inc.
- Abstract
-
脳神経細胞の小胞体に高発現する分子シャペロンER-60はアルツハイマー病の原因と考えられているアミロイドβペプチドの線維化を阻害する。生化学的な解析から、ER-60のbb’ 領域がアミロイドβペプチドと結合することで線維化を阻害することが明らかとなっている。本研究では、結晶構造解析によりbb’ の境界領域にアミロイドβペプチドが一定の決まった配位ではなくフレキシブルな結合様式で結合することを明らかにした。
キーワード: アミロイドβペプチド、小胞体、分子シャペロン、X線結晶構造解析
背景と研究目的:
動物細胞の小胞体には約20種類のPDIファミリータンパク質が存在し、粗面小胞体で生合成された新生タンパク質のジスルフィド結合形成を伴う高次構造形成(フォールディング)や品質管理を担っている。個々のPDIファミリータンパク質が特異的な生理的役割を分担すると予想されているが、そのうちの1つであるER-60はタンパク質生合成の盛んな臓器に広く発現する多機能生のタンパク質であり、レクチン型分子シャペロン(カルネキシン及びカルレチキュリン)との複合体形成を介して糖タンパク質のフォールディングに特に重要な役割を果たしていることが明らかにされている[1-4]。さらに、ER-60は肝臓細胞からのリポタンパク質 (VLDL) の分泌量の制御 [5-8]、抗原提示におけるMHC class Iと抗原ペプチドとの複合体形成作用 [9]、及び小胞体のカルシウムホメオスタシスの調節作用 [10]などが報告されている。疾病との関係では2型糖尿病を引き起こすインスリン抵抗性に伴うER-60の発現低下[11]や狂牛病の原因であるプリオンタンパク質の沈着に対するER-60の抑制作用が報告されている[12]。これまでの研究で、ER-60が脳では神経細胞に特異的に高発現し、アルツハイマー病モデルマウスのアミロイド斑に濃縮されていること、ER-60がアミロイドβペプチド(Aβ)の重合化を抑制することによりAβの毒性を緩和する作用があること、また、レプチン及びインスリン情報伝達系に障害のあるマウスでは肝臓と脳特異的にER-60の発現が減少していることを見いだした。そして、ノックアウトマウスなどを用いた検討によりER-60がAβの毒性に対する防御作用を有することを見いだしている。さらに、abb’ a’ というドメイン構成であるER-60のbb’ 領域にAβとの結合部位が存在し、bb’ 領域だけでAβの線維化を阻害し細胞毒性にたいして耐性を付与することを明らかにしてきた。そこで、ER-60とAβの複合体の構造解析を行い、ER-60とAβとの結合様式からER-60の防御作用の分子機構を解明することを本研究の目的とした。
実験:
ER-60のbb’ 領域断片を大腸菌によってリコンビナントタンパク質として発現し、精製した。bb’ 断片と結合するAβのN末端側28残基のアミノ酸からなるAβ1-28は化学合成品(株式会社ペプチド研究所)を用いた。bb’ 断片とAβ1-28の複合体結晶は地上あるいは微少重力環境下にある国際宇宙ステーション日本実験棟きぼうのJAXA crystallization box内で、カウンターディフュージョン法により作製した。沈殿剤には20% (w/v) PEG3350/50 mM ammonium sulfate/50 mM HEPES (pH 7.0) 溶液を用いた。Fig. 1に地上及び日本実験棟きぼうで作製したbb’ 断片の結晶の写真を示した。回折実験はSPring-8のBL44XU において、Rayonix社製MX300HEカメラシステムを用いて行った。結晶は窒素気流によって 100 K に冷却し波長0.9ÅのX線で放射線損傷の影響を軽減するためヘリカルスキャン法によりデータを収集した。統計値はTable 1に示した。収集した回折写真は回折データ処理プログラム HKL2000 で処理し、遊離bb’ 断片の構造は既報(PDB ID:2H8L)[13]の構造を、bb’ 断片と Aβ1-28の複合体の構造は遊離bb’ 断片の構造をモデルとしてREFMAC5を用いて精密化を行った。さらに、bb’ 断片と Aβ1-28の複合体の構造解析から結合に関与する可能性が高いと推定された12個のアミノ酸残基をそれぞれアラニンに変異させたタンパク質を、野生型bb’ 断片と同様の手法で発現させ精製した。これらの変異タンパク質とAβ1-40との結合親和性をBIA Core装置 (Biacore 2000 )を用いて解析し、結合能が顕著に低下した変異bb’ 断片(T212A, D153A)と結合能が軽微に変化した変異bb’ 断片(L254A)について結晶化を上記と同様に行い、解析した。
Fig. 1 野生型bb' の結晶
(a)地上で作製した結晶 (b)微小重力下で作製した結晶
Table 1 回折データセットの統計値
結果および考察:
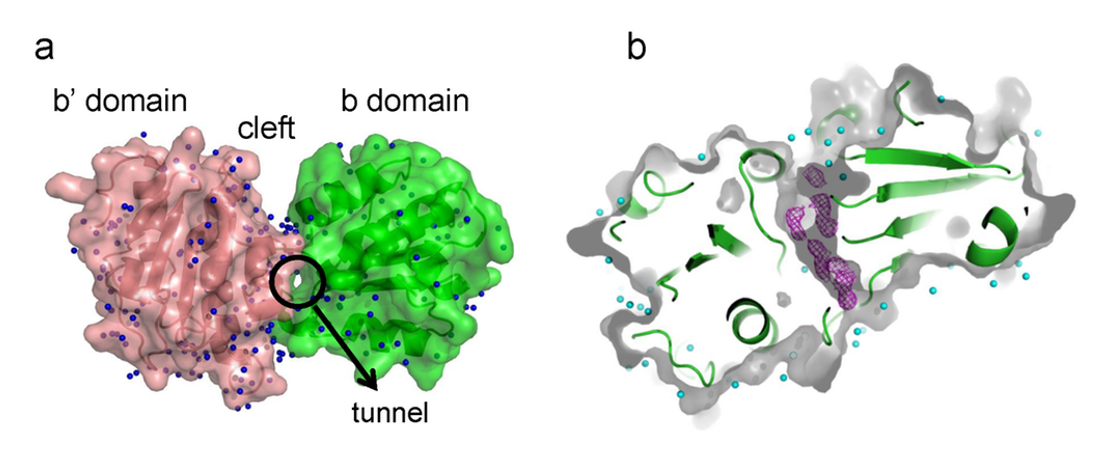
地上では、微少重力下に比較して回折実験に耐える良質な結晶ができる頻度が低かった。また、回折データを取得できた野生型bb’ 断片の地上結晶は最高分解能が2.2Å (Rwork = 0.231, Rfree = 0.286)であったのに対して、微小重力下で作製したほとんどの結晶の回折データの分解能は2Å以下であり最高値は1.39Å (Rwork = 0.198, Rfree = 0.237)であった。すなわち、対流のない微小重力下のほうがbb’ 断片の結晶化には適していた。したがって、以下に示す結晶構造の回折データはすべて微小重力下で作製した結晶から得た。野生型bb’ 断片の結晶の回折データから、b ドメインとb’ ドメインの間に直径20Åの長いトンネルが存在し、その内部は水分子による水素結合のネットワークが形成されていることが明らかとなった(Fig.2a)。一方、野生型に直径20Åの長いトンネルが存在し、その内部は水分子による水素結合のネットワークが形成されていることが明らかとなった (Fig.2a)。一方、野生型 bb’ 断片とAβ1-28の複合体の結晶の回折データからは、複合体のbb’ 断片の構造は遊離型と同じであるがトンネルの入り口付近からトンネル内部にかけて水分子とは明らかに異なる不明瞭な連続的な電子密度が観察された (Fig. 2b)。この電子密度にはペプチド断片として明瞭なモデルを構築するほどの連続性は確認できなかった。しかし、この電子密度は、複合体結晶でのみ観察され、また、他の分子領域にはペプチドに該当する電子密度が観察されなかったことから、Aβ1-28に由来し、Aβ1-28の一部がこのトンネル構造の一部と相互作用をしてbb’ 断片にトラップされていると考えられた。この電子密度がAβ1-28に由来すると仮定して、電子密度の近傍領域に分布する12個のアミノ酸残基がAβ1-28との結合に関わる可能性が考えられた。この可能性を追求するために12アミノ酸残基をそれぞれアラニンに変異させたタンパク質による野生型bb’ のAβ1-28への結合阻害実験を行い、各アミノ酸残基のAβ結合に対する影響を検討した。変異bb’ 断片すべてが野生型bb’ 断片のAβの結合を部分的に阻害したが、変異させたアミノ酸残基により阻害率に大きな差があった。最も阻害効率が低い (すなわちAβとの結合親和性が最も低下した) 変異はThr212とAsp153であった。Leu254の変異は結合親和性にほとんど影響しなかった。これらの変異bb’ 断片とAβ1-28存在下で作製した結晶を解析した。これらのアミノ酸残基の変異が全体の構造にはほとんど影響しないが、Aβとの結合親和性が低下していたThr212とAsp153の変異bb’ 断片の結晶には野生型bb’ 断片とAβ1-28の複合体結晶でみられた不明瞭な連続的な電子密度が観察されず、bb’ 断片に見られたと同様の水分子の水素結合のネットワークが観察された。いっぽう、Leu254の変異bb’ 断片の回折データには不明瞭かつ連続的な電子密度が観察された。以上の結果から、bb’ の境界領域に見られる不明瞭な電子密度はAβ1-28に由来すると考えられる。複合体結晶の分解能が十分に高いにもかかわらずペプチドと考えられる電子密度が不明瞭であることから、ER-60のbb’ の境界領域にAβは一定の配位で結合するのではなくフレキシブルな様式で結合すること、また、その結合には複数のアミノ酸残基が関わっていることが示唆された。
Fig. 2 bb' の結晶構造
(a)遊離野生型bb' の結晶構造(青色の点は同定された水分子を示す) (b)野生型bb' とAβ1-28との複合体のトンネル内の不明瞭な電子密度(紫色の編み目:Fo-Fc, 3σ)(シアンの点は同定された水分子を示す)
今後の課題:
今後は、Aβとの結合親和性が明らかとなっている他の9種類の変異bb’ 断片について、Aβ1-28由来と考えられる電子密度の出現とAβとの結合親和性との相関を検討しその妥当性をさらに検証する必要がある。
参考文献:
[1] Zapun, A. et al., J. Biol. Chem., 273, 6009 (1998).
[2] Otsu, M. et al., J. Biol. Chem., 270, 14958 (1995).
[3] Urade, R. et al., Biochemistry, 43, 8858 (2004).
[4] Okudo, H. et al., J. Biochem., 138, 773 (2005).
[5] Adeli, K. et al., J. Biol. Chem., 272, 22489 (1997).
[6] MaCormic, A.L. et al., Biochemistry, 44, 5794 (2005).
[7] Qiu, W. et al., Biochemistry, 43, 4819 (2004).
[8] Rutledge A.C. et al., Arch. Biochem. Biophys., 537, 104 (2013).
[9] Garbi, N. et al., Nature Immuun., 7, 93 (2005).
[10] Li, Y., & Camacho, P. J., Cell Biol., 164, 35 (2004).
[11] Morand, J.-P.F. et al., J. Biol. Chem., 280, 17626 (2005).
[12] Hetz, C. et al., J. Neurosci., 25, 2793 (2005).
[13] Kozlov G. et al., Structure. 14, 1331-1339 (2006).
ⒸJASRI
(Received: July 26, 2017; Early edition: September 22, 2017; Accepted December 18, 2017; Published: January 25, 2018)