Volume5 No.1
SPring-8 Section A: Scientific Research Report
ジチオナイト還元型NADH-cytochrome b5 reductaseの高分解能結晶構造解析
High-resolution Crystallography on Dithionite-reduced NADH-cytochrome b5 Reductase
京都大学大学院理学研究科
Graduate School of Science, Kyoto University
- Abstract
-
NADH-cytochrome b5 reductase(b5R)はNADHからcytochrome b5(b5)へ電子を受け渡すタンパク質であり、その電子伝達機構を解明するためには活性中心に結合したFADの酸化還元状態の変化による分子構造の変化を明らかにする必要がある。本研究では、酸化型b5R結晶を還元剤ジチオナイトによって還元した結晶についてX線回折実験を行い、構造変化を検証した。
キーワード: FAD、酸化還元酵素、X線結晶構造解析
背景と研究目的:
NADH-cytochrome b5 reductase(b5R)は肝細胞の小胞体に局在し、脂質の代謝に関与する酸化還元酵素である。b5RはNADHからcytochrome b5(b5)への電子伝達を仲介する。分子中央にFAD(フラビンアデニンジヌクレオチド)を保持しており、NADH(ニコチンアミドアデニンジヌクレオチド)が結合するとH-還元によってFADH-となった後、2分子のb5に1つずつ電子を与える。酸化型、NADH結合還元型の結晶構造は既に明らかにされていたが[1、2]、分解能が不十分であり、結晶構造中のFADの酸化還元状態ははっきりしていなかった。b5R中でのFADの状態を決定し、電子伝達機構を解明するためには高分解能での構造解析が必要不可欠であった。
これまでの研究で、我々は酸化型b5Rの結晶構造を最高0.78 Åの超高分解能で決定し、プロトン化状態をもとにb5R中でFADが酸化型であることを確認した[3]。b5Rは強い還元剤のもとでNADH非依存的に還元されることが知られている[4]。本研究では還元剤によって得られるNADH非結合還元型b5Rの結晶構造を超高分解能で決定し、酸化型、NADH結合還元型との比較から、酸化還元による構造変化とNADHの結合による構造変化を分離して分析することを目的とした。
実験:
大腸菌によるリコンビナントとして発現、精製したブタ由来b5Rは吸光度から判断するに酸化型として安定であり、9-12% PEG 4,000、100 mM リン酸カリウム緩衝液pH7.7、5 mM DTTを沈殿剤とする蒸気拡散法によって角柱状の黄色結晶が得られた。この結晶を亜ジチオン酸ナトリウム(ジチオナイト)を含むバッファー(100 mM ジチオナイト、100 mM リン酸カリウム緩衝液pH7.7)に浸すと、結晶表面から中心部に向かい徐々に色が抜けていき、還元されていく様子が観察された。しかしながら、還元型b5Rは好気的条件下で容易に再酸化され、結晶は再び黄色を帯び始める。また、ジチオナイト自身も粉末を溶液に溶かしてから数分以内に還元能を失う。これをふまえ、以下のように回折実験用結晶を調製した。
まず好気的条件下において、上記の酸化型結晶を含む結晶化ドロップ2 μLに20%グリセロール、200 mMジチオナイトを1 μL添加し、数十秒後に40 Kのヘリウム気流によって凍結した。この結晶をRd1とした。同様に好気的条件下において、同様の酸化型結晶を30%グリセロールを含む沈殿剤溶液(抗凍結溶液)に2-3分浸漬した。次に同じ抗凍結溶液にジチオナイト粉末を飽和量以上に添加したドロップに結晶を浸漬し、約10分後に結晶が完全に消色したことを確認して100 Kの窒素気流によって凍結した。この結晶をRd2とした。図1にジチオナイト溶液に浸す前後の結晶を示した。
回折実験はBL41XUにおいて行った。結晶はヘリウム気流によって40 Kに冷却した。収集した回折データの統計値は表1に示した。放射線損傷の影響をさけるためヘリカルスキャンの方法によってデータを収集し、照射点あたりの吸収線量が1 MGyを十分下回るように露光量を設定した。吸収線量の見積もりには損傷評価プログラムRADDOSE[5]を使用した。得られた回折写真は回折データ処理プログラムHKL2000で処理し、酸化型構造をもとに構造精密化プログラムSHELXL[6]によって構造精密化を行った。
図1.b5Rの結晶 (a) 結晶化ドロップにジチオナイトを添加して凍結した結晶(Rd1) (b)ジチオナイト溶液に浸漬する前の状態(酸化型)(c)ジチオナイト溶液に浸漬してから10分後の状態 (Rd2)。黒い影は溶け残ったジチオナイト粉粒である。
表1.各回折データセットの統計値
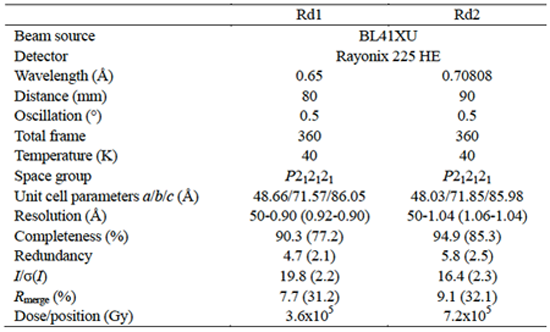
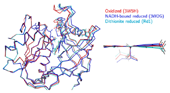
図2.酸化還元状態の異なるb5Rの結晶構造 酸化型(赤、PDBID:3W5H)、NADH結合還元型(青、PDBID:3W2G)とジチオナイト還元型(水色、Rd1)との重ね合わせ。 右はイソアロキサジン環部位の拡大。
結果および考察:
b5Rの酸化型結晶は最高で0.8 Åを超える良好な回折能を示す。還元処理によって結晶表面には凹凸が生じ、大きさもやや小さくなるなど激しい劣化の様子が観察され、回折斑点の形状に乱れが現れた。このため、今回得られた還元型データセットではcompletenessが比較的低くなり(90~95%)、分解能も0.90 Åと1.04 Åに留まった。
b5RはFAD結合ドメイン、NADH結合ドメインの2ドメインで構成されるが、精密化した還元型構造モデルは酸化型に比べ2ドメイン間の距離が僅かに広がっていた。これはNADH結合還元型にもみられた特徴である(図2左)。活性中心であるFADのイソアロキサジン環は、FAD単独の場合、酸化型で平面構造を取るのに対し還元型では湾曲することが知られている[7]。しかしながら、b5R中においては酸化型においても、今回得られた還元型構造においても、イソアロキサジン環はほぼ同じ平面構造を取っており(図2右)、周囲との水素結合も酸化型と同じものが維持されていた。
Rd1の電子密度図上では、イソアロキサジン環部位には炭素原子に結合する不活性な水素原子、および酸化還元反応に関与しない3位の窒素原子(N3)に結合する水素原子と思われる位置にピークが表れていたが、これに加えて1位の窒素原子(N1)付近にも強いピークが観察された(図3b)。このため、還元したb5R中でのFADの状態はFADH-であると推測した(図3a)。酸化型においてN1は水素結合を形成しないが、今回の構造ではFADH-リボース鎖4’位のOH基が一部酸化されてCO基へと変化しており、アクセプターとしてN1と水素結合を形成していた(図3b)。また、ジチオナイトの分解物と思われる亜硫酸イオンがArg63側鎖と水素結合をなす形で結合しているのが観察された(図3c)。Arg63はFADH-のリン酸基とも水素結合を形成することから、ジチオナイト(亜硫酸イオン)→Arg63→リン酸基→リボース鎖→N1へと電子が伝達されたことが示唆された。Rd2ではイソアロキサジン環の水素原子はノイズに埋もれて判別不能であったが、その他の特徴はRd1と一致していた。

図3.ジチオナイト還元型b5Rの活性部位の構造(Rd1)。(a) 電子密度図から推定した構造式。 (b) FADH-のイソアロキサジン環周囲の電子密度図。(c)リン酸基周囲の電子密度図。H原子、および酸化したリボース鎖4’位のO原子を除外したFo−Fc map (赤: 3σ、ピンク: 1.5σ)。2Fo−Fc map (青: 4σ)。イソアロキサジン環の各N原子とリボース鎖4’位のO原子については原子名をラベルした。
上述したように、還元型ではイソアロキサジン環は湾曲した構造がより安定である。しかしながらb5Rでは、酸化型、還元型の両方で平面構造を取っており、これはNADHが結合した還元型の場合でも同様である。すなわち、b5Rは周囲との水素結合によってイソアロキサジン環を常に平面構造に保ち、迅速な電子伝達を可能にしていると考えられる。NADH結合型においてはNADHが結合したことによってFADの位置がずれ、5位の窒素原子(N5)からb5結合領域へと連絡する水素結合網が形成されており、NADHの結合と同期してプロトン転移と協調的なb5への電子伝達が進行することが示唆された。
今後の課題:
本研究で使用した還元型結晶は還元操作中の劣化が激しく、もとの酸化型の結晶よりも回折能、データセットの統計値が非常に悪化した。好気的条件下でより良好なデータを収集するために再現性よく実験を行うことは非常に困難である。このため、嫌気的条件において還元型結晶の調製条件を再検討する必要がある。また、酸化型b5Rを結晶のまま還元したため、結晶格子によって構造変化が阻害されていた可能性も考えられる。溶液状態で還元後に結晶化したものを回折測定し、格子の影響を確かめる必要がある。
参考文献:
[1] H. Nishida, et al., Biochemistry, 34, 2763 (1995).
[2] M. Bewley, C. Marohnic & M. Barber, Biochemistry, 40, 13574 (2001).
[3] M. Yamada, et al., J. Mol. Biol., 425, 4295 (2013).
[4] T. Iyanagi, Biochemistry, 16, 2725 (1977).
[5] K. Paithanker, et al., J Synchrotron Rad., 16, 152 (2009).
[6] G. Sheldrick & T. Schneider, Methods Enzymol., 277, 319 (1997).
[7] Y. Zheng & R. Ornstein, J. Am. Chem. Soc., 118, 9402 (1996).
ⒸJASRI
(Received: September 12, 2016; Early edition: November 25, 2016; Accepted: December 12, 2016; Published: January 31, 2017)






