Volume3 No.1
SPring-8 Section A: Scientific Research Report
キノン型補酵素を持つ酵素の補酵素生成機構の解明
Implications for Quinone Cofactor Biogenesis in Amine-oxidizing Enzymes
a京都大学原子炉実験所, bカンサス大学化学科, c京都大学大学院理学研究科
aResearch Reactor Institute, Kyoto University, bDepartment of Chemistry, The University of Kansas, cGraduate School of Science, Kyoto University
- Abstract
-
ビルトイン型キノン補酵素依存性酵素であるアミンオキシダーゼ,リジルオキシダーゼは,活性中心のトーパキノン(DPQ)やリシンチロシルキノン(LTQ)を利用して生体内リジン末端アミノ基の酸化的脱アミノ化反応を触媒する.本研究では,特に不明な点が多いLTQの生合成機構について構造生物学的な知見を得ることを目指し,アミンオキシターゼ部位特異的変異体−ベンジルアミン複合体の結晶回折実験を行った.
キーワード: ビルトイン型キノン補酵素依存性酵素,リシルチロシルキノン,結晶構造解析
背景と研究目的:
銅含有アミンオキシターゼ(CAO)[1]やリジルオキシターゼ(LOX)[2]は,酵素活性中心にあるビルトイン型キノン補酵素を利用して,生体アミン,コラーゲンやエラスチンの末端アミノ基の酸化的脱アミノ化反応を触媒する酵素である.両酵素のキノン補酵素(CAOではトーパキノン(TPQ),LOXではLTQ,図1)は,それぞれの活性中心にある特定のチロシン残基が,銅を保持した酵素活性中心で分子状酸素と反応し,中間体としてDPQを経て生成すると考えられてきた.特にCAO中のTPQ生合成過程については構造生物学的研究も行われている[3].しかし,この生合成反応では反応中間体のdynamicsには不明な点が多く,詳細は明らかになっていない.また,LTQの生合成反応も不明である.
本研究は,補酵素TPQとLTQの生合成機構,酵素内における補酵素認識機構および反応機構を,CAOとLOXの構造生物学的研究により解明することを目的としている.TPQとLTQの生合成機構では共に酵素活性部位のチロシン残基が酸化されて生成したDPQが共通の反応中間体として推定されていることから,我々のうち牟礼らは,CAOとしてArthrobacter globiformis由来phenylethyl amine oxidase (AGAO)を用い,CAO中のDPQからLTQが生成されるかどうかを証明するための実験を行った[4].その結果,AGAOのAsp298をLysに変異させたD298K変異体において,DPQ中間体とLys298が反応し,LTQ様補酵素が生成することが判った.これは,DPQを介してLTQが生成することを示した初めての例であった.しかし生成したLTQ様補酵素は4位のヒドロキシル基が近傍のチロシン残基との水素結合により安定化されたイミノキノン型であり,かつ不活性型(キノン部位が基質アミンと反応不可能なconformationで固定化されたもの)であったため,オルソキノン型であるLTQ補酵素のモデルとしては最適ではなかった.そこで,Asp298をLysではなくAlaに変更したD298A変異体を作成し,外部からアルキルアミンおよびベンジルアミンを加えることでLTQ型補酵素生成が捕捉されるかどうかを調べたところ,本来のTPQとは異なるLTQ様のUV-VISスペクトルが得られた.またこの酵素はアミン酸化活性を保持していた.本研究では,このAGAO (D298A) -ベンジルアミン複合体の活性部位の詳細な立体構造情報を得ることを目的として,その結晶化と回折実験を行った.
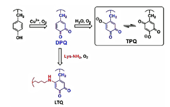
図1.DPQを経由するTPQ,LTQの生合成経路.
実験:
AGAO (D298A) − ベンジルアミン複合体は,D298Aにベンジルアミンを加えた後にCuSO4を加えて反応を進めることで得た.結晶化は,HEPBS(N-(2-Hydroxyethyl)piperazine-N′-(4-butanesulfonic acid))緩衝溶液(0.1 M, pH8.0)に溶解したタンパク質溶液(濃度22 mg/mL)を調製し,蒸気拡散法で行った.結晶化試薬として主にPEGまたは酒石酸を含む条件において,LTQの存在を示すピンク色を呈する菱形の板状結晶を得た.pH4.5〜9.5の条件で得られた結晶5種類について,BL38B1において回折実験を行った.回折実験を行った結晶が得られた条件(表1)と,得られた結晶のうち代表的なものの写真(条件:Cryst 2),およびその回折像の例を以下に示す(図2).
表1.結晶化条件(結晶化試薬)

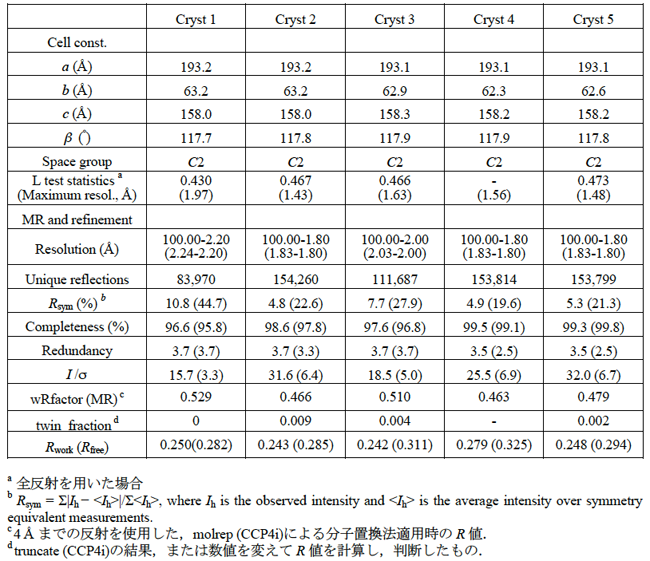
回折実験においては,細いビームを用い,各結晶の数カ所に照射してsnap shotを撮影し,良好な反射を与える場所を検索してデータ収集を行った.最も高分解能の回折データは,Cryst 2に対して,抗凍結剤にグリセロールを用いてフラッシュクーリングを行い,波長1 Å,frame毎の振動角1度,露光時間10秒,検出器までの距離220 mmの条件で測定した時に得られた.回折実験を行ったいずれの結晶からも,2 Å分解能を超える反射が得られた.回折データ処理プログラムHKL2000[5]で反射強度データを処理したところ,全てのデータで空間群はC2,格子定数は概ね,a=193 Å, b=63 Å, c=158 Å,β=117°であった(表2).

図2.AGAO (D298A) - ベンジルアミン複合体結晶(Cryst 2)写真(A)と回折像(B).
表2.回折強度データ統計値(空間群C2で処理)
結果および考察:
すでに構造が明らかになっているAGAO- 5-phenoxy-2,3-pentadienylamine複合体(PDB code: 3kii)[6]をモデルとした分子置換法を適用し,プログラムMolrep[7]を用いて分子置換法による構造解析を進めた.分子置換法で得られた解は非対称単位内にホモ二量体を1つ含む,妥当なものであった.しかし,収集した5セットの反射データのうち4セットは構造解析プログラムで双晶性を示し(表2,図3,[8]),残る1セットも精密化を進める中で得られた電子密度はいずれも不連続な部分を含んだものであり,その解釈が困難であった.図3(A)に,プログラムtruncate (CCP4i)で表示されたL statisticsを示す.
そこで,一定強度以上の反射データのみを使ってのデータ処理と双晶率を変化させての剛体近似精密化,結晶格子軸の入れ替え,空間群の変換などを試みた.それらに加えてTLS-refinement[9]も試みた.しかし,いずれの場合も結晶学的R値と電子密度図を改善することはできなかった.軸の入れ替え操作はR値が悪化し,またdetwinも顕著な効果は確認できなかった.図4に,それぞれの電子密度図を示す(Cryst 1,4以外はdetwin操作後の反射を使用).βシート内で構造が安定していると考えられるにもかかわらず,一義的に主鎖を追うことが困難であったり,manual fittingにより分子モデルを電子密度へ収束させることが適切に行えなかった.分子構造の対称性および分子の形状が,この原因となる結晶の並び方に影響している可能性がある.
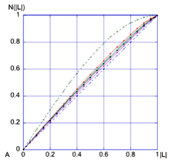
図3.(A):回折データのLtest[8].青破線:完全な非双晶データ,緑破線:完全な双晶データ,赤線:Cryst 1,緑線:Cryst 2,シアン線:Cryst 3,紫線:Cryst 4,黒線:Cryst 5.Cryst 4以外が部分的双晶であると検出されている.L=(I1-I2)/(I1+I2),N(|L|)は|L|の累積分布関数.
今後の課題:
現時点で得られている反射データはいずれも高い分解能の反射を呈するが,双晶性または何らかの問題を持っており,それが構造解析を妨げている.そのため,1.双晶性を示さない結晶を得る,2.活性に関与しない分子表面のアミノ酸残基の特異的変異体を作り分子のパッキングを変化させる,という対策が考えられる.また,ベンジルアミンではなく他のアミンを用いて複合体を作ることが結晶系を変える可能性もある.
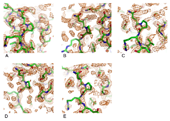
図4.各結晶の電子密度図の一部(2Fo-Fc, 1σ).
A:Cryst 1, B:Cryst 2, C: Cryst 3, D: Cryst 4, E: Cryst 5.
参考文献:
[1] Janes, SM., Mu, D., Wemmer, D., Smith, AJ., Kaur, S., Maltby, D., Burlingame, AL., Klinman, JP., Science (1990), 248, 981-987.
[2] Wang, SX., Mure, M., Medzihradszky, KF., Burlingame, AL., Brown, DE., Dooley, DM., Smith, AJ., Kagan,HM., Klinman, JP., Science (1996), 273, 1078-1084.
[3] Kim, M., Okajima, T., Kishishita, S., Yoshimura, M., Kawamori, A., Tanizawa, K., Yamaguchi, H., Nat.Struct. Biol. (2002), 9, 591-596.
[4] Moore, RH., Spies, MA., Clupepper, MB., Murakawa, T., Hirota, S., Okajima, T., Tanizawa, K., Mure, M., J.Am. Chem. Soc. (2007), 129, 11524-11534.
[5] Otwinowski, Z., Minor W., Methods Enzymol (1997), 276, 307-326.
[6] Ernberg, K., Zhong, B., Ko, K., Miller, L., Nguyen, YH., Sayre, LM., Guss, JM., Lee, I., Biochim. Biophys.Acta, (2011), 1814, 638-646.
[7] Vagin, A., Teplyakov, A., Acta Crystallogr. D Biol. Crystallogr. (2010), 66, 22-25.
[8] Padilla, JE., Yeates, TO., Acta Crystallogr. D Biol. Crystallogr. (2003), 59, 1124-1130.
[9] Winn, MD., Isupov, MN., Murshudov, GN., Acta Crystallogr. D Biol. Crystallogr. (2001), 57, 122-133.
ⒸJASRI
(Received: October 8, 2014; Early edition: December 25, 2014; Accepted: January 16, 2015; Published: February 10, 2015)






