Volume8 No.3
X線自由電子レーザーによる巨大ウイルスの単粒子構造解析
X-ray Free Electron Laser Single Particle Imaging of a Huge Virus
a大阪大学蛋白質研究所, b広島大学大学院医系科学研究科
aInstitute for Protein Research, Osaka University, bGraduate School of Biomedical and Health Sciences, Hiroshima University
- Abstract
-
X線自由電子レーザーを用いた巨大ウイルスの単粒子構造解析を目指した研究に取り組んだ。直径約 160~200 nm の巨大ウイルスを用い、単粒子構造解析のための試料調製法の最適化とその評価方法の開発を行った。最適化した試料を SACLA BL3A における XFEL 回折実験に供し、単粒子由来の回折像を得ることに成功した。
Keywords:X線自由電子レーザー、単粒子構造解析、ウイルス
背景と研究目的:
蛋白質の立体構造を原子分解能で決定する手法としては高輝度低発散角の放射光を利用したX線結晶構造解析が幅広く利用されている。結晶化可能な試料に対しては非常に有用な手法である一方、結晶化できない試料への適用は不可能である。そこで、X線自由電子レーザー (X-ray free electron laser; XFEL)を用いた構造解析手法である Coherent X-ray diffraction imaging (CXDI) が注目されている。この手法は構造解析対象となる分子にコヒーレントかつ短パルス光である XFEL を照射し、試料がクーロン爆発を起こす前に散乱強度を観測[1]し、単粒子からの構造情報を取得するという手法である[2]。我々はこれまでに正二十面体対称を有する巨大球状ウイルスである Paramecium bursaria chlorella virus 1(PBCV-1)[3]をモデル分子として選択し、XFEL を用いた回折実験に適した試料調製法の最適化に取り組んできた[4, 5]。本研究ではさらなる試料調製法の最適化とオフラインでの試料評価方法を確立し、効率的な単粒子構造解析のための回折像取得を目指した。
実験:
PBCV-1 はこれまでに報告されている手法[6]を元にスケールアップを実施し、大量かつ高純度に調製した。調製した試料を Si3N4 膜グリッド(フレーム厚 200 μm、Si3N4 膜厚 100 nm、ウィンドウサイズ 500×500 μm、Norcada 社製)に展開し、ろ紙によるブロッティング、試料展開溶媒による洗浄、モリブデン酸アンモニウムによる染色を実施し、完全に乾燥させたものを XFEL 回折実験の試料とした。展開する試料濃度(吸光度 A260 nm = 20~80)、ろ紙によるブロッティング時間、試料展開溶媒によるグリッド洗浄方法とその回数、染色剤の濃度(2~10%)をパラメータにグリッド作製条件の最適化を実施した。作製した試料グリッド評価のために、電子顕微鏡(H-7100, 日立社製)によるオフラインでの観察を実施した。500×500 μm のグリッドウィンドウ(図1a)を均等に9分割し(図1b)、約 20×20 μm の範囲をモンタージュモードで撮影した(図1c)。その際の倍率は PBCV-1 粒子が十分観察可能な 5,000 倍とした。20×20 μm の撮影範囲から SACLA での実験の際のビームサイズを想定した 1×1 μm の範囲でランダムに切り出し(図1d)、画像のノイズを除去し2値化し面積を計測した。計測した面積と直径 200 nm の粒子面積から粒子数を算出し、0~4 粒子および、凝集と判断するプログラムを MATLAB(The Mathworks, Inc, USA)で実装し(図1e, f)、試料グリッド上の粒子の分布を決定した。このプログラムを用い、グリッド作製条件の最適化を定量的に評価した。
図1. 電子顕微鏡観察によるサンプル展開条件評価法の概要 (a)Si3N4 グリッド(φ3 mm)の光学顕微鏡像 (b, c) グリッドウィンドウ中の9点を均等に電子顕微鏡で撮影 (d) XFEL ビームサイズで 16 点をランダムに切り出す (e) ノイズ除去、2値化し粒子数をカウント (f) 計 144 イメージから試料グリッド上の粒子数の分布を算出
XFEL による回折実験は、2013A-2014A 期に SACLA BL3 において実施した。試料への XFEL の照射には生体分子単粒子解析用のクライオ試料固定照射装置壽壱号[7]を使用した。サンプルグリッドを壽壱号にロードし、ゴニオメーターにマウントした。ゴニオメーターを2次元に 50 μm 間隔で走査し、各点に半値幅 1-2 μm に集光した XFEL(5.5 KeV)を1ポジションに1パルスずつ 1 Hz で照射し回折強度を Multiport Charge Coupled Device(MPCCD)[8]により検出した。散乱強度の検出はサンプル位置から 1642.5 mm に MPCCD を8枚並べた octal detector、3024.9 mm に2枚並べた dual detector を配置し実施した。dual detector はダイレクトビームを避けるために 3.5 mm の開口幅を確保した。
得られた回折パターンをプログラム Owl [https://github.com/FXIhub/owl]で評価し、PBCV-1 粒子サイズ(直径 160-200 nm)に適した回折パターンを選択した。選択したイメージから 512×512 pixel の範囲で切り出し、CXDI のプログラムパッケージである Hawk [9]を用い、回折データにランダムな初期位相を与え Shrinkwrap-algorithm による2次元像の再構築を実施した。
結果および考察:
これまでの SACLA における XFEL 回折実験では PBCV-1 の微結晶試料を用いたが、単粒子よりも散乱断面積が大きい微結晶においても試料からのX線散乱の水による吸収は無視できないと結論づけた[4, 5]ことから、単粒子からの回折像を得るには乾燥試料が適当であると判断した。さらに、高分解能の回折像を得るために重金属による染色を実施することとした。試料濃度を吸光度 A260 nm = 20~80 の範囲で展開し、染色剤であるモリブデン酸アンモニウムの濃度を 2~10% の範囲で検討した。さらに展開した試料の洗浄方法を最適化し、粒子数カウントプログラムにより評価したところ図2に示した様に、電子顕微鏡像と粒子数の分布が非常によく一致しており、定量的に試料調製法を評価できる系を構築することができた。オフラインでの試料調製とその評価を実施した結果、サンプル濃度が A260 nm = 40 の際に最も粒子の凝集が少なく、単粒子として分散している割合が高く、さらに推定されるビームサイズ中に粒子が存在しない確率も低いということが明らかとなった。また、染色剤濃度は 5% を超えると染色剤由来と推測される凝集が確認されたため、電子顕微鏡観察で一般的に用いられている 2% とより高いコントラストが得られることを期待した5%を染色剤濃度とした。さらに粒子もしくは染色剤由来の凝集を減らすためにはサンプル展開後のグリッドを試料展開溶媒である純水で複数回ピペッティングする洗浄が有効であることも明らかとなった。
図2. サンプルを展開したグリッド上サンプルの電子顕微鏡像と自動判別プログラムにより算出された粒子数の割合 (a) 凝集体が多く XFEL 回折実験に適さないと判断したグリッド (b) 粒子数が少なく回折実験に適さないと判断したグリッド (c) 凝集が少なく均一に粒子が展開されたグリッド
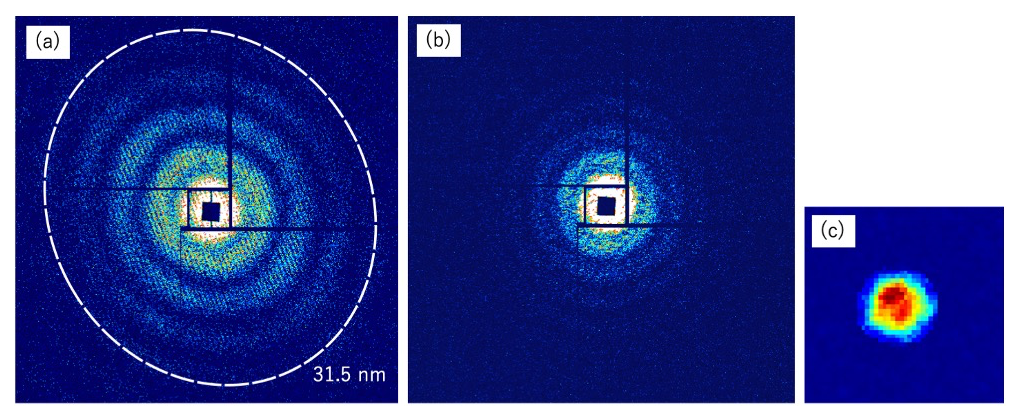
オフラインで最適化した試料グリッドを SACLA での XFE L照射実験に供したところ、複数の試料グリッドから 70% を超えるヒット率で単粒子由来と推測される回折像を得るに至った。最も分解能の高い回折像を図3aに示した。分解能 31.5 nm までのスペックルが観測されたが、スペックル中に干渉縞が観測されたことから2粒子由来の回折像であると考えられる。2つの粒子は約 1.4 μm の間隔をあけて 20° の角度で配置していると推測した。2粒子由来の回折像と比較すると分解能は低いが単粒子由来の回折像も複数観測された(図3b)。得られた回折像の2次元像の再構築を実施したところ、4倍ダウンサンプリングした回折像から図3cに示した様な2次元像の回復に成功した。
図3. 観測された回折像と2次元再構成像 (a) PBCV-1 2粒子由来の回折像 (b) 単粒子由来の回折像、検出器 edge の分解能は 28.9 nm (c) 単粒子由来の回折像から得られた2次元位相回復像
今後の課題:
PBCV-1 粒子をグリッド上で乾燥し染色した試料をオフラインで評価する系を確立したことにより単粒子由来の回折パターン取得の効率は向上した。その結果、単粒子の回折パターンから2次元像の回復には成功したが、未だ分解能が不十分であるため高分解能データ取得が次の課題である。
謝辞:
本研究は、文部科学省X線自由電子レーザー重点戦略研究課題「球状構造体を利用した生体超分子複合体の構造解析法の開発」の支援を受けて実施しました。SACLA の実験にあたっては、京都大学大学院理学研究科・三木邦夫教授、竹田一旗准教授、大阪大学蛋白質研究所・岩崎憲治准教 授、山下栄樹准教授のご協力を得て実施しました。また、慶應義塾大学理工学部・中迫雅由教授および中迫研の皆様、理化学研究所放射光科学総合研究センター・山本雅貴博士、高輝度光科学研究 センター・登野健介博士、城地保昌博士を始めとするビームラインスタッフの方々のご支援に深く感謝致します。データ解析は、Biomedical centre, Uppsala university の Janos Hajdu 教授のグループの皆様のご協力を得て行いました。ここに感謝致します。
参考文献:
[1] R. Neutze et al., Nature, 406, 752-757 (2000).
[2] M. M. Seibert et al., Nature, 470, 78-81 (2011).
[3] J. L. Van Etten et al., Trends Plant Sci., 17(1), 1-8 (2012).
[4] A. Higashiura and A. Nakagawa, SPring-8/SACLA Research Report, 7(2), 321-324 (2019).
[5] A. Higashiura and A. Nakagawa, SPring-8/SACLA Research Report, 8(2), 457-460 (2020).
[6] I. V. Agarkova et al., J. Virol., 80, 8114-8123 (2006).
[7] M. Nakasako et al., Rev. Sci. Instrum., 84, 093705 (2013).
[8] T. Kameshima et al., AIP Conf. Proc., 85, 033110 (2014).
[9] F. R. N. C. Maia et al., J. Appl. Cryst., 43, 1535-1539 (2010).
(Received: September 24, 2020; Accepted: October 23, 2020; Published: October 29, 2020)