Volume8 No.2
SPring-8 Section A: Scientific Research Report
ミトコンドリア AAA-ATPase Msp1 のX線結晶構造解析
X-ray Crystallographic Analysis of the Mitochondrial AAA-ATPase Msp1
京都産業大学
Kyoto Sangyo University
- Abstract
-
真核生物においてタンパク質の誤配送は細胞毒性を引き起こすため、細胞内には誤局在したタンパク質を速やかに除去する機構がある。ミトコンドリア外膜に局在する AAA-ATPase Msp1 は、ミトコンドリア外膜に誤って挿入したテイルアンカー型タンパク質を膜から引き抜く機能を持つ。本研究は、Msp1 のX線結晶構造解析を行い、この基質を膜から引きずり出す仕組みを構造生物学的に解明することを目的とした。これまでに、Msp1 の AAA-ATPase ドメインの結晶を得ることに成功し、3.0 Å 分解能の native データおよび 3.5 Å 分解能の Se-SAD データを取得した。
Keywords: ミトコンドリア、AAA-ATPase、Msp1
背景と研究目的:
真核生物の細胞には、生体膜で仕切られた様々なオルガネラが高度に発達し、細胞機能の発現を支えている。そしてオルガネラが正常に機能するためには、合成されたタンパク質が正確に各オルガネラへ配送される必要がある。シグナル配列と呼ばれる N 末端に付加された行き先を指定するアミノ酸配列を持つタンパク質は、リボソームの翻訳反応と同時に、あるいは直後に適切なオルガネラに配送される。一方、C 末に1本の膜貫通配列をもつテイルアンカー(TA)タンパク質は翻訳反応が完了した後、小胞体膜やミトコンドリア外膜に送られ、膜に挿入される。小胞体の TA タンパク質は、出芽酵母から高等真核生物まで進化的に保存された GET(Guided-Entry of TA proteins)システムによって、ATP 依存的に各膜系に挿入される[1]。GET システムを構成する遺伝子を欠損した細胞では、本来小胞体膜等に挿入されるはずの TA タンパク質はミトコンドリア外膜に誤配送される(ミスターゲッティング)。最近、ミトコンドリア外膜に局在する AAA-ATPase ファミリータンパク質 Msp1(Mitochondrial Sorting of Proteins 1)がミスターゲットした TA タンパク質をミトコンドリア外膜から引き抜くことで、分解を促進する働きを持つことが報告された[2]。本研究は、Msp1 のX線結晶構造解析を行い、この基質を膜から引きずり出す仕組みを構造生物学的に解明することを目的とした。
実験:
Msp1 は、N 末にミトコンドリア外膜にアンカーする膜貫通配列を持ち、サイトゾル側に向いた AAA-ATPase ドメインからなる。Msp1 は、出芽酵母からヒトまで真核生物内で高く保存され、出芽酵母の Msp1 は全長 362 アミノ酸残基(分子量 40 kDa)である。申請者は、耐熱性酵母 Kluyveromyces marxianus 由来の Msp1(KmMsp1)について、N 末端の膜貫通配列を除去した可溶性の AAA-ATPase ドメイン(32-360)の構造解析を目指した。KmMsp1 は、N 末に His-tag を融合した組換えタンパク質として大腸菌で大量発現を行い、Ni アフィニティ精製、3C プロテアーゼによる His-tag の切断そしてゲルろ過の順番で高純度に精製した。KmMsp1 は 20 mg/ml まで濃縮し、結晶化実験に用いた。結晶化試薬は、0.2 M 硫酸アンモニウム、0.1 M MES-NaOH(pH 6.5)、12-15 %(w/v)PEG3350、結晶化温度は 20 度、結晶化方法はハンギングドロップ蒸気拡散法を用いて、結晶を作製した。また、得られた KmMsp1 の結晶は、アポ体であることが予想されたため、KmMsp1 とヌクレオチド(ADP や AMP-PMP)との共結晶化に向けたスクリーニングも並行して行った。セレノメチオニン置換の KmMsp1 は、上記の native タンパク質と同じ結晶化方法によって得られた。KmMsp1 結晶は、結晶化試薬の組成の溶液に 10-15%(v/v)グリセロールを加えた溶液に浸し、クライオプロテクタントとした。BL44XU での回折実験では、native データ収集およびセレン原子の単波長異常散乱法による位相決定のためのデータ収集を行った。
結果および考察:
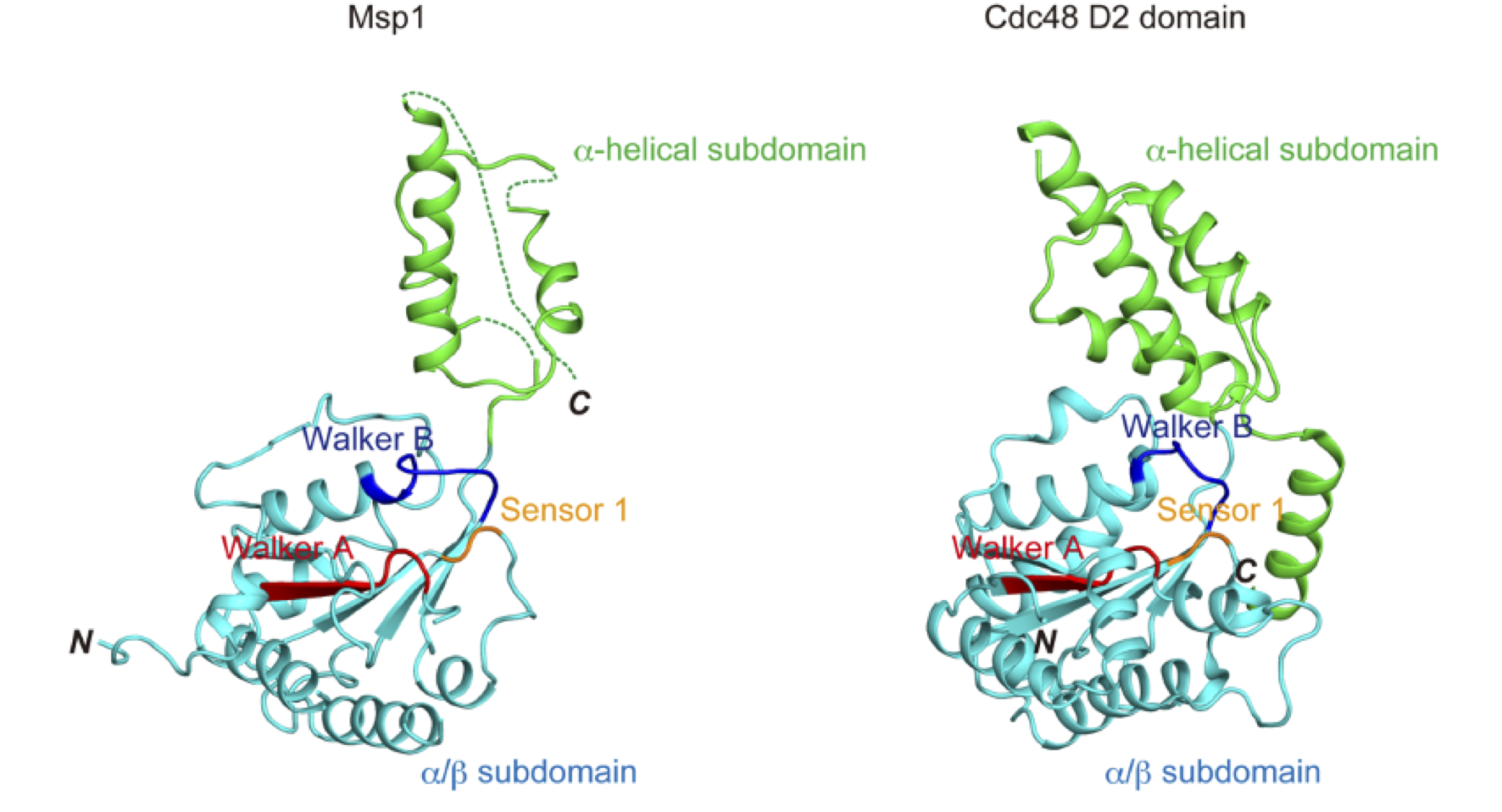
本申請の以前より、BL44XU での回折実験を行った結果から、KmMsp1 の結晶から 3.0 Å 分解能の native データおよび位相決定のための 3.5 Å 分解能 Se-SAD データを取得することに成功していた(表1)。これらの結果から KmMsp1 の空間群は P43212 に属し、結晶格子は、a = 70.5、b= 70.5、 c = 154.2 であった。Se-SAD データは AutoSol(PHENIX)を用いることで、初期位相が得られた。得られた電子密度をもとに、モデル構築を行った後、KmMsp1 の native データを使い、モデル構築およびphenix.refine(PHENIX)を用いた構造精密化を行った。現在までに、全 320 残基中 222 残基の原子モデルを構築したが、Rfree/Rwork = 38.8 / 33.6(%)となり、Rfree 値をこの値より低くすることできなかった。これは,KmMsp1 の AAA-ATPase ドメインを構成する α/β サブドメインのモデルは構築できたが、C 末端の領域に相当する α ヘリカルサブドメインとなる領域のモデルは電子密度が弱く、十分にモデル構築できなかったことが原因の1つではないかと考察した。本申請における回折実験において、この問題を解決するために native データセットの取得に成功したが、上述のデータセット以上に精密化が進むものは得られなかった。これまでに構築した Msp1 構造モデルは、Cdc48 の AAA-ATPase D2 ドメイン(PDB ID: 3CF3)と類似していた。これは、アミノ酸配列の相同性から予想されたことだが、AAA-ATPase ドメインを構成する α/β サブドメインと α ヘリカルサブドメインの配向が Cdc48 D2 ドメインと異なっていた(図1)。KmMsp1 の構造は、パッキングの様式からモノマーのアポ体であったため、KmMsp1 とヌクレオチド(ADP や AMP-PMP)の結晶化も行った。しかしながら、析出する結晶はアポ体の結晶化条件と同じであることから、ヌクレオチドとの共結晶は得られなかった。
| Native | Se-SAD | |
| Data collection statistics | ||
| Beamline | SPring-8 BL44XU | SPring-8 BL44XU |
| Wavelength (Å) | 0.9000 | 0.9791 |
| Oscillation range (°) | 180 | 600 |
| Space group | P43212 | P43212 |
| Cell dimensions | 180 | 600 |
| Cell dimensions | a = b = 70.5 Å, c = 154.2 Å | a = b = 70.5 Å, c = 154.2 Å |
| Resolution range (Å) | 50.0 – 3.00 (3.05 – 3.00) | 50.0 – 3.40 (3.46 – 3.40) |
| Observed reflections | 103353 | 254568 |
| Unique reflections | 8218 | 9911 |
| Completeness (%) | 98.6 (100.0) | 99.6 (99.4) |
| Rmerge(I)a | 0.136 (> 1) | 0.147 (> 1) |
| <I / σ(I)> | 37.9 (3.3) | 61.9 (5.8) |
| CC1/2 | 0.732 | 0.867 |
| Refinement statistics | ||
| Resolution (Å) | 41.9 – 3.00 (3.05 – 3.00) | |
| No. of protein atoms | 1696 | |
| No. of water molecules | 0 | |
| Rwork/Rfreeb | 0.336/0.388 | |
| rmsdc from ideality bond length (Å) | 0.04 | |
| angles (°) | 2.049 | |
| Ramachandran plot (%)d | ||
| Favored region | 77.1 | |
| Allowed region | 14.3 | |
| Outlier region | 8.6 |
Values in parentheses are for the highest resolution shell.
aRmerge(I) = (ΣΣ|Ii - <I>|)/ΣΣIi, where Ii is the intensity of the ith observation and <I> is the mean intensity.
bRwork/Rfree = ∑|FO - FC|/∑|FO|. Rwork was calculated from the working set (95% of the total reflections). Rfree was calculated from the test set, using 5% of the total reflections. The test set was not used in the refinement.
crmsd, root mean square deviation.
図1 KmMsp1 と Cdc48 の D2 ドメインの構造比較
今後の課題:
今後は、他の生物種由来のMsp1の結晶化を行うことを検討している。これまでにゼブラフィッシュ由来 Msp1 の AAA-ATPase ドメインが高い可溶性を示し、精製することができている。この Msp1 についても現在微結晶が得られているので、結晶化条件の最適化を行うことで、構造決定を目指す。また、Msp1 と基質複合体の結晶化に向けて、モデル基質 Pex15Δ30 の精製系の確立を目指す。
参考文献:
[1] M. Schuldiner et al., Cell, 128, 1147 (2008).
[2] V. Okreglak, P. Walter, Proc. Natl. Acad. Sci. USA., 111, 8019 (2014).
(Received: April 15, 2020; Early edition: June 26, 2020; Accepted: July 6, 2020; Published: August 21, 2020)