Volume8 No.2
SPring-8 Section A: Scientific Research Report
DNAを高感度に検出可能な新規蛍光センサーとDNAとの複合体の X線結晶構造解析
X-ray Crystallographic Analysis of a Highly Sensitive DNA Fluorescence Sensor Bound to DNA
兵庫医療大学薬学部
School of Pharmacy, Hyogo University of Health Sciences
- Abstract
-
新たに開発した蛍光 DNA センサーの DNA 検出メカニズムを原子レベルで解明することを目的として、センサー分子- DNA 複合体の単結晶作成およびX線結晶構造解析を試みた。種々の短鎖 DNA を用いて、センサー分子共存下で結晶化を行ったところ、2種の単結晶が得られたため、それらを放射光を利用したX線結晶構造解析に供した。
Keywords:DNA、蛍光 DNA センサー、X線結晶構造解析
背景と研究目的
DNA に特異的に結合する分子は DNA を認識したり阻害したりする化合物であり、抗がん剤や DNA センサーとして応用されている。近年では核酸医薬が注目される一方で、DNA に結合する小分子についても応用例は数多い。小分子は一般的に大量合成や化学修飾が容易なため、DNA に結合する小分子を改良して副作用の少ない抗がん剤や DNA センサーの開発を目指す研究は、現在も盛んに行われている[1]。
DNA に対する結合様式のひとつとして、平面芳香環分子が二重らせん DNA の塩基対-塩基対間に、各塩基対に対して平行に挿入するインターカレーションが知られている。DNA に特徴的な結合様式のため、インターカレーターは DNA を特異的に認識および阻害する分子として利用可能である。一方で、インターカレーターは平面芳香環分子に特徴的な強い疎水性相互作用や π-π スタッキング相互作用を示すため水に難溶のものが多く、利用が制限される。特に、炭素と水素のみで構成されたピレン等の応用例は、その疎水性ゆえ、代替塩基や DNA 鎖の蛍光標識などの用途に限られていた。しかしながら、そのような分子は優れた蛍光特性を有する場合が多く、その単純な構造のため化学的安定性が高く化学修飾が容易といった利点も有するため、機能性分子としての利用価値は高く、未だ開発の余地が残されている。
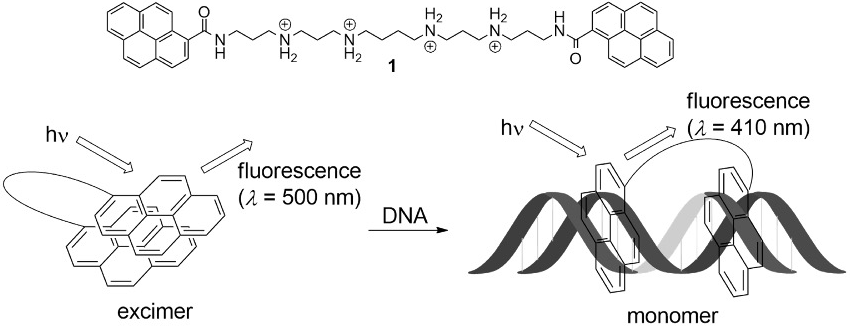
このような背景の下、筆者は、疎水性インターカレーターとして知られているピレンを利用し、ポリアミンリンカー型蛍光 DNA センサー 1 を開発した(図1)。水中において、分子内の2つのピレン部位は疎水性相互作用により二量体を形成するため、1 はエキシマー蛍光と呼ばれる波長約 500 nm の蛍光を発するが、DNA 存在下ではピレン二量体が DNA との相互作用により解離し、波長約 410 nm のモノマー蛍光を発する。それらの波長における蛍光強度比を観測することで、DNA を検出、可視化できる。
開発したセンサー 1 は合成が容易、かつ化学修飾や分子変換の余地が多いため、さらなる高機能化が望める。しかしながら、センサー分子の高機能化を戦略的に行うには、センサー分子と DNA との結合様式を原子レベルで解明する必要がある。
以上を踏まえ、センサー分子の高機能化に資する構造情報を得る目的で、開発したセンサー分子 1 と DNA との共結晶を作成し、その立体構造をX線結晶構造解析により明らかにすることとした。
実験:
DNA として、B-DNA のX線結晶構造解析で頻繁に用いられている Dickerson-Drew dodecamer [2] をはじめ、さまざまな短鎖 DNA を用い、合成したセンサー分子 1 との共結晶化を試みたところ、d(CGAAGCTTCG) および d(CGGAAATTACCG) を用いた以下の (1) および (2) の条件で、X線回折実験が可能な結晶を得ることに成功した。なお、緩衝液として 20 mM カコジル酸緩衝液 (pH 6.5) を用い、結晶化はハンギングドロップ蒸気拡散法により行った。
図1.開発した蛍光DNAセンサー 1 と予想されるシグナリング機構
(1) d(CGAAGCTTCG) を 2 mMとなるように緩衝液に溶解し、363 Kで1分間加温した後、室温まで冷却し、1 mM double-stranded (ds) DNA溶液とした。1 mM dsDNA 溶液および緩衝液に溶かした 1 mM 1 溶液を体積比 1 : 1 で混合して得られた溶液 1 µL に、5 mM BaCl2、5 mM NaCl および 60% (w/v) 2-メチルペンタン-2,4-ジオール (MPD) から成る沈殿剤溶液 0.6 µL を加えて結晶化母液とし、293 K で静置したところ、0.2 × 0.05 × 0.05 mm3 の単結晶を得た。
(2) d(CGGAAATTACCG) を 2 mM となるように緩衝液に溶解し、363 K で1分間加温した後、室温まで冷却し、1 mM dsDNA 溶液とした。1 mM dsDNA 溶液および緩衝液に溶かした 1 mM 1 溶液を体積比 1 : 1 で混合して得られた溶液 1 µL に、5.5 mM MgCl2、11 mM NaCl および 45% (w/v) MPD から成る沈殿剤溶液 0.5 µL を加えて結晶化母液とし、293 K で静置したところ、多結晶が析出した。得られた多結晶の一部を切り出し、0.1 × 0.05 × 0.05 mm3 の単結晶を得た。
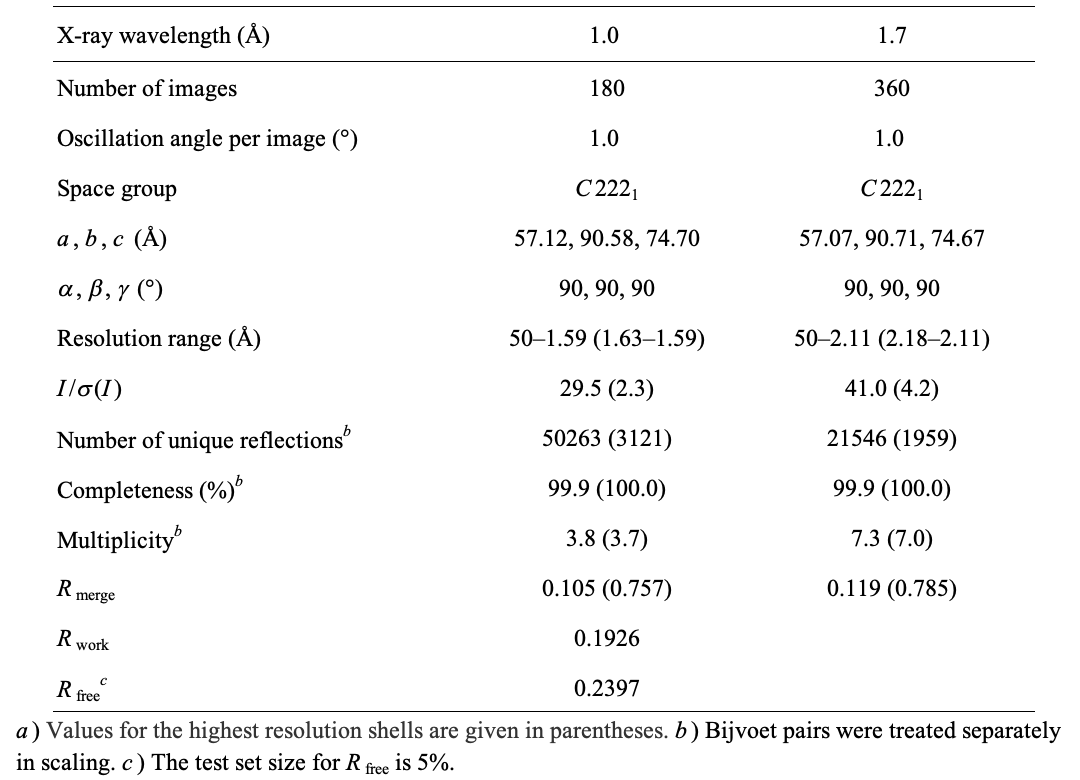
得られた結晶のそれぞれについて、BL38B1 において、窒素気流下で 95 K に冷却し、X線回折データを収集した。d(CGAAGCTTCG) の結晶については、X線波長 1.0 Å の回折データセットを収集した後、Ba2+ を利用した単波長異常散乱 (SAD) 法による位相決定のため、X線波長1.7 Åのデータセットも収集した。得られた各データセットのプロセッシングおよびスケーリングは、プログラムパッケージ HKL2000 [3]を用いて行った。d(CGGAAATTACCG) の結晶については、X線波長 1.0 Å の回折データセットを収集し、CCP4 の iMosflm/SCALA [4]を用いてプロセッシングおよびスケーリングを行った。
d(CGAAGCTTCG) 結晶の位相決定は、X線波長 1.7 Å のデータセットを用い、プログラム SHELXC/D/E[5]を利用した SAD 法により行った。構造モデルの構築にはプログラム Coot [6]を用い、精密化にはプログラムパッケージ PHENIX の phenix.refine [7]を用いた。ディスオーダーにより複数の結合サイトを占有する Ba2+ の占有率は、合計 1.0 になるように束縛をかけた場合と束縛をかけなかった場合で精密化における統計値に有意な差が見られなかったため、合計1.0になるように束縛をかけて精密化を行った。
結果および考察:
1. d(CGAAGCTTCG) の結晶構造解析
d(CGAAGCTTCG) の結晶については、波長 1.0 Å のX線を用いることで、分解能 1.59 Å までの良質な回折データが得られた(表1)。沈殿剤として BaCl2 を用いており、Ba2+ による異常分散効果を利用した位相決定が可能と考えられたため、より大きな異常分散シグナルが得られる波長 1.7 Å のX線を用いた回折データも収集し、SAD 法による位相決定を行った。その結果、良好な初期位相が得られ、容易に解釈可能な電子密度マップが得られた。構造モデルの構築および精密化を繰り返し、最終的に結晶構造を決定することに成功した。
表1.d(CGAAGCTTCG) 結晶のX線回折実験および構造精密化における統計値a
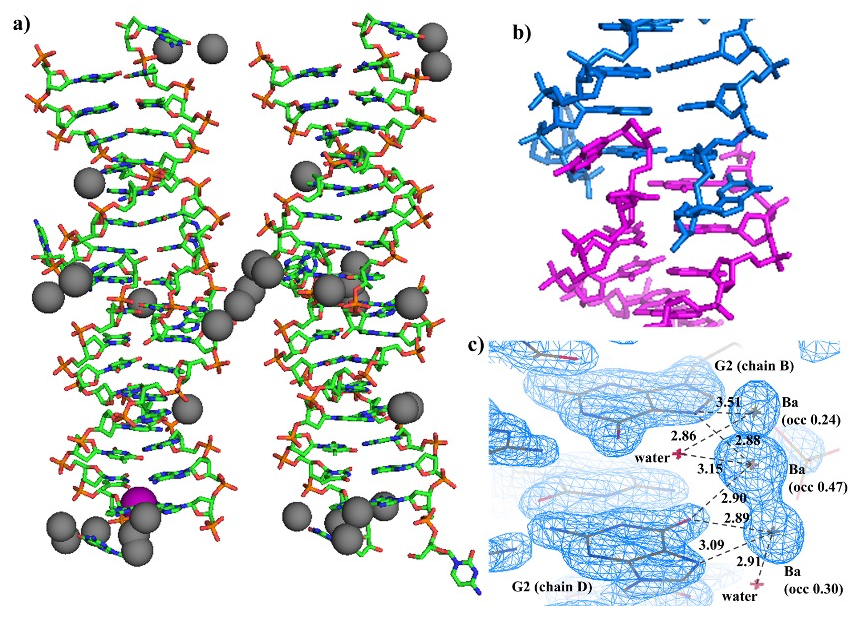
しかしながら、得られた構造は望みの DNA -センサー分子 1 複合体ではなく、1 が含まれない dsDNA の結晶構造であった(図2a)。非対称単位中に dsDNA が4つ含まれ、複数の Ba2+ および1つの Na+ が結合していた。DNA は自己相補的配列を有しているため二重鎖を形成するが、それぞれの dsDNA においては、両端を除いた中央の8残基のみが B-DNA 構造を形成し、両端の C-G ペアは開裂していた。これは Spink らによって最初に見出された d(CGCAATTGCG)2 の結晶構造[8]と同様であり、らせん軸に沿って隣り合う dsDNA それぞれの末端 G 残基が、互いのマイナーグルーブに入り込み、結合しあっていた(図2b)。また、開裂した末端 C 残基はフレキシビリティーが高く、ディスオーダーによって帰属できないものもあった。Ba2+ は DNA に対して緩やかに結合することが知られているが[9]、本結晶構造においても、DNA に結合している Ba2+ はディスオーダーしているものが多く、2つあるいは3つの結合サイトを占有する Ba2+ も複数見られた(図2c)。
図2.a) d(CGAAGCTTCG) の結晶構造(gray spheres: Ba2+; a purple sphere: Na+)、b) 開裂した末端 G 残基付近の DNA 構造、c) ディスオーダーした Ba2+ サイトの例(occは占有率、点線に付随する値は結合距離 (Å) を示す)
用いた d(CGAAGCTTCG) は、溶液中で 1 と相互作用することが蛍光シグナル測定により明らかになっているため、複合体の構造を得るには、結晶化条件を検討し直す必要があると考えられる。また、DNA と 1 との親和性は、用いる DNA 配列にも影響されると考えられる。例えば、マイナーグルーブに結合する有機分子は、立体的な制約から、連続した A-T 配列に結合しやすいことが知られており、例えば前述の Dickerson-Drew 型の dsDNA では中央の d(AATT)2 に結合しやすい。1 の配列選択性については現在のところ不明であるが、用いる DNA の配列もより広く検討する必要があると考えられる。一方で、Ba2+ は、Mg2+ や Zn2+ 等と比べて核酸に対する結合安定性は低く[9]、本構造においても多数の Ba2+ サイトでディスオーダーが見られるが、位相決定には有用であり、DNA の結晶化でも積極的に検討すべき沈殿剤の一つであると考えられる。
2. d(CGGAAATTACCG) の結晶構造解析
d(CGGAAATTACCG) の結晶において、波長 1.0 Å のX線を用いて、分解能 2.30 Å までの回折データが得られた。空間群は P6122 もしくは P6522 と考えられたため、それぞれの空間群でプロセッシングしたデータを用いて、分子置換法による位相決定を行うこととした。サーチモデルとして12残基の B-DNA モデルおよび A-DNA モデルを用い、さまざまなパラメーター条件下で分子置換を試みた。しかしながら、良好な初期位相が求められず、解釈可能な電子密度を得ることはできなかった。位相決定に至らなかった理由として、用いた DNA が A-A ミスマッチを含む相補鎖であるため、典型的な二重らせん構造を形成していない可能性が考えられる。また、回折データ測定には多結晶から切り出した単結晶を用いたが、単結晶に付随した分離不能な微結晶由来の回折点もいくらか観測され、良質な回折データが得られなったことも原因の一つとして考えられる。今後、構造解析を進めるためには、位相決定のための重原子の利用や、結晶化条件の更なる最適化を検討する必要がある。
今後の課題:
本研究では、開発した DNA センサー 1 と DNA との結合様式を明らかにするため、さまざまな DNA と 1 との共結晶化および構造解析を試みたが、望みの DNA-1 複合体の立体構造を得ることは出来なかった。今回、構造解析に至った d(CGAAGCTTCG) は、溶液中では 1 と相互作用することが明らかになっているが、結晶中では複合体を形成していなかった。このことから、用いる DNA 配列および結晶化条件をさらに広く検討する必要があると考えられる。今回、DNA -有機分子複合体の構造解析で多くの実績がある Dickerson-Drew dodecamer やその類似配列を有する DNA を用いた検討も行っており、回折データが得られるほどの品質ではないものの、結晶が析出することが分かっている。これらの得られた知見を基に、今後、更なる条件検討を重ね、望みの複合体構造を得たいと考えている。
参考文献:
[1] a) R. Martinez, L. Chacon-Garcia, Curr. Med. Chem. 12, 127 (2005); b) B. Dumat et al., J. Am. Chem. Soc. 135, 12697 (2013); c) C. Mari et al., Chem. Sci. 6, 2660 (2015).
[2] V. Tereshko, G. Minasov, M. Egli, J. Am. Chem. Soc. 121, 470 (1999).
[3] Z. Otwinowski, W. Minor, Methods Enzymol. 276, 307 (1997).
[4] M. D. Winn et al., Acta Cryst. D67, 235 (2011).
[5] G. M. Sheldrick, Acta Cryst. D66, 479 (2010).
[6] P. Emsley et al., Acta Cryst. D66, 486 (2010).
[7] P. V. Afonine et al., Acta Cryst. D68, 352 (2012).
[8] a) N. Spink et al., Proc. Natl. Acad. Sci. USA 92, 10767 (1995); b) N. Valls et al., Acta Cryst. D60, 680 (2004).
[9] a) E. Ennifar, P. Walter, P. Dumas, Nucleic Acids Res. 31, 2671 (2003); b) M. Gilski et al., Acta Cryst. D72, 211 (2016).
(Received: February 6, 2020; Early edition: May 27, 2020; Accepted: July 6, 2020; Published: August 21, 2020)