Volume8 No.2
X線自由電子レーザーによる単粒子構造解析の試料調製法の最適化
Optimization of Sample Preparation Method for Single Particle Imaging by X-ray Free Electron Laser
a大阪大学蛋白質研究所, b広島大学大学院医系科学研究科
aInstitute for Protein Research, Osaka University, bGraduate School of Biomedical and Health Sciences, Hiroshima University
- Abstract
-
X線自由電子レーザーを用いた単粒子構造解析の手法開発を目指した研究に取り組んだ。直径約 160~200 nm のウイルスを用い、単粒子構造解析のための試料調製法の最適化を実施した。試料調製法の評価のために SACLA BL3A において、XFEL 回折実験を実施した。
Keywords:X線自由電子レーザー、単粒子構造解析、ウイルス
背景と研究目的:
蛋白質の立体構造を原子分解能で決定する手法としては高輝度低発散角の放射光を利用したX線結晶構造解析が幅広く利用されている。結晶化可能な試料に対しては非常に有用な手法である一方、結晶化できない試料への適用は不可能である。そこで、2010 年に米国 Linac Coherent Light Source (LCLS) にて、2012 年より SACLA において共用利用が開始されたX線自由電子レーザー (X-ray free electron laser; XFEL) を用いた構造解析手法である Coherent X-ray diffraction imaging (CXDI) が注目されている。この手法は構造解析対象となる分子にコヒーレントかつ短パルス光である XFEL を照射し、試料がクーロン爆発を起こす前に散乱強度を観測[1]し、単粒子からの構造情報を取得するという手法である[2]。我々はこれまでに XFEL を用いた蛋白質やその複合体の立体構造解析手法の開発を目指し、正二十面体対称を有する球状ウイルスである Paramecium bursaria chlorella virus 1(PBCV-1)[3]とイネ萎縮ウイルス(Rice dwarf virus; RDV)[4]をそのモデル分子として選択し、XFEL を用いた回折実験に取り組んだが、凍結固定したウイルス粒子からの単粒子回折パターンを得ることはできなかった[5]。そこで、直径約 200 nm の PBCV-1 を用い、単粒子構造解析のためのサンプル調製法の最適化に取り組むこととした。
実験:
PBCV-1 はこれまでに報告されている手法[6]を元にスケールアップを実施し、大量かつ高純度に調製した。調製した PBCV-1 をハンギングドロップ蒸気拡散法による結晶化に供した。260 nmの 吸光度をA260nm=100 とした PBCV-1 溶液を pH8.0 100 mM HEPES、0.5% PEG35,000 溶液と 1 µl:1 µl の割合で混合し、20℃ に静置した。得られた微結晶を結晶化溶液で懸濁したものを回折実験に供した。また、微結晶試料は Si3N4 膜グリッド(フレーム厚 200 μm、Si3N4 膜厚 100 nm、ウィンドウサイズ 500×500 μm、Norcada 社製)に展開し、乾燥させたものを電子顕微鏡(H-7100, 日立社製)により観察した。
回折実験に用いる凍結試料調製は Si3N4 膜グリッド上に、結晶化溶液で懸濁した PBCV-1 微結晶溶液を展開し、自動浸漬凍結装置 Leica EM GP(Leica 社製)を用いて液体窒素温度の液体エタン中において急速凍結した。凍結直前のろ紙によるブロッティング時間でサンプル量と氷の厚みを調整した。PBCV-1 微結晶の乾燥試料調製には凍結試料と同型のグリッドを用い、微結晶溶液を展開し自然乾燥した。PBCV-1 溶液の乾燥試料は微結晶試料と同様にグリッドに展開し、自然乾燥した。ウイルス溶液の濃度はA260nm=10 とした。
XFEL による回折実験は SACLA BL3 において実施した。試料への XFEL の照射には生体分子単粒子解析用のクライオ試料固定照射装置壽壱号[7]を使用した。サンプルグリッドを壽壱号にロードし、ゴニオメーターにマウントした。ゴニオメーターを2次元に 50 μm 間隔で走査し、各点に半値幅 1-2 μm に集光した XFEL(5.5 KeV)を1ポジションに1パルスずつ 1 Hz で照射し回折強度を Multiport Charge Coupled Device(MPCCD)[8]により検出した。散乱強度の検出はサンプル位置から 1642.5 mm に MPCCD を8枚並べた octal detector、3024.9 mm に2枚並べた dual detector を配置し実施した。dual detector はダイレクトビームを避けるために 3.5 mm の開口幅を確保した。
結果および考察:
PBCV-1 の結晶化では約一週間で最大数十 μm の微結晶を得た(図1(a))。得られた微結晶を電子顕微鏡で観察したところ、数 μm の微結晶が存在している様子が確認された。さらに、ウイルス粒子が部分的に二次元結晶の様に周期的に配置している様子が観察されたが、完全な三次元の結晶は観察されなかった(図1(b))。
図1. PBCV-1 の微結晶 (a)光学顕微鏡像、(b)電子顕微鏡像
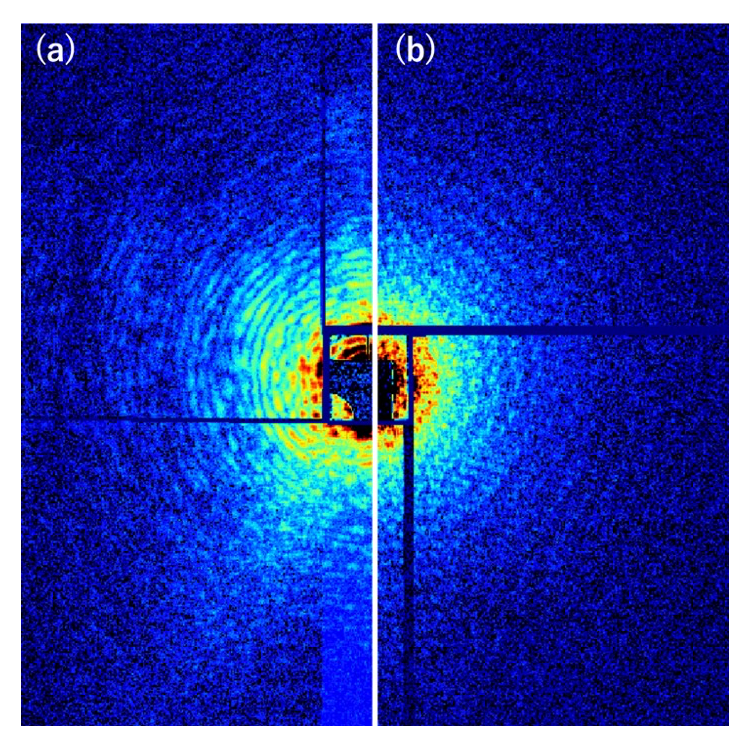
単粒子よりも散乱断面積が大きい微結晶を用いて氷の厚みの評価を実施した。氷の厚さを凍結直前のブロッティング時間により調整し検討したところ、0.5 秒の blotting では多数の散乱パターンを観測することができたが、サンプル由来と判断される散乱パターンを得ることはできなかった。さらに、blotting 時間を3秒とすると、径の大きな粒子由来と考えられるリング状の回折パターンが観測された(図2(a))。そのピッチは実空間で約 1 mm であったことから微結晶由来のパターンであると推察された。さらに blotting 時間を長くすると回折強度がほとんど観測されなかったため、長時間の blotting によってサンプル量が不足してしまうことが示唆された。さらに氷の影響を評価するため、微結晶サンプルをグリッド上で乾燥し回折実験に供したところ、図2(b)に示す様な回折点を観測するに至った(図2(b))。これは結晶様に配置したウイルス由来の回折点が重なりあったパターンであると推察した。図2(b)の様な回折パターンが凍結試料から全く観測されなかったのは氷による吸収が影響していると結論づけた。
図2. PBCV-1 の微結晶サンプルから得られた回折パターン (a) PBCV-1 微結晶を Si3N4 膜グリッドに展開し、急速凍結し XFEL 回折実験に供した (b) PBCV-1 微結晶を Si3N4 膜グリッドに展開し、乾燥し XFEL 回折実験に供した
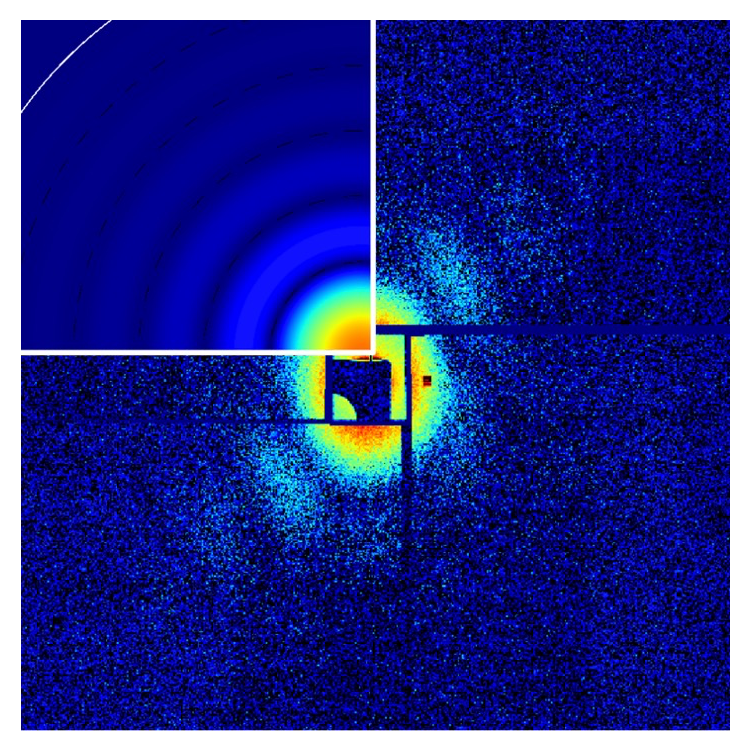
単粒子よりも散乱能が大きな微結晶においても氷による吸収は無視できないという結果から、溶液状態のウイルスをグリッド上に展開し乾燥した試料を回折実験に供した。非常に低いヒット率(約 1%)であったが、図3に示す様な回折パターンが観測された。この繰り返し周期がおおよそ 160 nm であったことからウイルス単粒子由来の回折パターンであると判断した。しかし、単粒子解析を行うには不十分な分解能であったため、構造解析には至らなかった。
図3. PBCV-1 の乾燥試料から得られた回折パターン、溶液状態の PBCV-1 を Si3N4 グリッドに展開し、乾燥させた、検出器 edge の分解能は 28.9 nm(左上)160 nm の球からの散乱パターンのシミュレーション像
今後の課題:
微小結晶の凍結試料を用いた回折実験より氷による散乱強度の吸収は無視できないと結論づけた。溶液状態のウイルスをグリッド上に展開し乾燥させた試料から低分解能であるが、単粒子由来と考えられる回折強度を観測することができたが、分解能とヒット率の低さから構造解析には至らなかった。ウイルスサンプルを均等にグリッドに展開する手法の最適化によりヒット率を向上させ、分解能向上を目指した重金属による染色方法の検討が今後の課題である。
謝辞:
本研究は、文部科学省X線自由電子レーザー重点戦略研究課題「球状構造体を利用した生体超分子複合体の構造解析法の開発」の支援を受けて実施しました。SACLA の実験にあたっては、京都大学大学院理学研究科・三木邦夫教授、竹田一旗准教授、大阪大学蛋白質研究所・岩崎憲治准教 授、山下栄樹准教授のご協力を得て実施しました。また、慶應義塾大学理工学部・中迫雅由教授および中迫研の皆様、理化学研究所放射光科学総合研究センター・山本雅貴博士、高輝度光科学研究 センター・登野健介博士、城地保昌博士を始めとするビームラインスタッフの方々のご支援に深く感謝致します。
参考文献:
[1] R. Neutze et al. Nature, 406, 752-757 (2000)
[2] M. M. Seibert et al. Nature, 470, 78-81 (2011)
[3] J. L. Van Etten et al. Trends Plant Sci., 17(1), 1-8 (2012)
[4] A. Nakagawa et al. Biophys. Rev., 10(2), 659-665 (2018)
[5] A. Higashiura and A. Nakagawa SPring-8/SACLA Research Report, 7(2), 321-324 (2019)
[6] I. V. Agarkova et al. J. Virol., 80, 8114-8123 (2006)
[7] M. Nakasako et al. Rev. Sci. Instrum., 84, 093705 (2013)
[8] T. Kameshima, S. Ono et al., AIP Conf. Proc., 85, 033110 (2014)
(Received: February 10, 2020; Early edition: May 27, 2020; Accepted: July 6, 2020; Published: August 21, 2020)