Volume7 No.2
SPring-8 Section A: Scientific Research Report
CTX-M型β-ラクタマーゼの基質特異性に対するAla219の役割
A Role of Ala219 on the Substrate Specificity of CTX-M Type β-Lactamase
奈良県立医科大学化学教室
Department of Chemistry, Nara Medical University
- Abstract
-
CTX-M型β-ラクタマーゼ(cefotaxime分解酵素)で高度に保存されている Ala219 の 役割を解明するために、野生型CTX-M-2、及び変異型酵素 A219V の構造解析を行った。野生型に対して A219V の主鎖の root mean square deviations (RMSD)は 0.135 Å であり、構造にほとんど差がなかった。また、酵素学的パラメータも差が見られなかった。従って、このアミノ酸置換によりセフォタキシムに対する活性が大きく低下する現象は、CTX-M-1 に特異的であることが示唆された。
Keywords: β-ラクタマーゼ、基質特異性、結晶構造解析
背景と研究目的:
基質特異性拡張型β-ラクタマーゼ(Extended spectrum β-lactamase, ESBL)は、活性中心にセリン残基を持つβ-ラクタマーゼで、Ambler のアミノ酸配列を基とした分類では、クラスAまたはDに分類される[1]。ESBLはペニシリン系の抗菌薬に加え、セフォタキシムに代表されるオキシイミノセファロスポリン系抗菌薬(第3世代セファロスポリン)やモノバクタム系抗菌薬にわたる広範囲の加水分解活性を有する酵素である。 ESBLには大きく分けて2つのタイプがあり、1つは TEM-1 型(患者名に由来)[2]や SHV(sulphydryl variable)-1 型[3]などのペニシリナーゼの親酵素に、1~数か所のアミノ酸置換が生じたことで基質特異性が拡張した酵素群である。もう1つは TEM-1 型等とはアミノ酸配列の相同性が低い酵素群で、その代表として CTX-M 型が挙げられる。CTX-M 型ESBLは、1990年代から亜型が次々と分離され、それらを産生する腸内細菌科細菌の蔓延が世界的に問題となってきた[4-6]。本邦においては、TEM-1 型や SHV-1 型より CTX-M 型ESBLの分離例が多く報告されており、特に CTX-M-2、CTX-M-3 やそれらにアミノ酸配列の相同性が高い亜型の分離例が多い[7, 8]。
CTX-M 型ESBLは、これまでに数種の亜型について立体構造が解明されている[9, 10]。これらによると、CTX-M 型ESBLの全体構造は非常によく似ている。また、基質特異性に関与するアミノ酸残基についても、よく調べられている[11, 12]。これらのアミノ酸残基のすべては基質結合に関与する部位に位置していることが分かっている。ところが、José らは CTX-M-1 にランダムなアミノ酸置換を導入した実験を行い、A219V のアミノ酸置換によって、セフォタキシムに対する活性(kcat/Km)が 1/10000 以下になることを示した[13]。Ala219 は基質結合部位とは異なる位置にあり、このアミノ酸置換により何らかの全体の構造変化が生じることが示唆された。また、A219 は現在までにDNA配列が報告されている224種の CTX-M のほぼすべてで保存されているため、何らかの重要な役割を果たしているものと考えられた。申請者は、2015A6554 において、IMP型β-ラクタマーゼにおいて、アミノ酸置換の位置とは異なる、離れた位置における構造変化が生じる可能性を示した[14]。そこで、IMP型β-ラクタマーゼと比較する目的で、同様の構造変化の可能性が示唆される、CTX-M 型β-ラクタマーゼのA219V変異型酵素の構造解析を行った。CTX-M-1 の立体構造は解明されていないため、本邦での分離例の多い CTX-M-2 にアミノ酸置換(A219V)を導入し、酵素活性と立体構造がどのように変化するかについて調べた。
実験:
発現ベクター pET28a に CTX-M-2 遺伝子をクローニングした。また、Site-Directed Mutagenesis kit (Stratagene, La Jolla, CA)を用いて部位特異的変異を導入し、A219V を得た。これらで大腸菌 BL21(DE3)を形質転換した株を用いて、野生型、変異型 CTX-M-2 を高発現させた。培養した形質転換体を超音波破砕機を用いて破砕、遠心分離した上清を陽イオン交換カラムクロマトグラフィー(CM-Toyopearl 650S (Tosoh))に添加し、電気泳動的に単一にまで精製した。精製タンパク質を用いて市販の結晶化溶液キットを用いたスクリーニングを行った。最終的にハンギングドロップ法を用い、5 mM HEPES pH 7.3 中でタンパク質濃度 20 mg/mL の溶液と、結晶化母液として 1.9 M ammonium sulfate, 0.1 M sodium citrate pH 5.6, 0.2 M potassium sodium tartrate を等量混合して 15℃ で静置することにより、0.1×0.1×0.1 mm の6面体状結晶を得た。
得られた結晶を 20 %PEG200を 含む結晶化母液に浸漬し、BL44XUにおいて、100 K の窒素気流下、露光時間 0.2 秒、振動角 1º 、波長 0.9000 Å、カメラ長 175 mm で回折データを収集した。検出器は MAR300HE(Rayonix)を使用した。その結果、いずれも分解能 1.30 Å までの回折データを収集した。得られた回折データは、プログラム HKL2000 で処理した。
CTX-M-2 の構造は、CTX-M-44(Toho-1)(PDB code: 1IYS)の構造を用い、CCP4 suite のプログラム Molrep を用いて分子置換法により求めた。得られた初期解を CCP4 suite のプログラム REFMAC 5.5 により、分解能 50.00 – 1.30 Å の範囲で精密化した。
セフォタキシム(Δε260 = –7,500 M-1cm-1)の酵素学的パラメータ(Km, kcat)の測定は、30℃ で 3.0 mL の 50 mM sodium phosphate buffer (pH 7.0)中、Jasco V-730 BIO 分光光度計を用いて行った。酵素は失活を避けるために、20 µg mL-1 bovine serum albumin を加えた測定用緩衝液で希釈して使用した。酵素学的パラメータは、基質濃度を段階的に変えて初速度を測定し、Lineweaver-Burk plot を作成することにより求めた。
結果および考察:
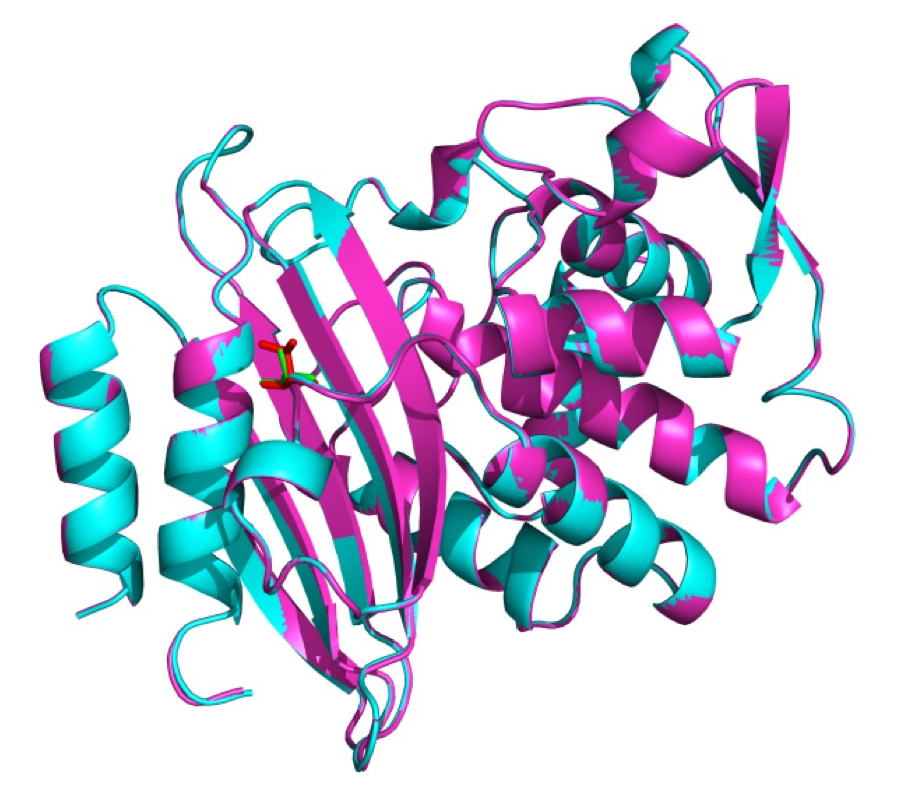
野生型 CTX-M-2、A219V ともに良好な回折像を示し、50 – 1.30 Å 分解能のデータを収集した。また、分解能 50.00 – 1.30 Å の範囲で構造を精密化した。結晶学的統計値と精密化の統計値を表1に示した。分子置換法により求めた電子密度図は、いずれも29アミノ酸からなるリーダーペプチドとN末端の2アミノ酸を除く Ser28—Phe290 で主鎖、側鎖の帰属が可能であった。アミノ酸のナンバリングは Ambler らによって提唱されたものを使用した[15]。野生型 CTX-M-2 と、CTX-M-1 と1アミノ酸のみが異なり、その構造が解明されている CTX-M-15 との主鎖の root mean square deviations (RMSD)は 0.425 Å であることから、CTX-M-2 と、CTX-M-1 の構造は非常に似通っていることが推察された。従って、Ala219 の機能は CTX-M-2 でも保存されているものと考えられた。ところが、野生型 CTX-M-2 と A219V の主鎖のRMSDは僅かに 0.135 Å であり、構造に差は見られなかった(図1)。最も異なる部分でもC末端のα炭素で 0.69 Å であった。さらに、セフォタキシムに対する kcat/Km は野生型 CTX-M-2 で 2.28 µM-1 s-1、A219V で 1.82 µM-1 s-1 と差が見られなかった。
| 野生型 CTX-M-2 | A219V | |
| Data collection | ||
| Space group | P3221 | P3221 |
| a (Å) | 72.50 | 72.78 |
| b (Å) | 72.50 | 72.78 |
| c (Å) | 97.49 | 97.95 |
| Resolution range a (Å) | 50-1.30 (1.32-1.30) | 50-1.30 (1.32-1.30) |
| Observed reflections a | 767055 (36916) | 771818 (35532) |
| Unique reflections a | 73312 (3615) | 74238 (3662) |
| Completeness a (%) | 99.1 (99.3) | 99.6 (99.8) |
| Redundancy a | 10.6 (10.3) | 10.4 (10.2) |
| Average I/σ a | 13.8 (10.2) | 18.7 (5.7) |
| CC1/2 | 0.980 (0.973) | 0.981 (0.949) |
| Rmerge a, b (%) | 0.169 (0.237) | 0.102 (0.385) |
| Refinement | ||
| Resolution limit (Å) | 50.0-1.30 | 50.0-1.30 |
| Rwork c / Rfree d | 0.186 / 0.212 | 0.200 / 0.219 |
| Number of protein atoms | 1966 | 1968 |
| Number of water molecules | 292 | 230 |
| RMSD | ||
| Bond length (Å) | 0.029 | 0.034 |
| Bond angle (°) | 2.674 | 2.78 |
| Average B-factor (Å2) | ||
| Main-chain | 10.3 | 14.7 |
| Side-chain | 13.9 | 18.5 |
| Water molecules | 21.5 | 23.8 |
a Values for the highest resolution shells are given in parentheses.
bRmerge = ΣhklΣi|I(hkl)i-‹I(hkl)›|/ΣhklI(hkl).
cRwork = Σ(Fobs-Fcalc)/Σ(Fobs).
dRfree: crystallographic R-factor based on 5 % of the data withheld from the refinement for cross-validation.
図1. 野生型 CTX-M-2 (cyan)と A219V (magenta)の重ね合わせ。Ala219 は green、Val219 は red で示した。
Ala219 は CTX-M-208 を除くすべての CTX-M 型ESBLで保存されていることから、酵素の機能に何らかの重要な役割を果たしていると考えられた。また、我々は以前にIMP型β-ラクタマーゼにおいて、基質結合にかかわらない部位のアミノ酸置換が、別のドメインに存在する基質結合にかかわるループの構造に影響を与えることを示した[14]。アミノ酸置換部位とは異なる遠距離の相互作用が生じる現象は、β-ラクタマーゼに広くみられることを期待したが、CTX-M-2 ではそれが見られなかった。従って、この現象は CTX-M 型ESBLの中でも CTX-M-1 に特異的な現象であると考えられた。
今後の課題:
A219V のアミノ酸置換によりセフォタキシムに対する活性が 1/10000 以下になることは、CTX-M 型ESBLの中でも CTX-M-1 に特異的な現象であると考えられた。そこで CTX-M-1 遺伝子をクローニングし、アミノ酸置換を導入した。野生型、及び変異型酵素を大腸菌で発現させ、結晶化することを試みている。しかしながら、現時点までに発現、結晶化とも成功していないため、一旦構造解析を中断することとした。
参考文献:
[1] Ambler R. P., Philos. Trans. R. Soc. Lond. B. Biol. Sci. 289, 321-331 (1980).
[2] Medeiros A. A., BR. Med. Bull. 40, 18-27 (1984).
[3] Chaves J. et al., Antimicrob. Agents Chemother. 45, 2856-2861 (2001).
[4] Bradford P. A., Clin. Microbiol. Rev. 14, 933-951 (2001).
[5] Rossolini G. M., D’Andrea M. M., Mugnaioli C., Clin. Microbiol. Infect. 14, 33-41 (2008).
[6] Cantón R. et al., Clin. Microbiol. Infect. 14, 144-153 (2008).
[7] 金森政人、遠藤英子 杏林医会誌、35, 205-214 (2004).
[8] 若松篤 ら 日本化学療法学会雑誌、54, 447-452 (2006).
[9] Ibuka A. S. et al., Biochemistry 42, 10634-10643 (2003).
[10] Chen Y. et al., J. Mol. Biol. 348, 349-362 (2005).
[11] Delmas J. et al., J. Mol. Biol. 400, 108-120 (2010).
[12] Ibuka A. S. et al., Antimicrob. Agents Chemother. 55, 284-290 (2011).
[13] José F. et al., Antimicrob. Agents Chemother. 55, 4361-4368 (2011).
[14] 山本惠三 SPring-8利用成果集、Section A 6, 226-229 (2018).
[15] Ambler R. P. et al., Biochem. J., 276, 269-270 (1991).
(Received: December 3, 2018; Early edition: February 26, 2019; Accepted: July 16, 2019; Published: August 29, 2019)