Volume7 No.1
SPring-8 Section A: Scientific Research Report
CD200-CD200R複合体の結晶構造解析
Crystal Structure of the CD200/CD200R Complex
a熊本大学、b東京医科歯科大学
a Kumamoto University, b Tokyo Medical and Dental University
- Abstract
-
CD200は幅広い種類の細胞上で発現しており、骨髄細胞上の阻害型受容体CD200R(マウスではCD200R1)との結合を介して骨髄細胞を抑制的に制御する。この抑制機構の構造生物学的解明を目的として、マウスのCD200-CD200R1複合体の結晶構造解析を 2.7 Å 分解能で行った。解析の結果、正に荷電したCD200のドメイン1と負に荷電したCD200R1のドメイン1が静電相互作用により複合体を形成することが明らかになった。
Keywords: CD200-CD200R複合体、分子認識、結晶構造
背景と研究目的
リガンドであるCD200は様々な細胞上に発現し、骨髄細胞上のCD200受容体(R)と結合することにより、免疫応答を制御する。マウスにおけるCD200Rは、阻害型がCD200R1の1種類、活性化型がCD200R2-5の4種類存在する。CD200およびCD200Rは細胞表面受容体に分類され、アミノ酸配列から Immunoglobulin (Ig) V-set からなるドメイン(D)1 と Ig C2-set からなるD2からなる細胞外領域を持つことが予想されている。CD200R1はCD200と結合するが、CD200R2-4はいずれもCD200とは結合せず、これらのリガンドは明らかにされていない[1]。ヘルペスウイルスやポックスウイルスの中には宿主のCD200とアミノ酸配列の相同性の高いCD200様分子を持ち、これらのウイルスCD200が宿主の骨髄細胞上の阻害型受容体CD200R1と結合し、免疫を抑制的に制御する[2,3]。この免疫抑制機構を構造生物学的に解明することを目的として、CD200-CD200R1複合体の構造解析を行った。
実験:
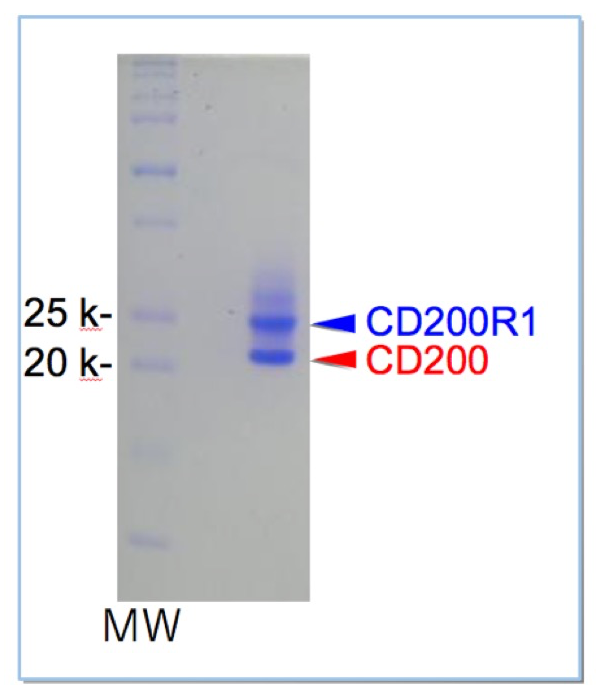
α annosidase 阻害薬 kifunesine の存在下で、CD200とCD200R1の両分子のC末端側にHisタグを付加して、HEK293T細胞を用いた共発現により複合体として発現させた。Niカラムを用いて精製を行なった後、Endo F1を用いてAsnに結合したN-アセチルグルコサミン1残基を残して糖鎖を切除した。その後、イオン交換カラムやゲルろ過クロマトグラフィーを用いて精製を行なった。精製した試料がCD200-CD200R1複合体であることをSDS-PAGEにより確認し(図1)、結晶化を行なった(図2)。SPring-8 BL44XUにおいて得られた結晶の回折実験を行い、結晶がタンパク質の結晶であることを確認したが、分解能が 6-8 Å と低く、解析に至らなかった。結晶化条件の精密化して、0.1 M Imidazole pH 7.5、0.2 M Lithium sulfate、5 % (w/v) PEG3350 の条件下で成長させた結晶を用いて、PF BL-1Aにおいて 2.7 Å 分解能までのデータを収集した。HKL2000を用いてデータ処理を行なった結果、CD200-CD200R1複合体結晶の空間群はP4122であり、解説強度データの Rmerge は 13.0% であった(表1)。さらに、20 mM K2PtCl4 の存在下で誘導体結晶を調製し、3.5 Å 分解能までの回折強度データをPF BL-1Aにおいて収集した。SAD法により 3.5 Å 分解能で位相を付けて、2.7 Å 分解能まで位相拡張を行った後、モデルの構築を行なった。Phenixを用いて Rcryst が 0.217、Rfree が 0.275 まで精密化を行った。
図1. CD200-CD200R複合体の結晶化試料。
図2. CD200-CD200R複合体結晶。
| CD200 - CD200R1 | CD200 - CD200R1 Pt | |
| Data Collection | ||
| Space group | P43212 | P43212 |
| Unit cell parameters | ||
| a=b (Å) / c (Å) | 126.97 / 114.98 | 125.6 / 114.58 |
| Resolution range (Å) | 30.0 - 2.70 | 30.0 - 3.5 |
| Wavelength (Å) | 1.100 | 1.0712 |
| Completeness (%) | 98.3 (88.4) | 97.4 (92.3) |
| Rmerge (%) | 13.0 (49.0) | 13.3 (45.9) |
| < I / sigmaI > (%) | 18.2 (2.0) | 12.1 (1.8) |
| Refinement | ||
| Rcryst /Rfree (%) | 21.7 / 27.5 | |
| R.m.s.d in bonds (Å) | 0.009 | |
| R.m.s.d in angles (°) | 1.501 | |
| Ramachandran plot | ||
| Most favored (%) | 86.0 | |
| Additional allowed (%) | 14.0 |
結果と考察:
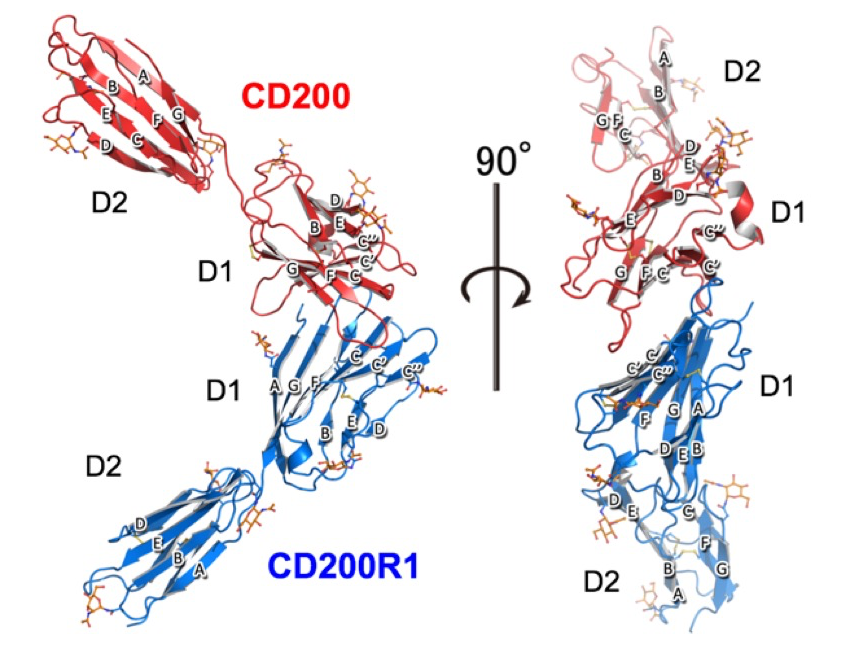
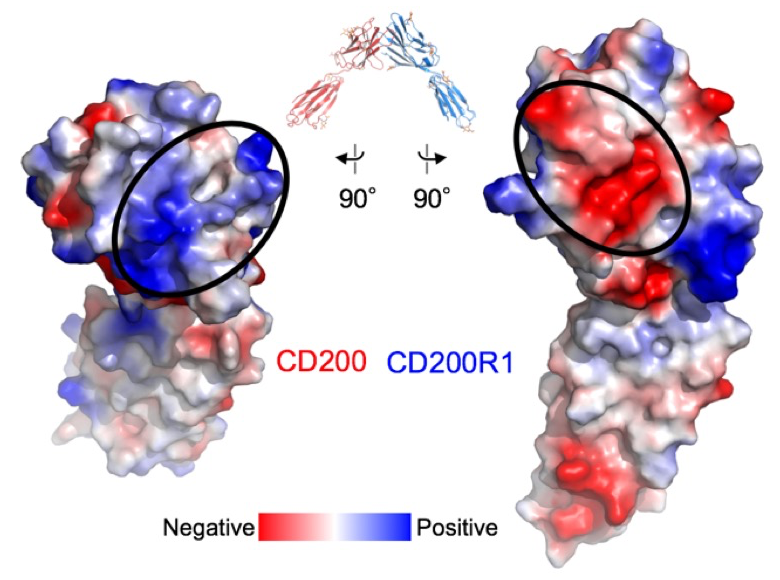
構造解析の結果、CD200及びCD200R1は、Ig V-set からなる D1 と Ig C1-set から D2 からなることが明らかになった。一般的に Ig C1-set を持つタンパク質は抗原認識に直接関わる抗体、MHC、T細胞受容体などであり、細胞表面受容体は Ig C1-set を持たない。細胞表面受容体で Ig C1-set を持つことが初めて明らかにされた分子は補助刺激分子 B7-1 であり[4]、CD200は限定的ではあるがアミノ酸レベルで B7-1と 相同性がある分子として同定された[5]。CD200とCD200R1は、N末端の Ig V ドメインを介して複合体を形成することが明らかになった(図3)。これらの分子の結合親和力は Kd = 0.6 µM と非常に強く[6]、正に荷電したCD200と負に荷電したCD200R1が静電相互作用により複合体を形成するためであることが分かった(図4)。我々が構造解析を終えるとほぼ同時にOxfordのグループからこの複合体の構造が報告され、彼らの結晶は我々の結晶と同型であり、その構造は我々のものとほぼ同一であった[6]。
図3. CD200-CD200R複合体の構造。
図4. 静電ポテンシャル
今後の課題:
CD200-CD200R1複合体の構造解析に成功し、CD200とCD200R1の分子認識機構を構造生物学的に解明した。今後、近年CD200R1のリガンドとして同定されたiSEC1 [7]とCD200R1の認識機構の解明を構造生物学的に行いたい。
謝辞:
本研究におけるデータ測定は PF BL-1A 及び PFAR-NW12A(課題番号:12G721)、SPring-8 BL44XU (課題番号:2012A6703)で行った。これらのビームラインスタッフに感謝致します。
参考文献:
[1] Wright, G. J. et al. J. Immunol. 171, 3034-3046 (2003).
[2] Shiratori, I. et al. J. Immunol. 175, 4441-4449 (2005).
[3] Cameron, C. M., et al. J. Virol. 79, 6052-6067 (2005).
[4] Ikemizu, S., et al. Immunity 12, 51-60 (2000).
[5] Borriello, F., et al. J. Immunol. 158, 4548-4554 (1997).
[6] Hatherley, D., et al. Structure 21, 820-832 (2013).
[7] Kojima, T., et al. Sci. Rep. 6, 36457 (2016).
ⒸJASRI
(Received: September 28, 2018; Early edition: December 26, 2018; Accepted: December 17, 2018; Published: January 25, 2019)